群落多樣性組成譜測(cè)序
群落多樣性組成譜測(cè)序
群落多樣性組成譜測(cè)序以細(xì)菌 / 古菌 16S rRNA 基因可變區(qū)、真菌 18S r RNA 基因可變區(qū)或真菌 ITS(Internal transcribed spacer)內(nèi)轉(zhuǎn)錄間隔區(qū)等微生物特征序列(Signature sequence)為靶點(diǎn),通過(guò)檢測(cè)序列的變異和數(shù)量,反映群落 中各類(lèi)微生物物種的身份和豐度,從而快速解析群落物種分布特征,闡明樣本間多樣性和組成差異,進(jìn)而發(fā)現(xiàn)差異相 關(guān)物種。
產(chǎn)品特點(diǎn)
1. 直接對(duì)群落樣本中的微生物特征序列進(jìn)行擴(kuò)增檢測(cè),簡(jiǎn)單快速,并克服了大部分微生物無(wú)法
純培養(yǎng)的難題;
2. 自動(dòng)化、專(zhuān)業(yè)的 DNA 提取流程,配合高保真 DNA 聚合酶進(jìn)行 PCR 擴(kuò)增,并嚴(yán)格把控?cái)U(kuò)增循環(huán)數(shù),確保
群落組成客觀真實(shí);
3. 使用 Illumina NovaSeq/MiSeq 和 PacBio Sequel II 進(jìn)行測(cè)序, 2×250 bp、2×300 bp、>1 kb 的多種長(zhǎng)
度均可適配,每個(gè)樣本都能一次性獲得數(shù)萬(wàn)條序列,即使低豐度物種也能定量;
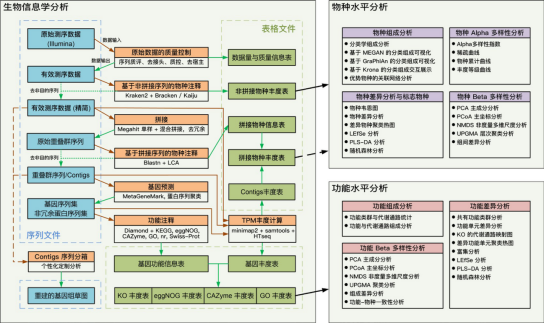
4. 采用 QIIME 2 分析流程, DADA2、物種注釋、PICRUSt2 功能潛能預(yù)測(cè)等方法覆蓋;
5. 提供分析結(jié)果,和發(fā)表等級(jí)的動(dòng)態(tài)交互可視化效果。
項(xiàng)目流程
微生物組總DNA提取→目標(biāo)片段PCR擴(kuò)增→擴(kuò)增產(chǎn)物檢測(cè)定量→測(cè)序文庫(kù)制備→上機(jī)進(jìn)行高通量測(cè)序→生信分析
分析內(nèi)容