
2023-07-17

《Environmental Microbiome》 影響因子 7.9
近日,中國科學技術大學在環境科學與生態學領域的《Environmental Microbiome》發表新研究成果!本研究揭示了偽魚腥藻Chao1811的噬藻體Pan1~Pan5的基因組特征以及進化多樣性;系統發育分析為藍細菌和α-變形菌建存在的由噬菌體噬菌體介導的門間基因水平轉移提供了直接證據。此外,首次在噬藻體中發現了水平基因轉移不僅可能發生在種間,甚至可能發生在門間的進化痕跡,這表明噬菌體或噬藻體可以作為一種強大的工具在不同物種或門之間進行基因操作。 本研究的噬菌體基因組測序和分析由上海派森諾生物科技股份有限公司完成。
一、研究背景
隨著發展中國家的快速發展和城市化,不斷發展的城市周邊水體污染嚴重,富營養化程度高,從而導致藍藻的季節性爆發。系統地分離和鑒定淡水噬藻體可能為控制可怕的水華提供一種生物解決方案。然而,迄今為止,對淡水噬藻體的基因組序列和相關研究仍然非常有限。本研究以巢湖產的一株偽魚腥藻Chao 1811為寄主,進一步分離純化了4個長尾和1個短尾淡水噬藻體。比較基因組學和系統發育分析表明,這五種噬藻體在進化上彼此不同。
二、研究材料與方法
1.實驗材料:偽魚腥藻Chao1811中分離得到的5個噬菌體,命名為Pan1~Pan5 2.測序平臺:Illumina 3.分析內容:噬菌體基因組測序、系統發育樹構建、共線性分析等。
三、研究結果
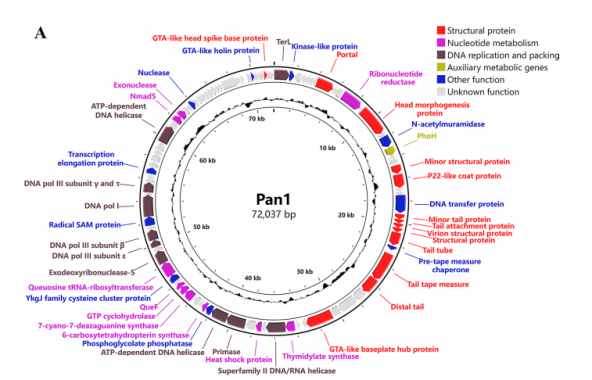
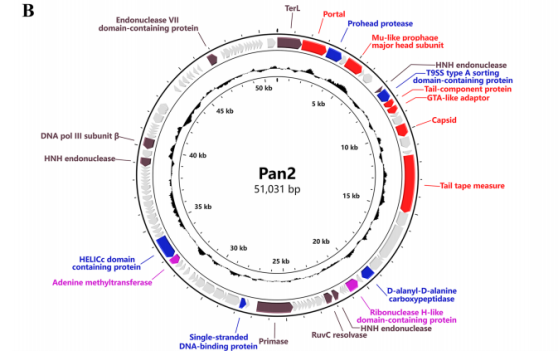
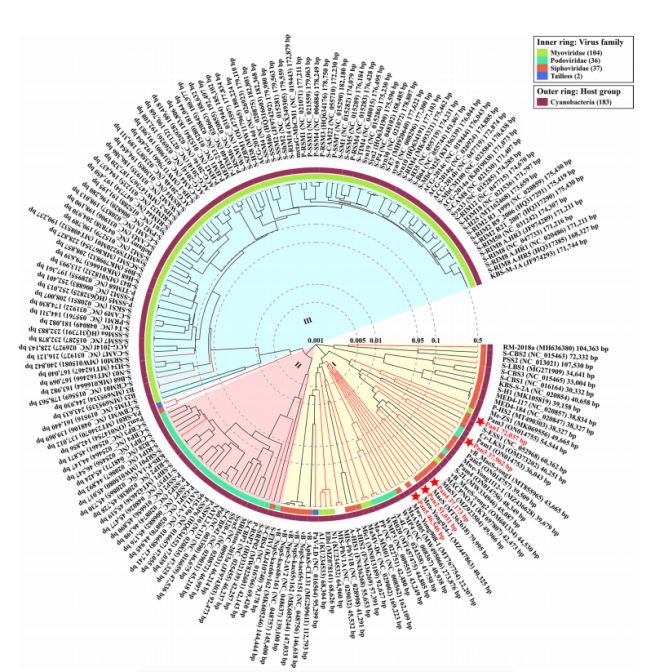
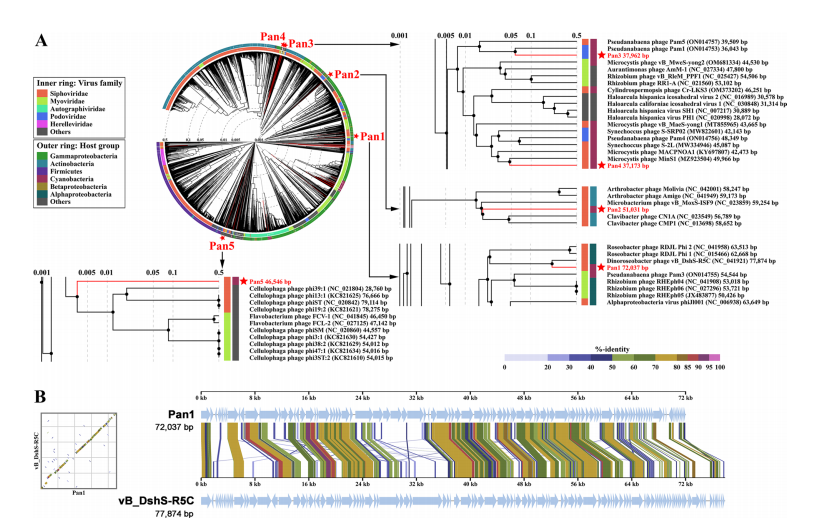
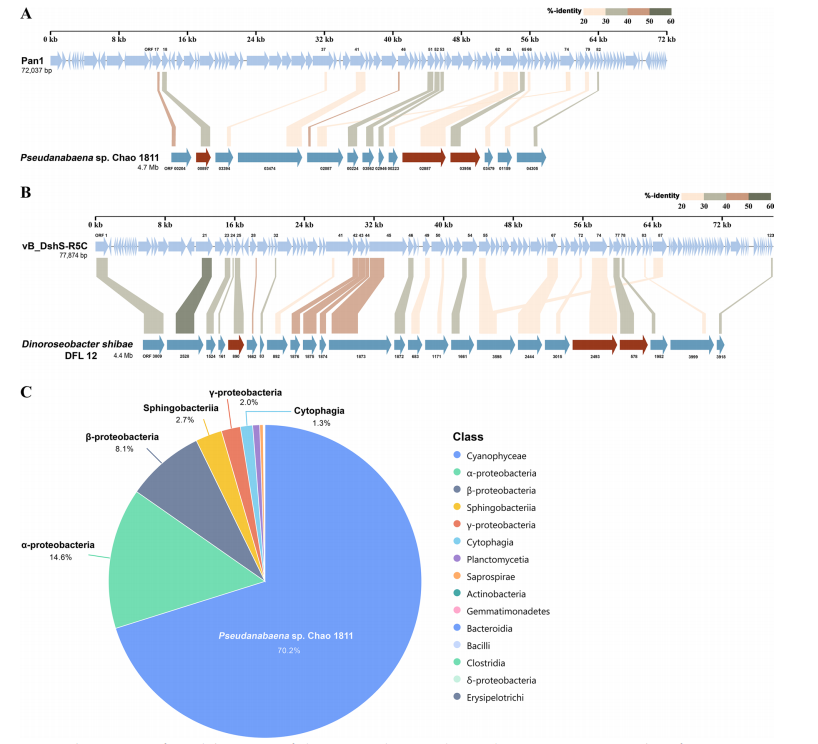
偽魚腥藻 Chao 1811及5種噬藻體的鑒定 經過三輪篩選分離得到一株藍藻,16S分析將該菌株歸屬于Pseudanabaena屬,命名為偽魚腥藻Chao 1811。全基因組測序結果表明,偽魚腥藻Chao 1811擁有4,729,581 bp的基因組,GC含量為43%,其中包含4,329個假定的開放閱讀框。分離純化了5株噬藻體,這些噬菌體以Chao 1811為寄主。電鏡顯示,Pan1具有長94 nm、寬58 nm的長型二十面體頭部,與T4噬菌體相似,Pan2~Pan5均具有直徑約60~70 nm的經典二十面體頭部。從尾巴形態上看,它們分屬兩科:Pan1、2、4、5,尾巴長而靈活;Pan3,尾巴短而不收縮。 圖1 偽魚腥藻Chao 1811與5種噬藻體的鑒定 噬藻體Pan1~Pan5的基因組序列 對5個噬藻體進行全基因組測序,結果顯示其均含有一個雙鏈DNA基因組,大小為72,037、51,031、37,962、37,173和46,546 bp。除Pan5外,Pan1~Pan4的GC含量均顯著高于宿主Chao 1811。值得注意的是,具有長尾頭部的Pan1具有高度緊湊的基因組,表明其具有獨特的基因組包裝模式。噬藻體Pan1~Pan5分別擁有104個、103個、65個、55個和71個ORF,這些注釋蛋白可分為結構蛋白、核苷酸代謝蛋白、DNA復制與包裝蛋白和其他功能4個功能基團;在Pan1中發現了一個典型的AMG phoH (gp18)。此外,首次在噬藻體中發現Pan1編碼一個DNA轉移蛋白gp25,該蛋白有助于將噬菌體DNA注射到宿主中。 圖2 噬菌體基因組圈圖(以pan1和pan2為例) Pan1~Pan5的比較基因組學分析 比較基因組分析發現Pan1~Pan5在基因組水平上存在差異,為了進一步研究這五種噬藻體的進化關系,對183個已測序的噬藻體基因組進行了蛋白質樹構建,將噬藻體分為三個群體:群體I包含海洋長尾噬藻體和大多數淡水噬藻體,群體II和III分別包含海洋短尾和收縮尾的噬藻體。進化樹結果顯示Pan1~Pan5位于兩個分支上:一個與Pan1、Pan3和Pan4,另一個與Pan2和Pan5。在蛋白質組學樹中,Siphoviridae Pan1和Podoviridae Pan3與先前分離的Myoviridae Pam3和Podoviridae Pam1的進化距離比較近。綜上所述,盡管感染同一個寄主Chao1811,但噬藻體Pan1~Pan5在進化上存在多樣性。 圖3 蛋白質進化樹 Pan1類似于感染α-變形菌的噬菌體 噬菌體全基因組進化樹分析顯示:Pan1~Pan5不屬于同一分支或相鄰分支,而是離散分布的,表明它們具有不同的進化起源。根據SG值,Pan1、Pan2和Pan5分別在一定程度上與噬菌體聚集在一個分支中。但Pan3和Pan4分別與具有相同尾部形態的噬藻體Pam1和MinS1聚集在一起。且進行基因組比對發現發現Pan1在基因組水平上與感染α-變形菌的噬菌體vB_DshS-R5C相似。 圖4 Pan1~Pan5的系統發育分析 噬菌體介導基因轉移的進化痕跡 為了篩選噬菌體與宿主之間潛在的重組,blast比對發現Pan1與Chao 1811共有14個同源蛋白,而vB_DshS-R5C與D. shibae DFL 12共有24個同源蛋白。值得注意的是,這兩種噬菌體及其宿主共有3個蛋白質PhoH、DNA聚合酶I和DNA聚合酶III亞基γ/τ,它們在Chao 1811和D.shibae DFL12之間的序列同源性分別為43%、36%和21%。系統發育分析表明,藍藻菌和α-變形菌的PhoH蛋白大部分是分開類群的;然而,來自α-變形菌門的一對PhoH蛋白被分組在藍藻門的分支中,表明兩個門之間的PhoH基因交換。 圖5藍藻與α-變形菌門間基因轉移的進化痕跡 16S測序發現除70.1%的偽魚腥藻Chao 1811外,α-變形菌屬為主要污染菌種。這表明Chao 1811確實傾向于與α-變形菌在同一群落中共存,這為門間基因轉移提供了可能。此外,Chao 1811的全基因組測序數據中還可以組裝出長度為3,135,941 bp的α-變形菌。序列分析表明,它還編碼一個輔助代謝蛋白PhoH,與Pan1和Chao 1811的序列同源性分別為26.87%和40.60%。值得注意的是,Chao 1811編碼多個重組酶、整合酶以及超過160個轉座酶,這可能使基因重組的頻率很高。噬藻體Pan1也編碼三種RcGTA-like蛋白gp37、gp96和gp101,進一步表明基因從α-變形菌轉移的痕跡。此外,上述phoH基因存在于兩種噬菌體(Pan1和vB_DshS-R5C)及其宿主(藍藻菌和α-變形菌)中,為噬菌體介導的門間基因轉移提供了直接證據。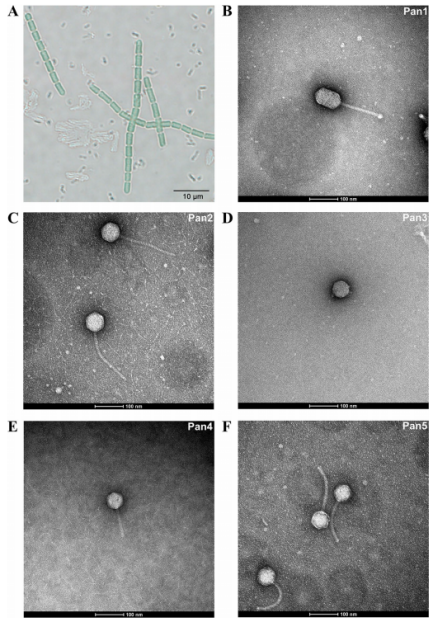
四、結 論
本研究分離并鑒定了5種感染Pseudanabaena sp. Chao1811的淡水噬藻菌Pan1~Pan5,對其進行比較基因組分析,使人們能夠更好地了解淡水噬藻的進化多樣性。此外,通過系統發育分析,在不同的噬菌體之間,或宿主藍藻與附著的α-變形菌之間,表征了一系列基因轉移的進化痕跡。這表明噬菌體介導的門間水平基因轉移將賦予細菌在進化過程中適應波動環境的新特征。
如需進一步討論,歡迎發郵件或者致電我們喲(郵箱地址:[email protected],聯系電話:021-80118168-8617)!
文章索引:Zhu J, Yang F,Du K, et al.Phylogenomics of five Pseudanabaena cyanophages and evolutionary traces of horizontal gene transfer[J]. Environmental Microbiome, 2023, 18:3