《Nature Communications》
影響因子:17.694
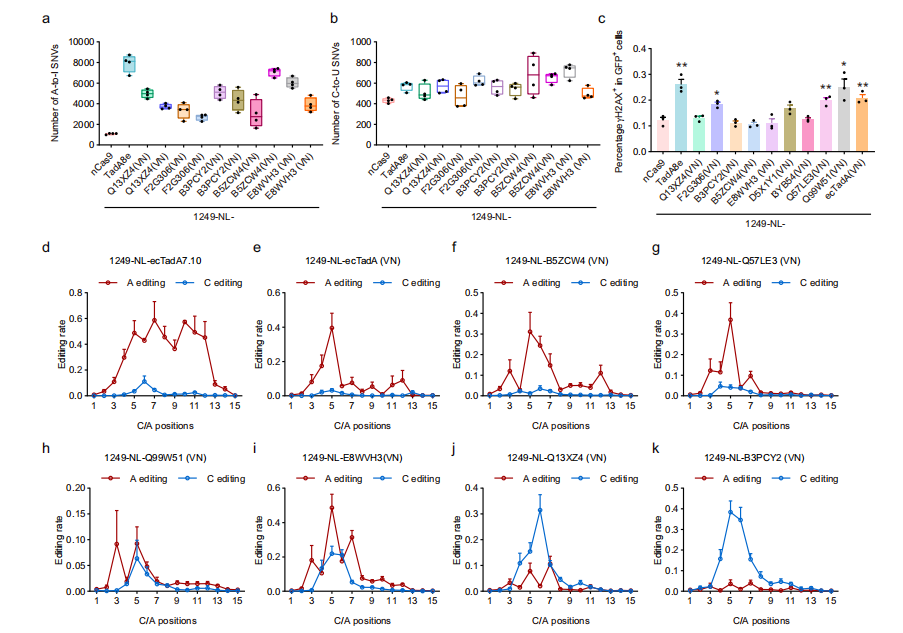
堿基編輯器的胞嘧啶和腺嘌呤編輯分別需要胞嘧啶和腺苷脫氨酶,沒有一種脫氨酶能夠同時(shí)進(jìn)行胞嘧啶和腺嘌呤編輯。此外,胞苷和腺苷脫氨酶的不同屬性易導(dǎo)致各種類型的脫靶效應(yīng),包括對(duì)胞嘧啶堿基編輯器(CBE)的cas9獨(dú)立DNA脫靶效應(yīng)和對(duì)腺嘌呤堿基編輯器(ABE)特別嚴(yán)重的RNA脫靶效應(yīng)。本研究表明,25個(gè)TadA同源基因可以通過單或雙突變產(chǎn)生功能性ABEs、CBEs或ACBEs,這些同源基因顯示出最小的cas9獨(dú)立DNA脫靶效應(yīng)和遺傳毒性,其中B5ZCW4、Q57LE3、E8WVH3、Q13XZ4和B3PCY2是有希望進(jìn)一步改造的候選同源基因。此外,通過額外的單堿基突變,可以進(jìn)一步降低甚至消除TadA直系同源堿基編輯器的RNA脫靶效應(yīng)。
1.項(xiàng)目類型:擴(kuò)增子測(cè)序
2.測(cè)序平臺(tái):Sanger、Illumina NovaSeq
3.分析內(nèi)容:TadA同源基因的系統(tǒng)發(fā)育比較、TadA同源物的篩選與工程研究、R-loop檢測(cè)cas9獨(dú)立的DNA脫靶分析等。
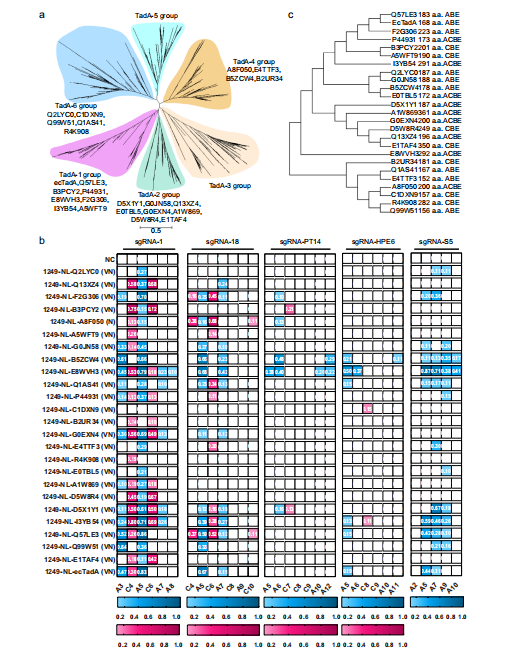
圖1 功能ABEs、CBE和ACBE的TadA同源性鑒定
圖2 脫靶效應(yīng)、具有代表性的TadA同源物衍生的堿基編輯器的遺傳毒性和編輯范圍
本研究對(duì)近千種TadA同源蛋白構(gòu)建了系統(tǒng)進(jìn)化樹,并挑選>50種其它物種來源的TadA同源蛋白進(jìn)行類似的改造和功能篩選。研究人員發(fā)現(xiàn)大腸桿菌來源的野生型ecTadA添加5’-NLS和3’-柔性接頭序列并融合于nCas9內(nèi)部的DS1249位點(diǎn)后,僅通過A106V和D108N雙點(diǎn)突變便可獲得具有明顯腺嘌呤和胞嘧啶編輯活性的功能性堿基編輯器1249-NL-ecTadA(VN),其A5的A-G突變率為83%,C4的C-T突變率為27%。
總而言之,本研究通過篩選和設(shè)計(jì)TadA同源蛋白質(zhì)工程化改造,證實(shí)了ABE、CBE以及ACBE在內(nèi)的各類堿基編輯器開發(fā)的可行性,并減少或最小化了脫靶效應(yīng)。本研究結(jié)果不僅擴(kuò)展了堿基編輯的工具包,而且為編輯脫氨酶的潛在演化過程提供了重要的證據(jù),同時(shí)也為堿基編輯器的開發(fā)拓展了新思路。
《Nature Communications》
影響因子:17.694
雖然微型CRISPR-Cas12f系統(tǒng)最近被開發(fā)出來,但衍生的微型胞嘧啶和腺嘌呤堿基編輯器(miniCBEs和miniABEs)的編輯功效和靶向范圍尚未得到全面解決。此外,功能型miniCBEs尚未建立。本文,研究人員生成了各種Cas12f衍生的miniCBEs和miniABEs堿基編輯器,開發(fā)出功能性的miniCBEs和具有全新活性窗口、多樣化的miniABEs。
1.項(xiàng)目類型:擴(kuò)增子測(cè)序
2.測(cè)序平臺(tái):Illumina NovaSeq
3.分析內(nèi)容:Cas獨(dú)立的DNA脫靶效應(yīng)分析、全基因組測(cè)序和數(shù)據(jù)分析、RNA編輯分析等。
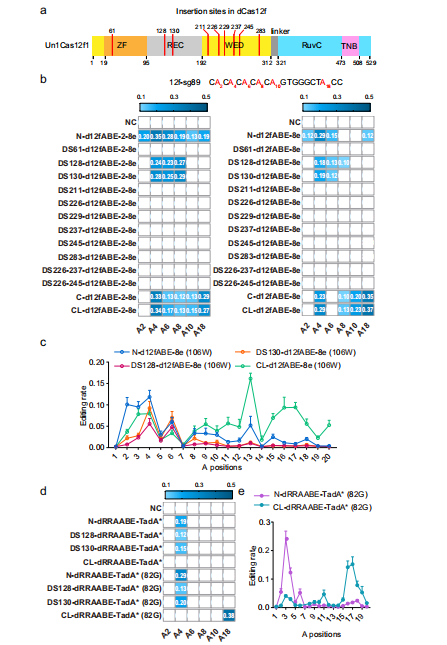
圖3活性高、范圍多樣的功能性d12抗體的開發(fā)
研究人員揭示了用傳統(tǒng)胞苷脫氨酶生成的miniCBEs具有更大編輯窗口和高脫靶效應(yīng)。為了改進(jìn)經(jīng)典miniCBEs和衍生miniABEs的編輯效率,研究人員對(duì)TadA脫氨酶進(jìn)行誘變篩選,以高精度生成有效的miniCBEs,并將off-target效應(yīng)降至最低。研究人員發(fā)現(xiàn),新設(shè)計(jì)的miniCBEs和miniABEs能夠糾正細(xì)胞系中的致病突變,并通過體內(nèi)大腦中的腺相關(guān)病毒遞送有效地引入基因突變。
總之,本研究有力地?cái)U(kuò)展了基礎(chǔ)編輯工具包,并為廣泛的遺傳疾病提供了更多的治療選擇,提高了編輯活性。本研究還證明了TadA重編程為堿基編輯優(yōu)化提供了另一種策略。本研究為CBE的發(fā)展提供了替代策略,擴(kuò)展了miniCBEs和miniABEs的工具包,并為臨床應(yīng)用提供了有前途的治療工具。
本研究的基因編輯序列測(cè)序由上海派森諾生物科技股份有限公司完成。如需進(jìn)行基因編輯序列測(cè)序分析,歡迎發(fā)郵件或者致電我們喲(郵箱地址:[email protected],聯(lián)系電話:021-80118168-6611)!
文章索引:
[1]Zhang, S., Yuan, B., Cao, J. et al. TadA orthologs enable both cytosine and adenine editing of base editors. Nat Commun 14, 414 (2023). https://doi.org/10.1038/s41467-023-36003-3.
[2]Zhang, S., Song, L., Yuan, B. et al. TadA reprogramming to generate potent miniature base editors with high precision. Nat Commun 14, 413 (2023). https://doi.org/10.1038/s41467-023-36004-2.