2022-11-18

《Nature》
影響因子:69.504
中國科學院分子植物科學卓越創新中心
恭喜中國科學院分子植物科學卓越創新中心巫永睿研究團隊在國際期刊《Nature》發表新研究成果!本研究成功從野生玉米中克隆出控制玉米高蛋白品質形成和氮素高效利用的關鍵變異基因THP9,能顯著提高雜交種籽粒蛋白含量。
一、研究背景
植物的種子含有如碳水化合物等儲存的代謝產物,這些產物對種子萌發、各種動物和人類生存發展都十分重要。因此育種家一直致力于基因改造,以創造出具有更大代謝產物比例的種子。隨著玉米大量用作牲畜飼料,淀粉含量成為主要關注點,而對蛋白質含量和風味的關注較少,導致現代玉米只含有5-10%的蛋白質;而大芻草(Teosinte),玉米的野生祖先,蛋白質含量為20-30%,其控制玉米總蛋白含量和高氮素利用的關鍵基因一直未找到,而植物蛋白質未來可能在人類飲食中將發揮更大作用。因此,未來的玉米育種必須具有更高氮利用率和高蛋白含量。
本研究為了確定導致玉米和Teosinte蛋白質含量差異的基因,分析了它們雜交后代,并對影響這一性狀的數量性狀基因座(QTL)進行了表征。對Teosinte單倍型基因組進行了測序,并定位了與種子中高蛋白含量相關的基因位點,并成功克隆Teosinte高蛋白基因座THP9,為玉米高蛋白種質培育提供了重要參考。
二、研究材料與文章思路
1、實驗材料
基于高蛋白含量的野生玉米(Teosinte)與B73自交系雜交構建高世代近等基因系群體,分別對群體BC4(n=500)、BC6(n=1314)以及BC8(n=1344)進行了3次大規模高蛋白遺傳群體的測序與定位,最后利用BC9 (n?=?2,000)進行了精細定位。
2、研究思路圖
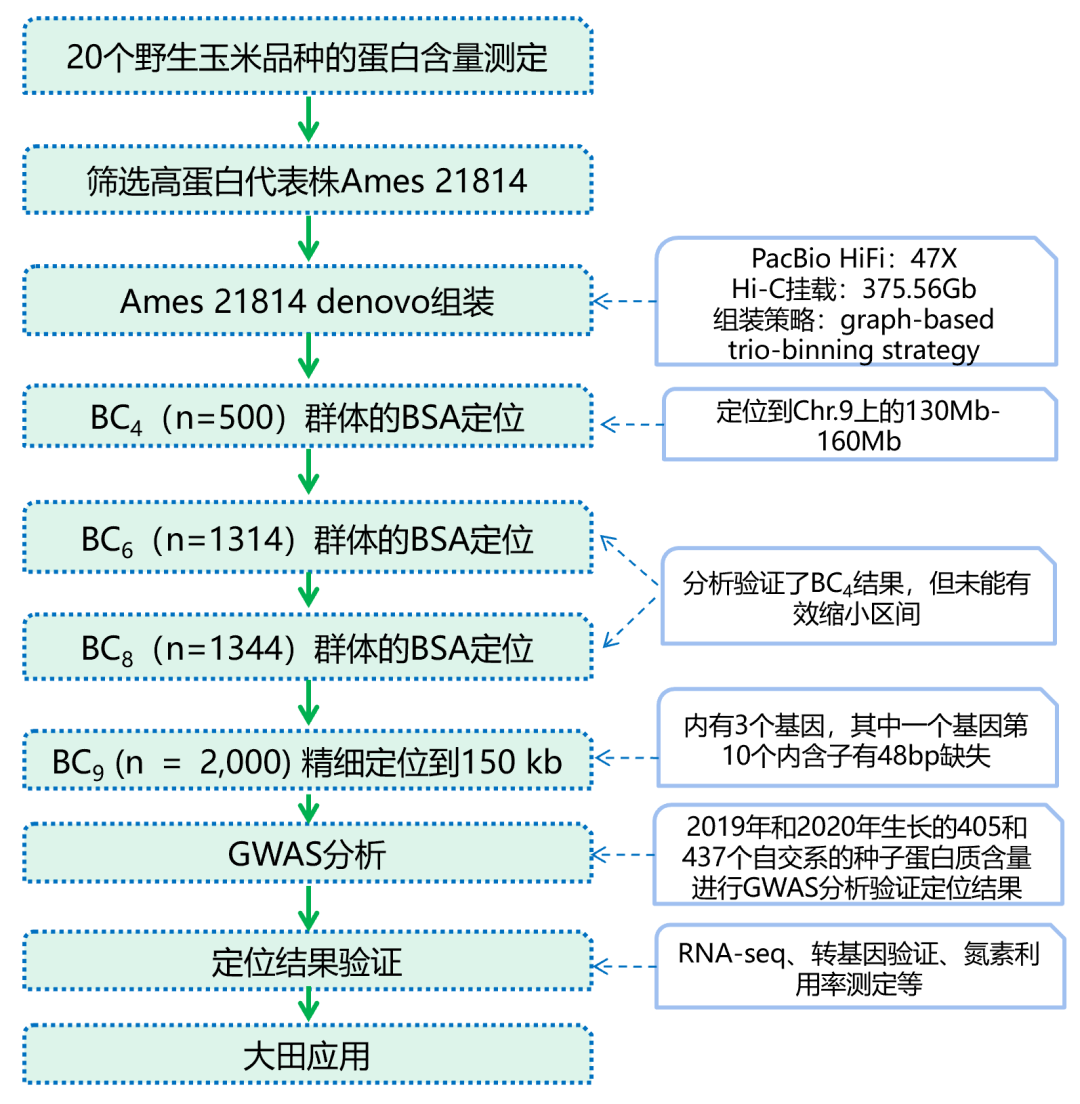
三、本文重要研究結果
本研究收集了20個野生玉米種,從中篩選出一個高蛋白含量的代表株,Ames 21814,基于HiFi數據和Hi-C數據成功組裝出該野生玉米高雜合的復雜基因組。
此外本研究成功攻克野生玉米與現代玉米B73雜交單向不親和難題,構建了超F10的高世代近等基因系家系群體,并對群體BC4(n=500)中的高低蛋白含量的子代構建了DNA混池,進行了BSA混池關聯分析,定位到Chr.9上的130Mb-160Mb的區域,該區域包含了315個Teosinte滲入基因,該位點被命名為TEOSINTE高蛋白9。此外,又對BC6(n=1314)以及BC8(n=1344)群體同樣進行了BSA混池關聯分析,驗證了BC4群體定位結果。后續又利用構建的BC9 (n?=?2,000) 群體進行了精細定位,最終將后續區域縮小到150 kb范圍內,包含有3個候選基因。
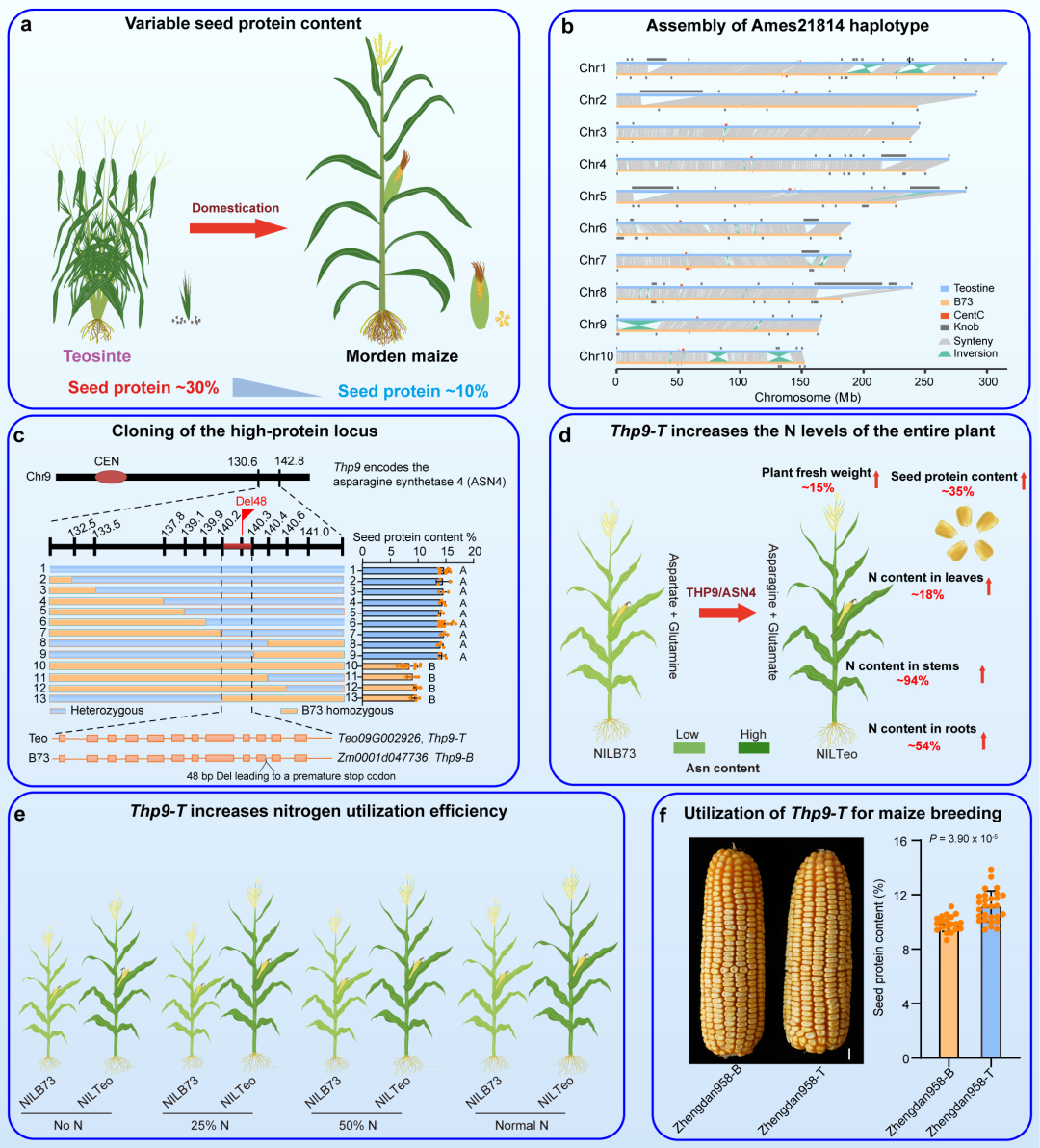
圖THP9基因的圖位克隆與蛋白含量測定
四、研究結論
該研究團隊在三亞南繁基地進行了大規模田間試驗,將野生玉米高蛋白基因Thp9-T雜交導入我國推廣面積最大的玉米生產栽培品種鄭單958中,可以顯著提高雜交種籽粒蛋白含量,表明該基因在培育高蛋白玉米中具有重要的應用潛能。
巫永睿課題組于2019年攜手派森諾,已成功在《Plant Cell》雜志發表玉米高分文章,基于BSA分析策略成功挖掘出玉米早期胚乳發育過程中的關鍵基因,揭示了早期胚乳細胞數目增殖決定最終籽粒大小的重要分子機理。
讓我們來看看小小BSA如何具有定位大功效
(一)BSA基本概念
BSA(Bulked-Segregant Analysis)即集群分離分析法,利用差異目標性狀的兩個親本構建家系,在子代分離群體中選取目標性狀個體構建DNA混合池,采用高通量測序技術對混池DNA進行建庫測序,開發全基因組范圍內的SNP、InDel分子標記,以通過計算SNP、InDel的基因型頻率,在基因組范圍內檢測與目標性狀相關聯的位點與基因,然后對候選基因進行功能注釋,進一步探究控制目標性狀的基因及其分子機制。
(二)BSA整體實驗+測序分析路線
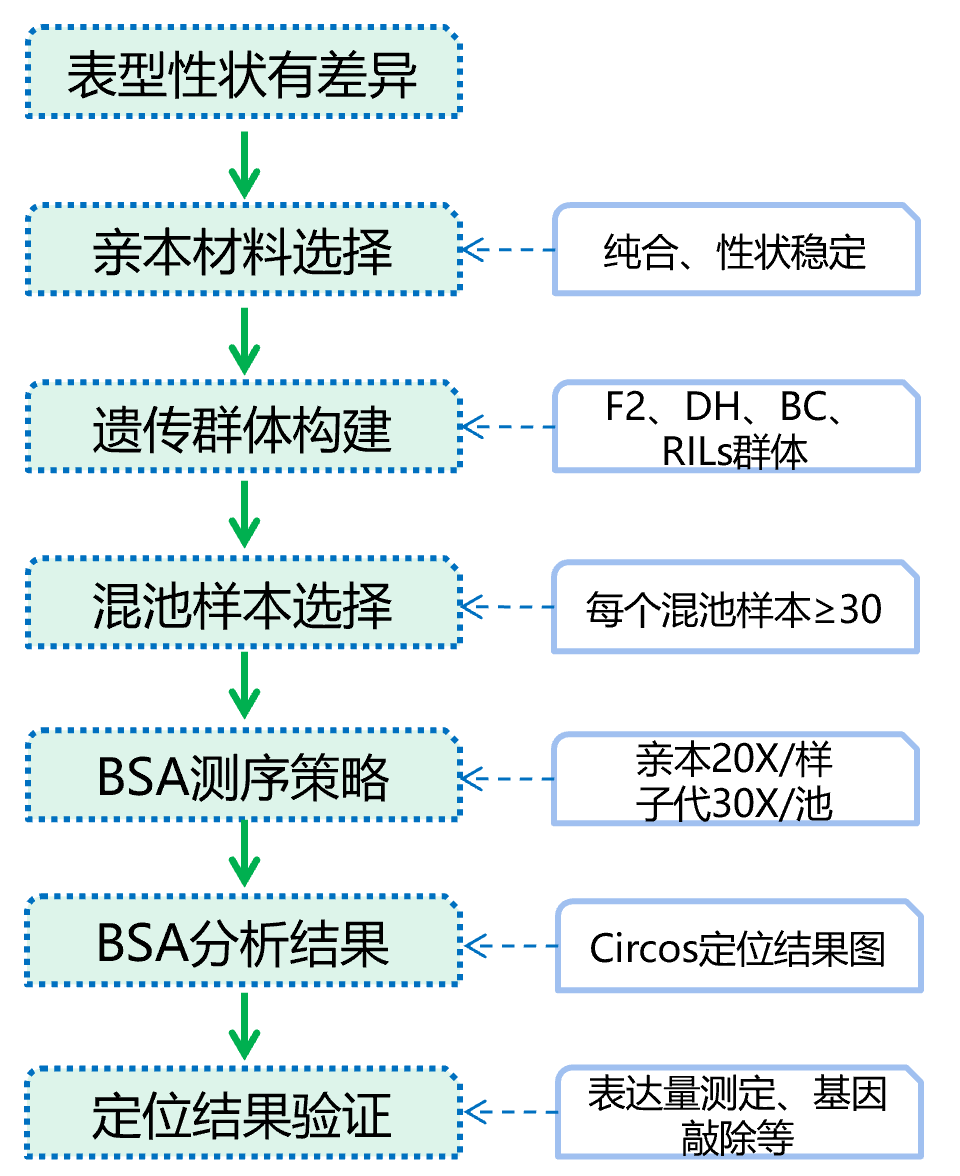
(三)派森諾BSA云平臺優勢
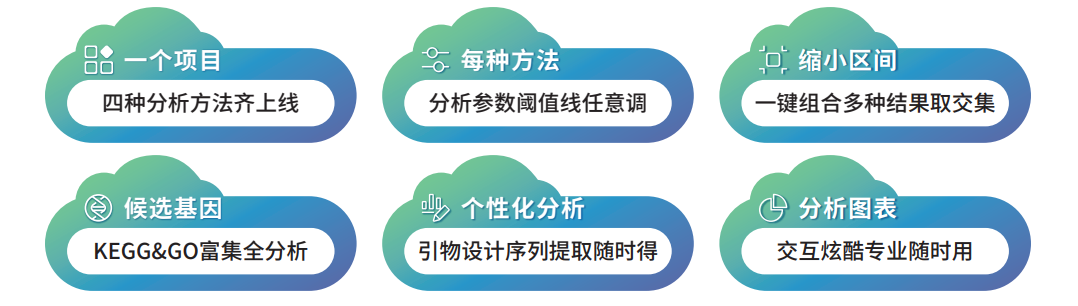
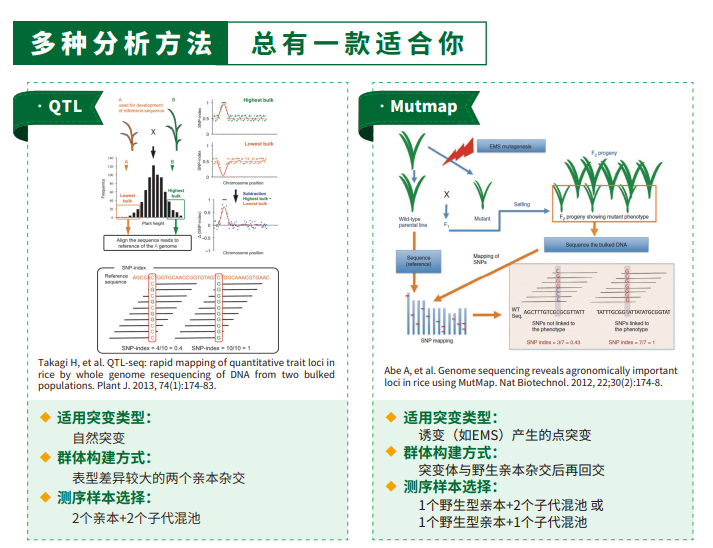
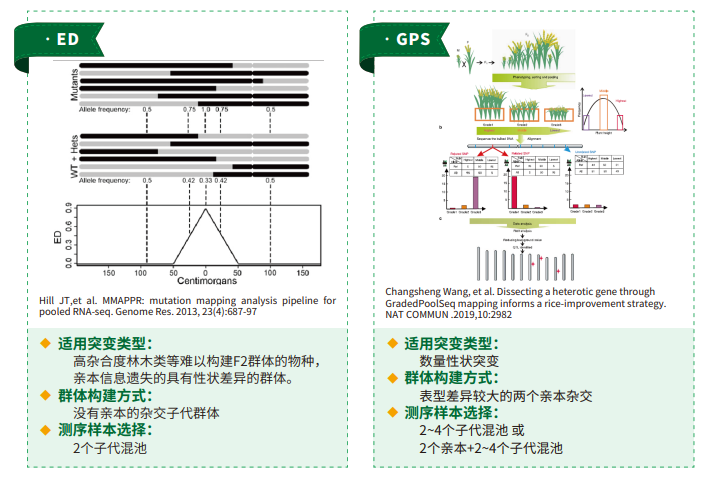
派森諾BSA性狀定位也擁有云平臺,聚力四種分析方法、更有自定義分析,一鍵幫您解鎖性狀定位的隱藏關卡!實現數據上傳、參數自主調整、多種方法在線交并集、目標區域序列提取、引物設計......眾多個性化分析服務,滿足您的定位需求。
本文章的BC4&BC6兩個群體的BSA性狀定位項目工作由上海派森諾生物科技股份有限公司完成。如需進一步討論,歡迎發郵件或者致電我們喲(郵箱地址:[email protected],聯系電話:021-64502808-8203)!
再次恭喜巫永睿老師恭喜黃永財博士!!十年磨一劍,出鞘天下知,從Plant Cell到Nature派森諾動植物基因組測序服務一路相伴!期待與更多的老師長期合作。
文章索引:
Huang,Y., Wang,H., Zhu,Y. et al. THP9 enhances seed protein content and nitrogen-use efficiency in maize. Nature(2022). doi.org/10.1038/s41586-022-05441-2.


