
2021-07-13

期刊:Molecular Ecology Resources
影響因子:7.093
文章題目:Single-molecule long-read sequencing reveals extensive genomic and transcriptomic variation between maize and its wild relative teosinte (Zea mays ssp. parviglumis)
技術手段:二代轉錄組測序、全長轉錄組測序、基因組測序
派森諾生物與華中農業大學攜手合作,于近期在Molecular Ecology Resources上發表了大芻草全長轉錄組的研究成果。
研究背景
玉米(Zea mays L.)是世界重要的糧食作物,目前,隨著人口增加和耕地減少,通過利用野生種質資源進行作物遺傳改良,進而提高作物產量勢在必行。大芻草(teosinte,Zea mays ssp. parviglumis),又名墨西哥野玉米,是現代栽培玉米的野生近緣種,它具有抗病性、直立性等優良特性,且可以與玉米自由雜交,是改良現代栽培玉米的重要種質資源。
但是,由于大芻草具有高度雜合、重復性高的基因組,給組裝帶來了困難,因此至今無可利用的高質量參考基因組。同時,由于缺乏合適的注釋工具,大芻草的全長轉錄組也無法得到很好的組裝,這阻礙了在玉米的育種中鑒定和利用有利的衍生等位基因。
轉座子(transposable element,TE)是重要的基因組元素,或與基因組結構變異有關,在植物的適應性進化中起著關鍵作用。有研究表明,TE在植物中發揮著重要作用,涉及基因失活,基因表達的調節,對外界應激變化的反應等。近85%的玉米基因組由TE組成,但其在玉米馴化中的作用仍不明確。
基于此,本研究對大芻草近交系TIL11進行全長轉錄組測序,通過自行設計的注釋工具:DenovoAS_Finder,用以在無參情況下將轉錄本進行分類,以獲得TIL11的準確轉錄組信息。隨后利用Nanopore測序技術構建TIL11的基因組草圖以鑒定TE,旨在加深對玉米馴化與改良的理解。
技術路線圖
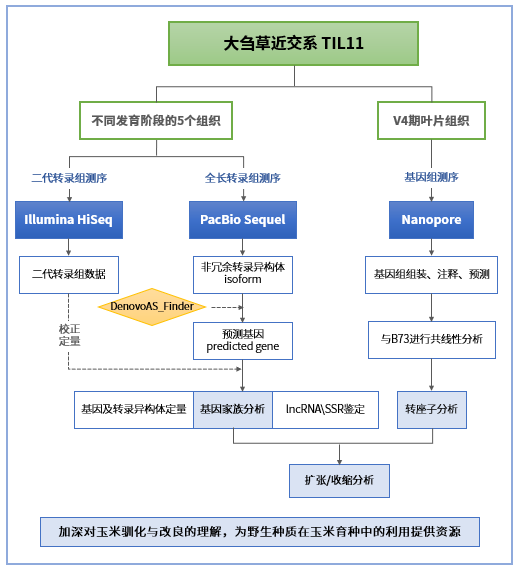
研究結果
1、 TIL11全長轉錄組的測序和注釋結果
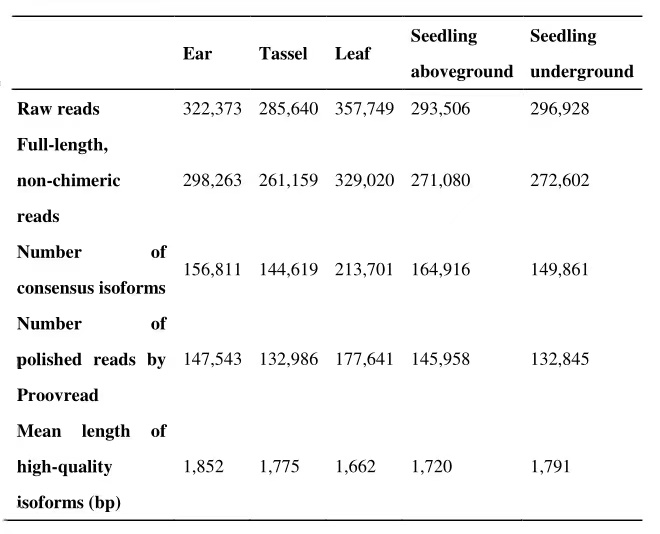
為構建大芻草全長轉錄組,從大芻草自交系TIL11不同發育階段的五個組織中提取高質量的RNA,并通過Pacbio Sequel測序產生1,556,196條reads。經過SMRT分析獲得829,908條一致的全長轉錄異構體(isoform)。利用illumina的二代測序數據對其進行矯正,最終合并得到70,024條非冗余轉錄異構體(表1)。
表1 TIL11轉錄組組裝情況統計
接下來設計了一個基于機器學習的注釋工具:DenovoAS_Finder,在沒有參考基因組的前提下對全長轉錄組進行注釋。該工具利用XGBoost分類方法,利用玉米B73的公開數據進行測試表明其注釋準確率高達91%(圖1)。該工具將TIL11的70,044條轉錄異構體注釋為23,527個預測基因(圖2 a),組裝出的TIL11參考轉錄組與其近緣栽培玉米相當:基因在端粒區域較多,著絲粒周圍區域較少,轉錄本的平均長度為1906 bp,比B73短;此外還鑒定了17,339個SSRs,9,904個lncRNAs;跨組織的轉錄組測序結果顯示,大多數基因和轉錄本在葉片中表達,而在雄穗中表達得最少(圖2 b,c)。
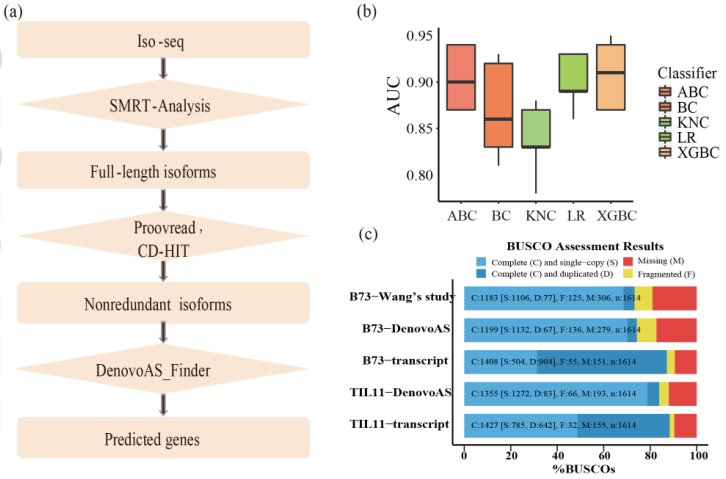
圖1 用集成機器學習的軟件:DenovoAS_Finder來注釋TIL11全長轉錄組
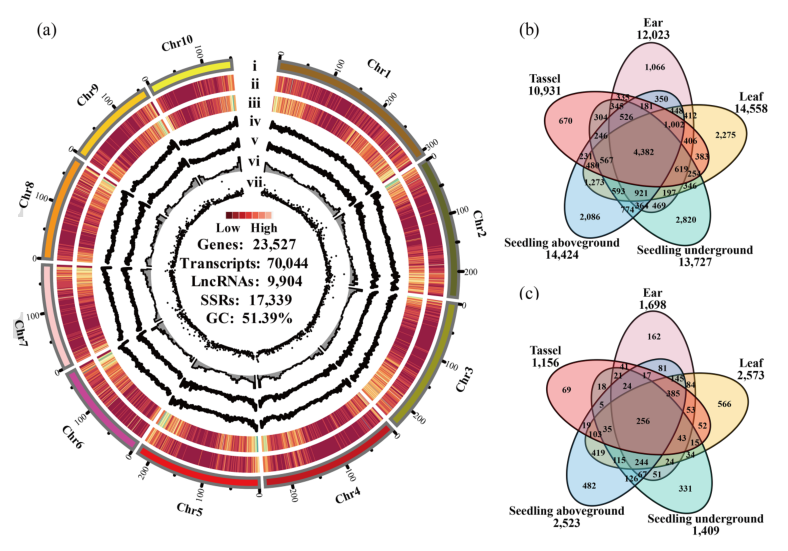
圖2 TIL11轉錄本的特征和組織特異性表達
2、 基于全長轉錄組的轉錄異構體和基因家族分析
提取基于全長轉錄組的TIL11和B73基因家族信息發現,相比大芻草,玉米有142個基因家族顯著擴張,30個基因家族顯著收縮(圖3 a)。進一步進行GO富集分析,結果表明,擴張的基因家族在“RNA修飾途徑”條目顯著富集(圖3 b),暗示它們在玉米馴化轉錄修飾中起重要作用。
以往針對玉米與其近緣種的轉錄組研究主要集中在基因表達水平,例如,大芻草和玉米之間的基因表達不一致,不同的表達模式與馴化和改良顯著相關等。而本研究專注于轉錄異構體的數量,并發現了TIL11基因和B73基因之間的異構體數量變化較為常見。例如,比較基因家族中每個基因產生的轉錄異構體數量,發現快速擴張的基因家族中,平均轉錄本顯著增加,反之快速收縮的基因家族的平均轉錄本顯著減少(圖3 c)。
進一步研究發現,轉錄本數量發生顯著變化的基因在馴化和改良相關基因中顯著富集。比較TIL11與B73的同源基因對的轉錄異構體數量,并將異構體數量增加最多的前5%的基因被命名為擴張組。已有研究鑒定,在玉米的馴化和改良過程中,分別有1429和1242個基因經歷了顯著選擇。本次結果中,擴張組包含了馴化期經歷選擇的59個基因和改良期經歷選擇的74個基因,且富集程度達到了顯著水平。這表明在大芻草和玉米之間,轉錄異構體數量增加最多的基因與選擇有關(圖3 f)。
以上結果均表明,玉米的馴化和改良不僅影響基因表達水平,而且影響轉錄異構體的形式。
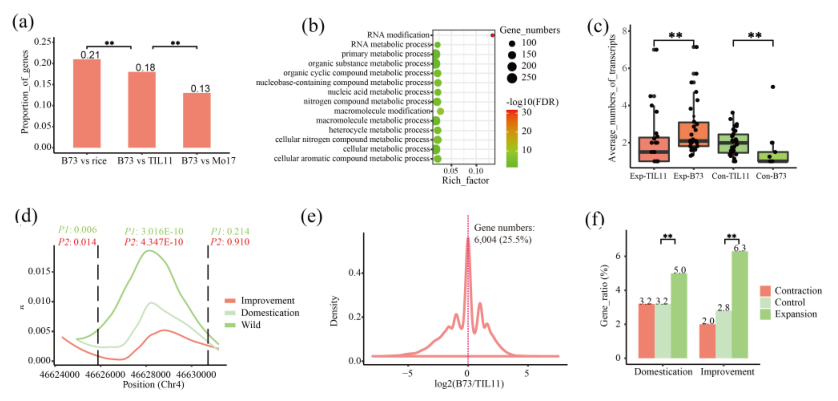
圖3 基因家族和轉錄本數量的變化
3、 基因組草圖的組裝和轉座子分析
為構建大芻草的基因組草圖,通過Nanopore測序得到了29.6 G數據,總計2,429,054條reads。組裝出的TIL11基因組大小約1.29 G,由16,633個高質量contigs組成,N50為112 kb。預計TIL11完整基因組的大小2.08 G,小于B73基因組(表2)。
表2 TIL11基因組組裝情況和預測統計

基因組共線性分析表明,TIL11基因和B73基因之間具有良好的保守性(圖4 a)。與之相對TIL11和B73之間的長末端重復轉座子(LTRs)存在顯著差異。具體而言,在玉米B73中,I類逆轉錄轉座子占期基因組變異的最大比例(62.8%),然而,TIL11中可歸因于這類轉座子的基因組變異比例顯著低于B73,僅47.2%。進一步研究發現,擴張和收縮的基因組區域主要在這類轉座子 (圖4 b),詳細的比較表明,基因組發生擴張的區域幾乎都是LTRs(圖4 c)。這說明LTRs的轉座可能促進了兩個物種之間的基因含量差異。
同時,觀察到轉座子Hopscotch對基因的表達水平起重要作用。在玉米馴化過程中,基因tb1對頂端優勢的增加起著重要作用,而據報道,插入tb1調控區的轉座子:Hopscotch可作為基因表達的增強子。本研究中,該轉座子在B73的相同位置被鑒定,但在TIL11基因組中缺乏(圖4 d),因此導致了TIL11中tb1表達量較低。
這些結果表明轉座子可能是馴化和改良過程中基因組結構變異和系統發育變化的重要因素。
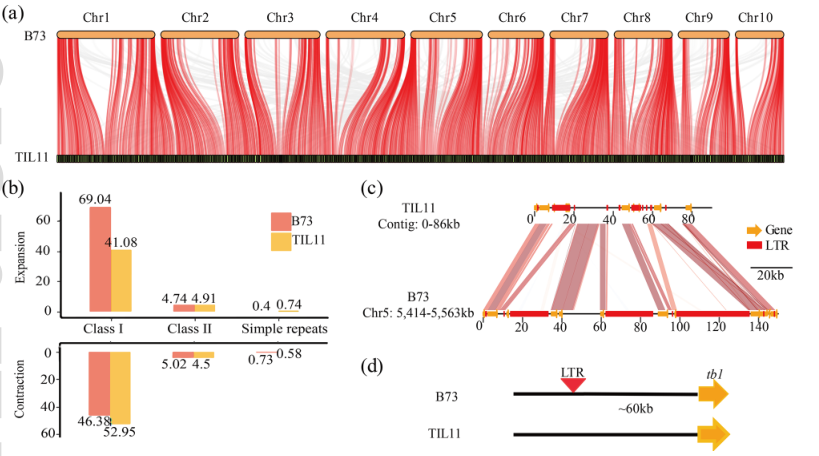
圖4 B73和TIL11之間基因組區域的收縮和擴張
小 結
A.本研究對大芻草近交系TIL11的五個組織進行全長轉錄組測序,鑒定了70,044條非冗余轉錄異構體,并設計了一個基于機器學習的軟件:DenovoAS_Finder,用于在沒有參考基因組的情況下注釋TIL11的轉錄組。
B.此外,通過Nanopore測序,構建了具有16,633個高質量contigs、 N50達112kb的TIL11基因組草圖。
C.與大芻草相比,在玉米中擴張的基因家族顯著富集于“RNA修飾途徑”這一GO條目下,且TIL11的轉錄本數量多于玉米自交系B73。
D.基因在TIL11和B73之間表現出共線性,基因間區被轉座子廣泛改變。
E.本研究加深了對玉米馴化的理解,為野生種質在玉米育種中的利用提供了資源。
本研究的二代轉錄組、全長轉錄組建庫測序由上海派森諾生物科技有限公司完成。
原文索引:
Li Z , Han L , Luo Z , et al. Single-molecule long-read sequencing reveals extensive genomic and transcriptomic variation between maize and its wild relative teosinte (Zea mays ssp. parviglumis). Molecular Ecology Resources, 2021.