2021-06-28

期刊:《Theoretical and Applied Genetics》
影響因子:4.439
如果您的材料純合致死或者不育,不妨看看本文;
如果您的定位因找不到標記走進死胡同,千萬不要錯過本文!
近日,派森諾生物與上海交通大學攜手在植物育種研究雜志《Theoretical and Applied Genetics》(IF:4.439)發表新一期研究成果!
1、研究背景
花是被子植物重要的生殖器官,是被子植物進化過程中比較復雜的結構。黃瓜(Cucumis sativus L.)由于其器官發生、發育、結構的復雜性,是研究花發育的一個模型。花和卷須是黃瓜重要的農藝和產量性狀,該研究利用BSA的方法首次克隆和解析了與黃瓜花和卷須性狀相關的基因,為花和卷須這兩個器官的產生過程提供了分子基礎。

圖1 野生型(A表示)與突變體(B表示)的表型特征:A1與B1為成熟植株,形態上無明顯差異;但在雄花(A2與B2)、卷須(A3與B3)、果實(A4與B4)、雌蕊(A5與B5)、雄蕊(A6與B6)雄芽(A7與B7)有明顯差異。
2、技術路線圖
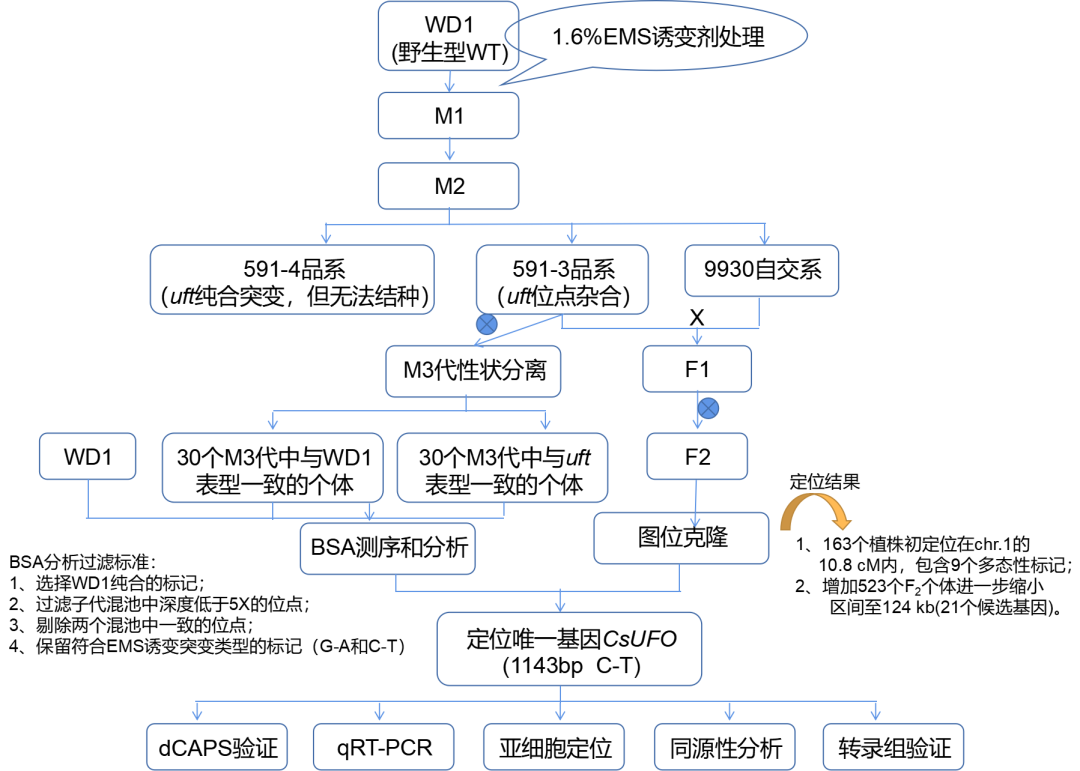
3、BSA候選區間的精細定位
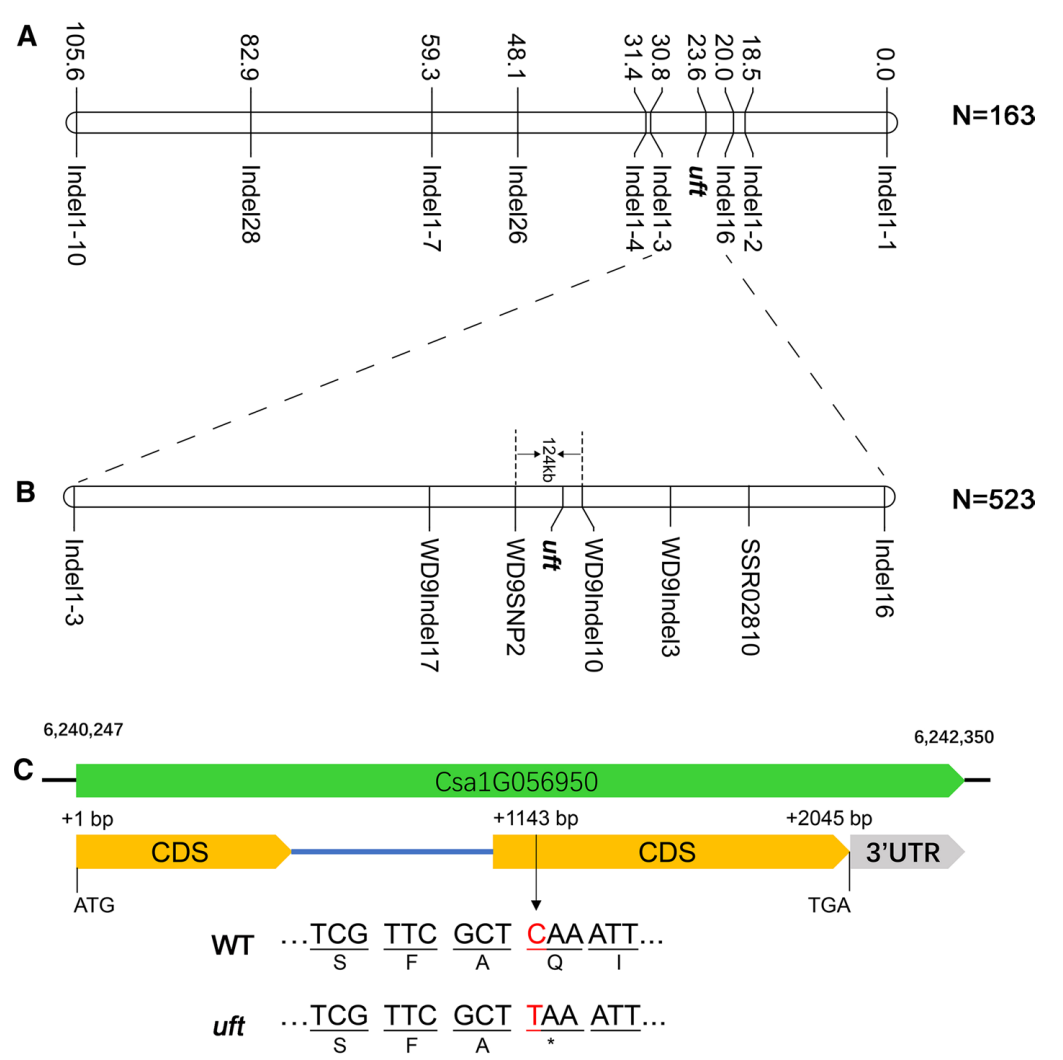
通過已發表文獻篩選出100個SSR和60個Indel分子標記,利用9930與591-3構建的F2群體,選擇兩個親本加163個子代和上面的分子標記信息然后構建遺傳圖譜,初定位到Chr.1上的20.0cM到30.8cM之間(~2.3Mb),該區間內包含有9個多態性標記,在該候選區間內進一步開發3個indel、1個SNP、1個SSR多態標記,把這5個分子標記增加上一共是165個分子標記,同時選擇另外一個F2群體再次進行遺傳圖譜構建,QTL定位到6,190,660 bp-6,313,980 bp(~124kb),該區間內有21個候選基因。
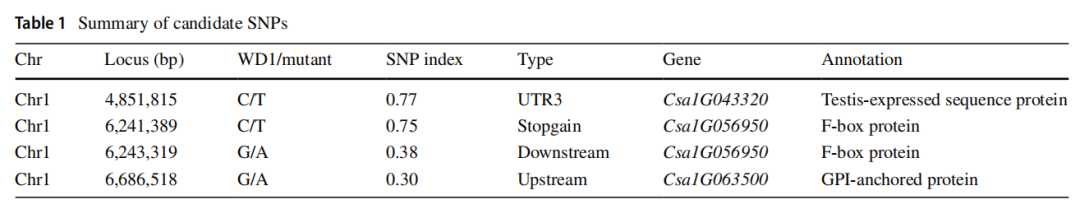
通過591-3自交后代中的30個野生型表型的個體DNA混池和30個uft表型的個體DNA混池,再加WD1親本進行高通量測序,基于MutMap+分析策略在候選區間內獲得4個候選SNP位點,進一步分析確定了候選標記SNP6241389。具體定位結果見表1。
由此說明BSA定位的準確性很高哦~
表1 BSA定位出的候選SNP位點
4、候選基因CsUFO的驗證
4.1候選基因CsUFO在qRT-PCR層面的驗證
突變導致氨基酸編碼提前終止,與正常基因相比在C端少300個氨基酸。通過5個UFO以及另外13個MADS-box蛋白序列構建進化樹表明UFO在同一科的不同物種中高度保守。qRT-PCR結果表明野生型頂芽中CsUFO高表達,而uft突變體的頂芽中該基因表達顯著下調。
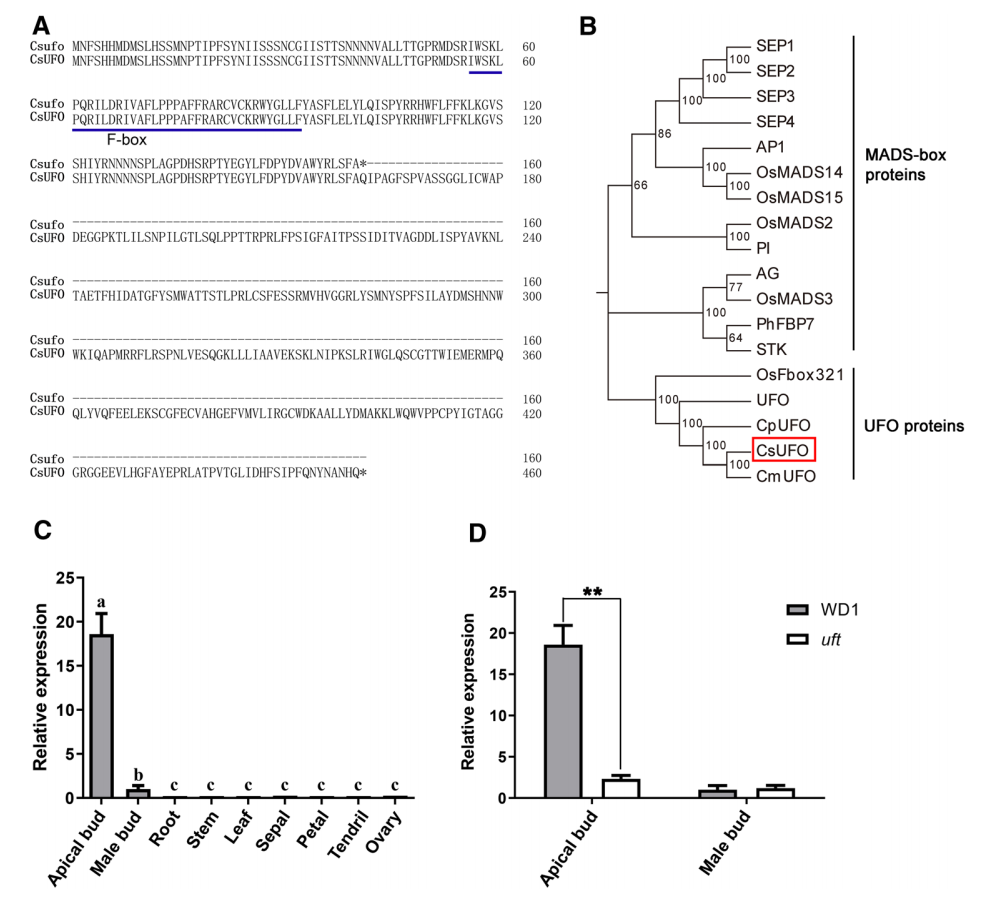
圖3 候選基因CsUFO的結構、保守性與表達分析。
4.2 候選基因CsUFO在轉錄組層面的驗證
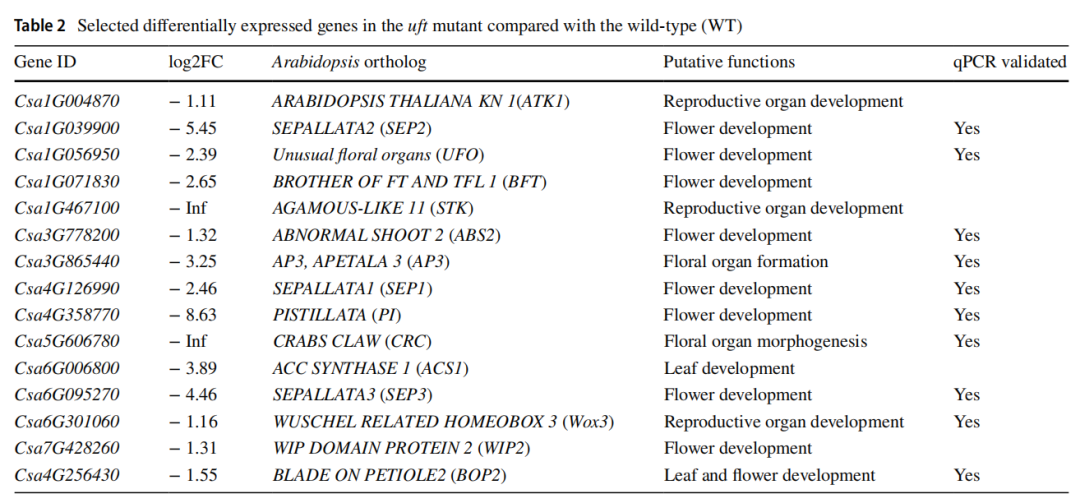
以黃瓜(Chinese Long) V2基因組為參考,對253個DEGs進行了GO富集分析。在最重要的20個GO term中,我們發現多個GO term參與了形態發育,如植物器官的形成、氣孔器官形成和胚胎后植物器官的形態發生。在頂端芽轉錄組中,至少有15個DEGs被鑒定為直接參與花和生殖器官的發育。
表2 突變體與野生型之間有差異表達的基因列表
5、結論
本文通過BSA+圖位克隆定位策略揭示了黃瓜花和卷須(uft)突變是由編碼F-box蛋白的Csa1G056950基因突變引起,利用圖位克隆將uft位點定位到黃瓜1號染色體的一個124 kb區域。基于高通量測序的MutMap+分析則進一步確定了候選區域CsUFO (Csa1G056950)的第二外顯子處有一個SNP突變(C-T),導致氨基酸編碼提前終止。同時,該研究向我們展示了BSA定位的準確性!
本研究的BSA和轉錄組測序分析工作由上海派森諾生物科技有限公司完成。期待您的選擇,發文快人一步!
參考文獻:
Yue Chen, Haifan Wen et al. CsUFO is involved in the formation of flowers and tendrils in cucumber [J] .Theor Appl Genet. 2021 Mar 19. DOI:10.1007/s00122-021-03811-4.
更多項目文章,請關注派森諾官網


