
2020-08-13

期刊:《Journal of Virology》
影響因子:4.501
最近,派森諾與上海海洋大學合作,在《Journal of Virology》發表論文,利用宏基因組測序,探究了上海滴水湖中新型噬病毒體和藻類大病毒的多樣性特點,并發現了以單細胞綠藻為真核宿主的新型Cell-Virus-Virophage(C-V-v)三相互作系統。
大伙可能都聽說過噬菌體,但是噬病毒體是什么呢?生命形式總是如此多樣而又出乎意料,今天就由小編帶領大家,走進噬病毒體的奇妙世界吧!
研究背景
噬病毒體(Virophage)是利用原生生物巨病毒(Giant virus)在真核宿主內形成的病毒工廠(Viral factory)中進行增殖的一類小型雙鏈DNA病毒,它可使巨病毒的正常增殖受到破壞,導致其產生侵染能力下降的畸形病毒顆粒,像是受到了侵染,噬病毒體因此而得名。噬病毒體可伴隨巨病毒一同侵染宿主細胞,而由噬病毒體、巨病毒和真核宿主細胞三者所構成的互作系統,稱之為C-V-v系統。目前,僅有少數噬病毒體如Sputnik,Mavirus,Zamilon等及其C-V-v系統已被發現和初步探究,而廣泛分布于水體環境中的噬病毒體及其C-V-v互作系統仍有待研究。
為進一步探究此類病毒與病毒之間,及病毒與真核宿主之間特殊的互作系統,本研究利用二代高通量測序技術,對滴水湖表層水體中的噬病毒體、藻類大病毒及它們之間的互作關系進行了研究。由于目前發現的噬病毒體都屬于DNA病毒,因此,我們既可以用宏病毒組的方法,富集病毒顆粒后進行測序研究;也可以直接采用宏基因組的方法,通過加大測序深度,從海量測序數據中提取病毒基因組的相關信息,進行深入研究。這樣,就能幫助我們更進一步地探索和理解神奇的噬病毒體啦~
研究內容
① 噬病毒體基因組挖掘與分析;
② 藻類大病毒基因組挖掘與分析;
③ 噬病毒體與藻類大病毒基因關聯分析;
④ 噬病毒體與藻類大病毒基因組特征分析;
⑤ 藻類大病毒中CRISPR-Cas-like系統分析;
研究目的
探究滴水湖中由噬病毒體、藻類大病毒、單細胞真核綠藻所構成的新型C-V-v三相互作系統。
材料與方法
測序技術:Illumina MiSeq高通量測序平臺
測序模式:宏基因組測序
實驗對象:滴水湖表層湖水
采樣方式:通過已滅菌的5 L采水器采集表層水樣(<1 m),于20 L采樣桶中快速混合潤洗后,棄去,再迅速裝滿。經現場測定環境因子后立刻轉移至實驗室冷庫,留待進一步處理。
實驗設計
① 采集滴水湖表層湖水樣品;
② 提取水樣微生物總基因組;
③ 利用Illumina MiSeq高通量測序平臺進行測序和生物信息學分析;
④ 噬病毒體和藻類大病毒的基因組組裝與特征分析;
⑤ 噬病毒體的潛在巨/大病毒宿主分析。
結果與討論
01、滴水湖中新型噬病毒體的發現及其基因組分析
本研究基于負壓過濾水體樣品至0.22 μm孔徑濾膜(已滅菌)的方法,對滴水湖中表層水進行了微生物的富集,總DNA的提取、驗證,并采用Illumina MiSeq高通量測序平臺完成了雙端測序工作(4個run的2×250 bp的Paired-end即PE文庫和2個run的2×250 bp的Mate-paired即MP文庫)。原始下機數據共計49.68Gb,約1億條reads。
如圖1所示,通過序列拼接,本研究從滴水湖宏基因組數據集中共獲得7株新型噬病毒體的全基因組序列,并將其分別命名為Dishui Lake Virophage 2/3/4/5/6/7/8(簡稱:DSLV2/3/4/5/6/7/8),其序列長度分別為31,238、31,512、30,873、26,593、28,714、29,961和26,605 bp。滴水湖噬病毒體(DSLVs)與已知噬病毒體基因關聯分析顯示,DSLVs與已知來源于淡水的噬病毒體共享更多的同源基因,而不與已知分離出來的噬病毒體(Sputnik家族、ALM、Mavirus、CpV-PLVs等)共享任何基因,表明DSLVs與來源于淡水的噬病毒體親緣關系較近(圖2A)。如圖2B所示,系統發育分析進一步表明,DSLVs與來源于淡水的噬病毒體進化關系較近,而與已知分離出來的噬病毒體進化關系較遠,提示它們可能擁有不同的病毒宿主和真核細胞宿主。
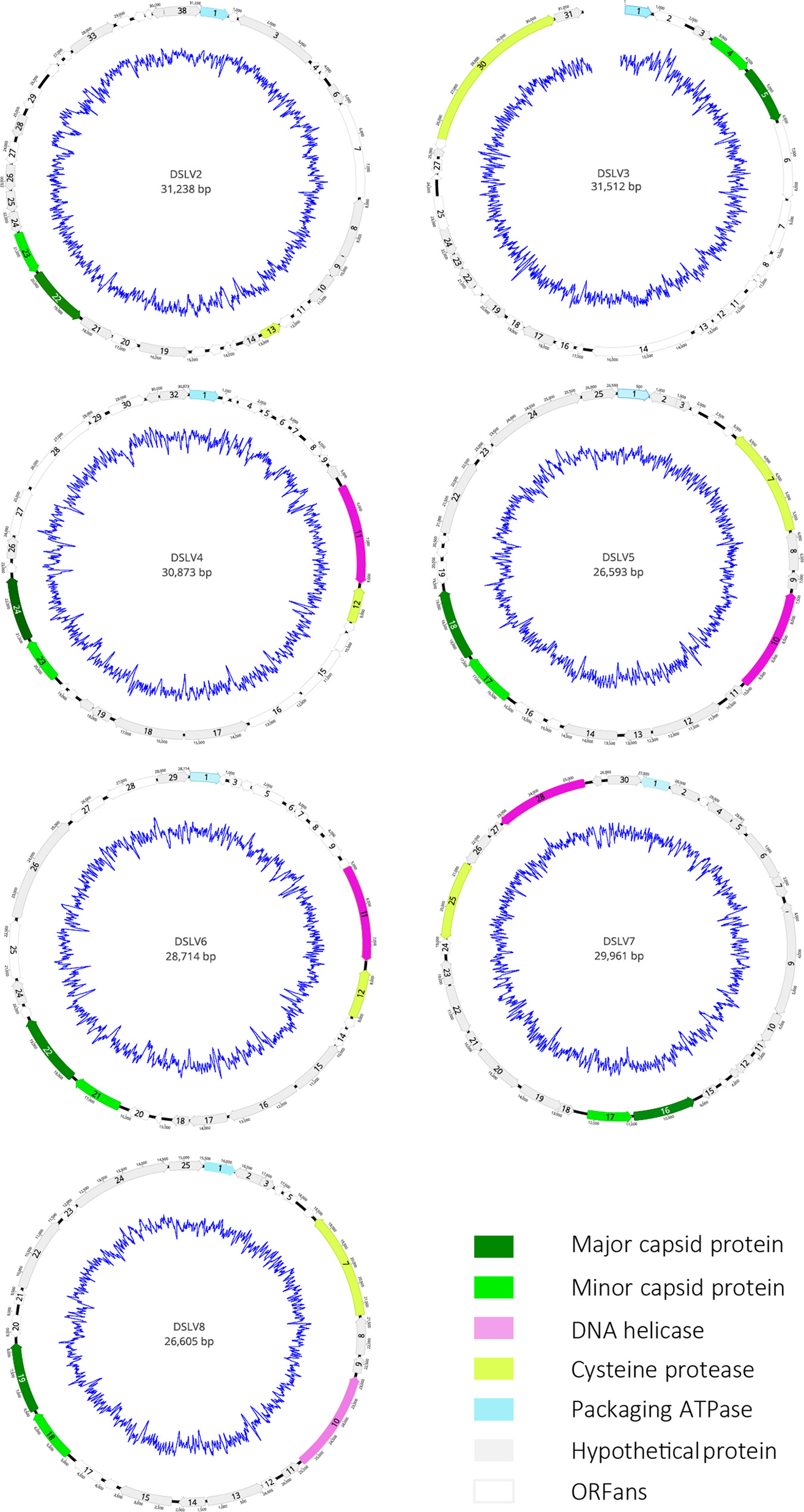
圖1 噬病毒體DSLV2/3/4/5/6/7/8基因組圖譜
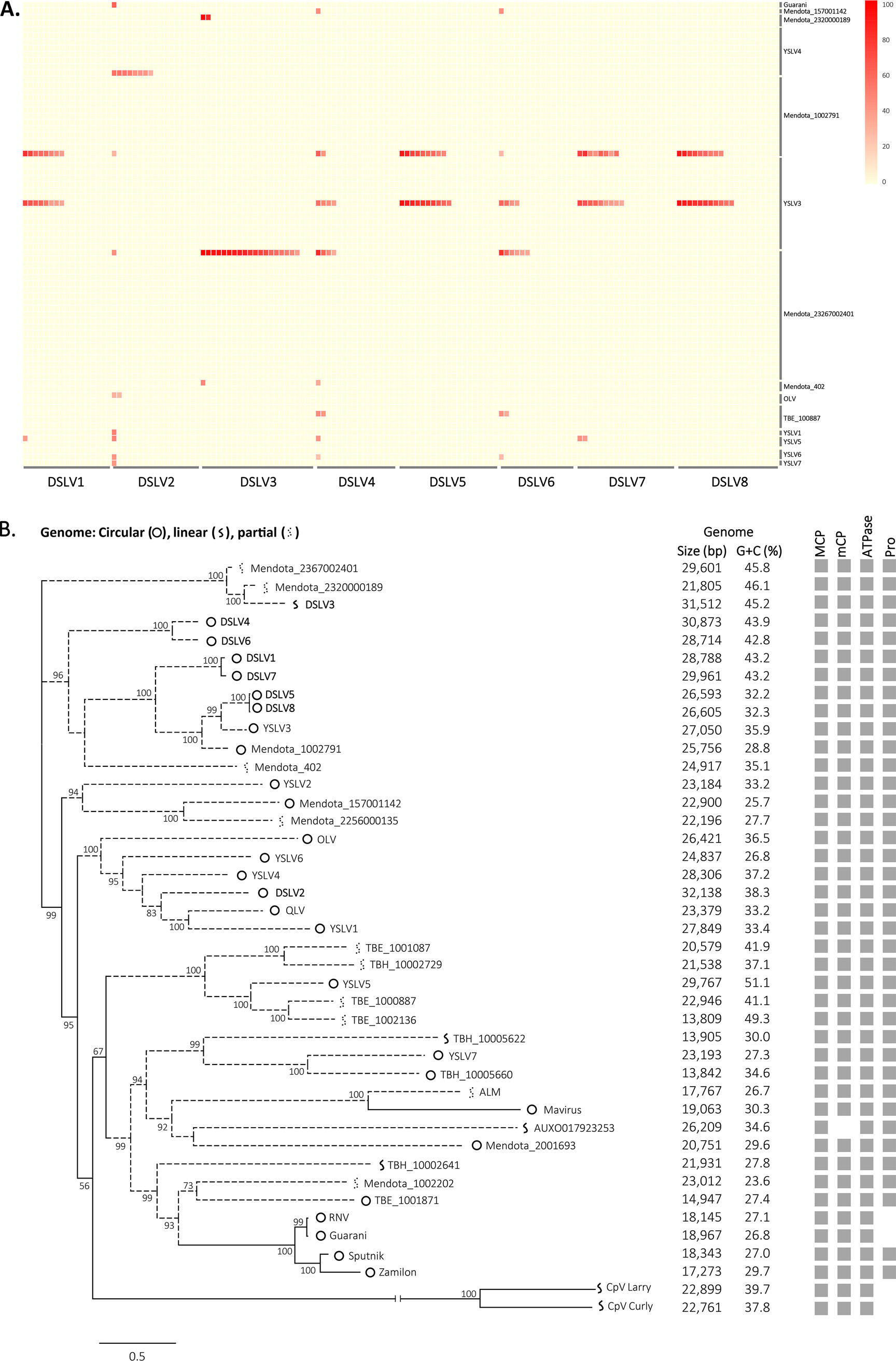
圖2 滴水湖噬病毒體與已知噬病毒體基因關聯分析(A)及系統發育分析(B)
02、滴水湖中新型藻類大病毒的發現及其基因組分析
通過相關序列的調取和基因組的組裝、注釋工作,作者從宏基因組數據集中共獲得了四條完整的藻類大病毒基因組,大小為190,426,194,169,193,666和392,531 bp,并分別將它們命名為Dishui Lake phycodnavirus 2/3/4(簡稱:DSLPV2/3/4)以及Dishui Lake large alga virus 1(簡稱:DSLLAV1),如圖3、圖4A所示。其中DSLPV2/3為線性基因組,分別編碼有247和236個ORFs;而DSLPV4與DSLLAV1為環狀基因組,分別編碼有220和366個ORFs。通過基因注釋發現,DSLPV2/3/4中分別有164、190、149個ORFs與綠藻大病毒科Phycodnaviridae中Prasinavirus所編碼的基因具有同源性,表明它們與Prasinavirus具有較密切的親緣關系,而在DSLLAV1中僅有104個ORFs與病毒編碼的基因具有同源性,其中有接近半數(44個)的ORFs與Mimiviridae第三分支(GroupIII)中的藻類大病毒具有同源性,且與其中的綠藻大病毒Tetraselmis virus 1(TetV-1)共享最多的同源基因(16個,圖4B),表明DSLLAV1與藻類大病毒中的綠藻大病毒具有較密切的親緣關系。
系統發育分析表明,在進化地位上,DSLPV2/3/4同樣與綠藻大病毒Prasinovirus屬距離較近,而DSLLAV1則作為Mimiviridae家族外支與Mimiviridae的距離較近,表明DSLLAV1是一類進化地位較為特殊的綠藻大病毒,并且滴水湖中藻類大病毒具有一定的多樣性。
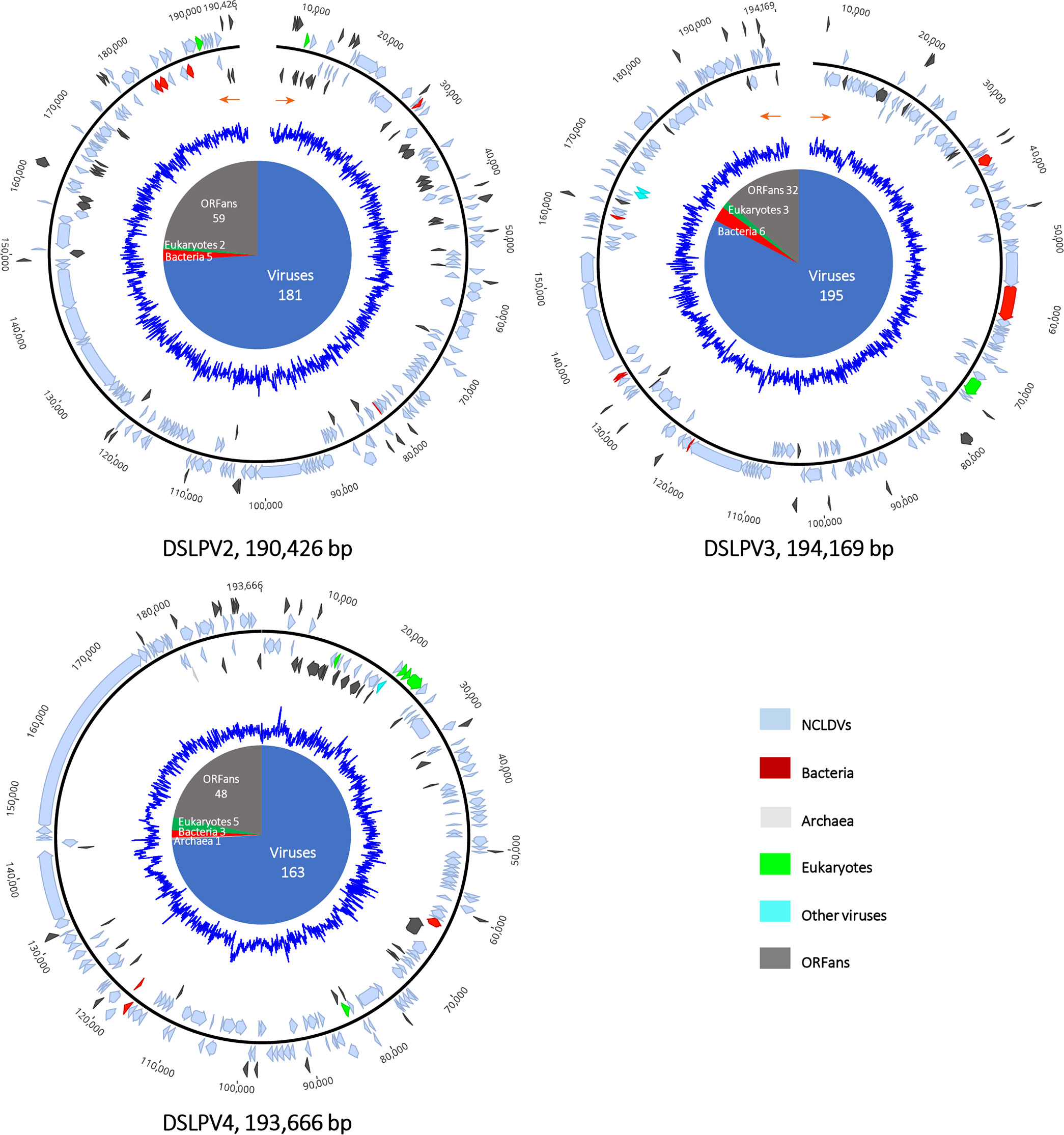
圖3 藻類大病毒DSLPVs基因組圖譜
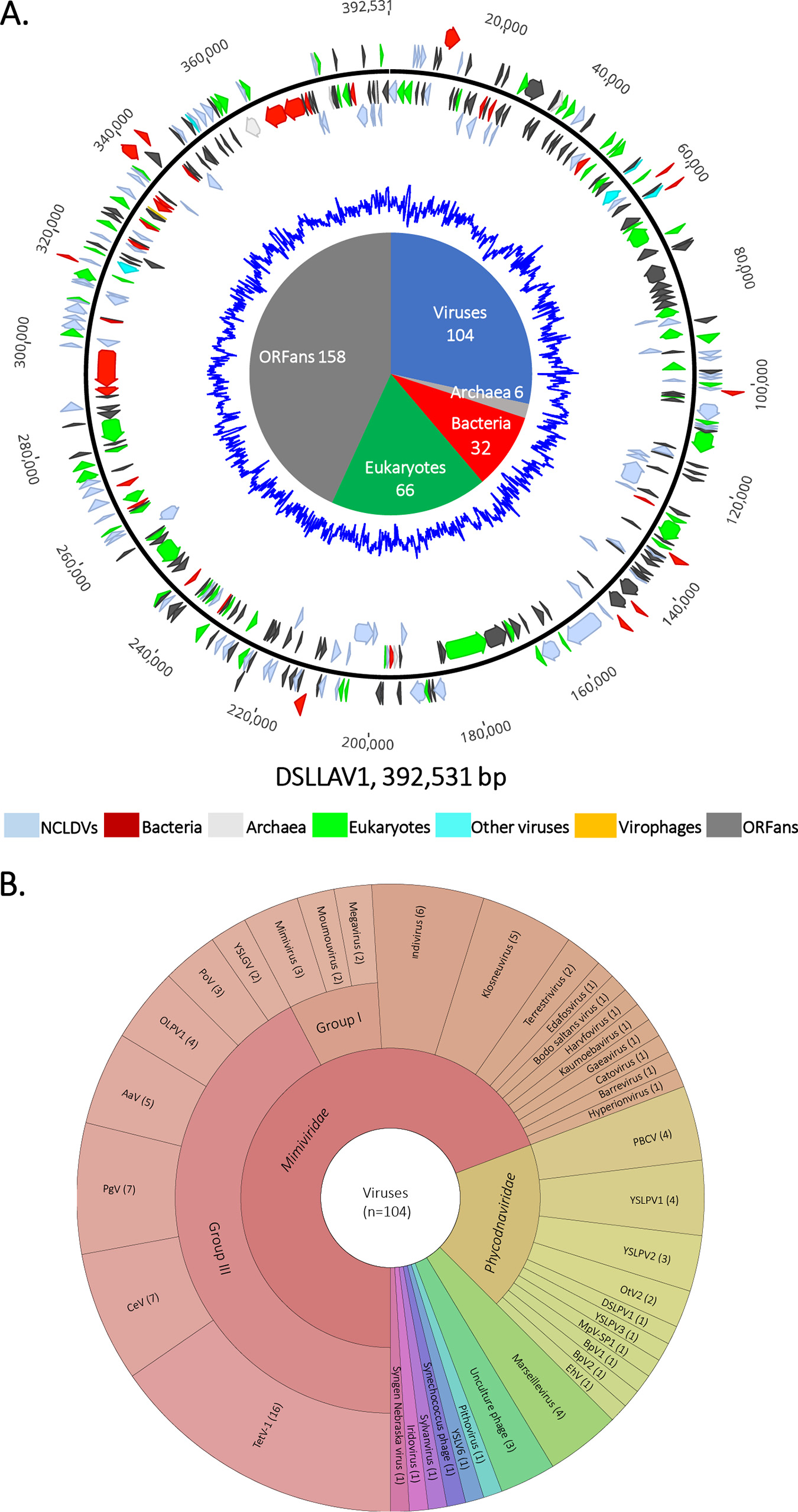
圖4 藻類大病毒DSLLAV1基因組圖譜(A)和與病毒相關的基因注釋信息分類(B)
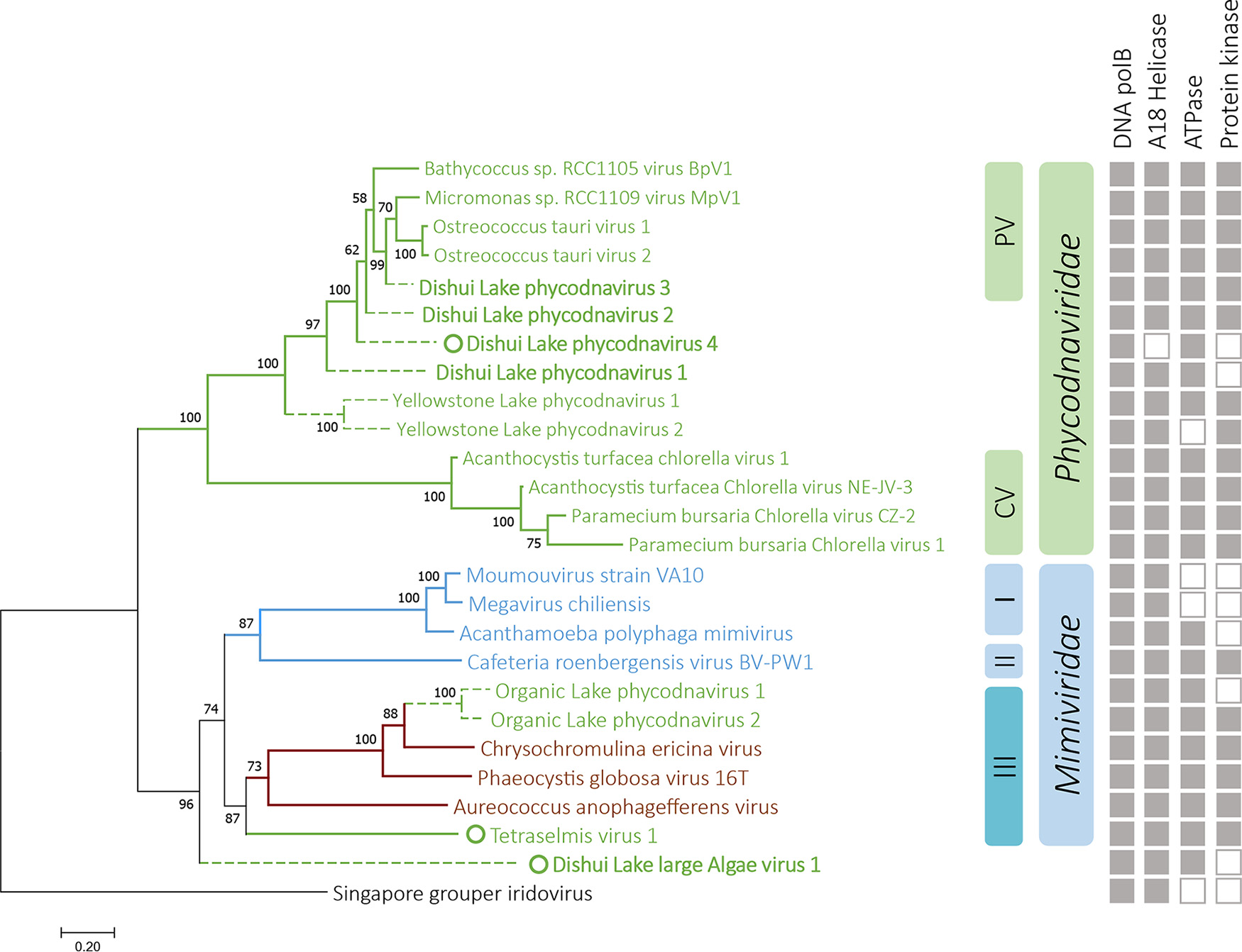
圖5 DSLPVs與DSLLAV1系統發育分析
03、噬病毒體與巨/大病毒基因關聯性分析
圖6中將噬病毒體與巨/大病毒共享的同源基因進行梳理,揭示噬病毒體與其潛在病毒宿主在基因水平上的關聯性。以圖6B中已建立的C-V-v系統為對照,其中噬病毒體均與其宿主巨病毒具有較好的基因關聯,如Sputnik與Mamavirus,Mavirus與Cafeteria roenbergensis virus(CroV)等,且同源基因的進一步系統發育分析同樣支持這種關聯性(圖6D)。通過分析發現,滴水湖中噬病毒體與藻類大病毒尤其是綠藻大病毒具有更好基因關聯,如DSLV1/4/6/7與Paramecium bursaria Chlorella virus(PBCV)(圖6A),并通過共享同源基因的系統發育分析重現了這種關聯性(圖6C)。由此表明,滴水湖噬病毒體可能以單細胞綠藻大病毒為病毒宿主并與其一同侵染綠藻細胞。
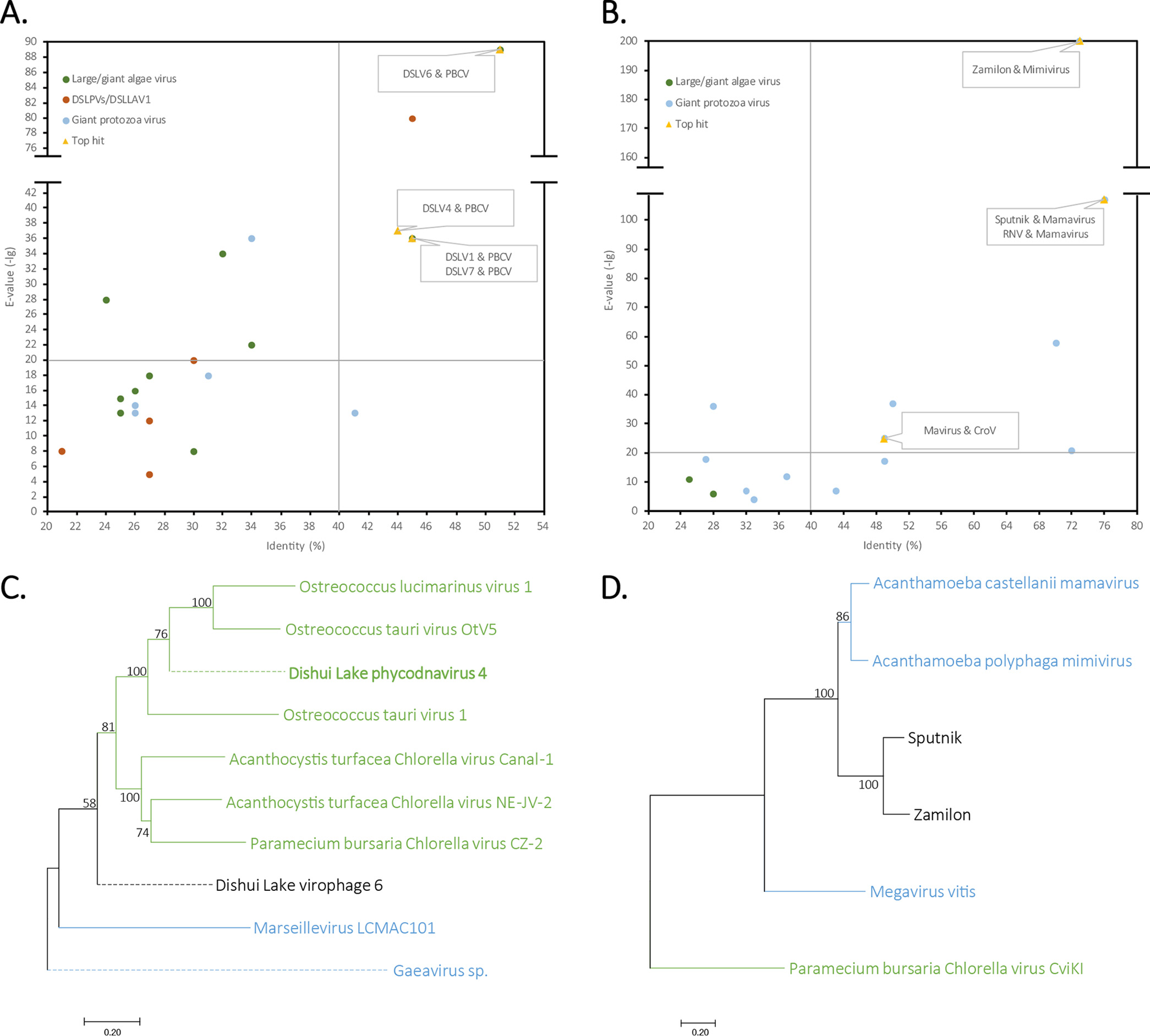
圖6 噬病毒體與巨/大病毒的基因關聯性
04、噬病毒體與巨/大病毒基因組特征分析
如圖7A所示,八個滴水湖噬病毒體共被分為兩組。其中,DSLV1-4、4-7的密碼子使用模式不僅與侵染綠藻的PBCV-1和TetV-1非常相似,也與Prasinovirus屬的其他綠藻病毒、DSLPV4和DSLLAV1非常相似。其中,DSLV5和DSLV8的密碼子使用模式則與侵染原生動物的巨病毒(Mimiviridae Group I and II)和侵染藻類的巨/大病毒(Mimiviridae Group III)更為相似。和預期的一樣,在對照組中,Sputnik、Zamilon 和Mavirus則分別與它們的宿主病毒Acanthamoeba castellanii mamavirus9(ACMV)和CroV聚在一起,驗證了該分析方法的可靠性。綜合所有結果表明,侵染綠藻的大病毒包括在滴水湖中發現的綠藻大病毒(如:DSLPV4,DSLLAV1及它們的近親病毒)更可能是滴水湖噬病毒體的潛在病毒宿主。
與密碼子相對使用頻率分析結果一致,寡核苷酸相對頻率分析結果同樣表明,侵染綠藻的大病毒更可能是滴水湖噬病毒體的潛在病毒宿主。如圖7B、C所示,DSLVs的二核苷酸相對頻率模式與侵染綠藻的大病毒(如PBCV-1、TetV-1、DSLPV4和DSLLAV1)非常相似,但顯然不同于對照組中侵染原生動物的巨病毒(如ACMV和CroV)。
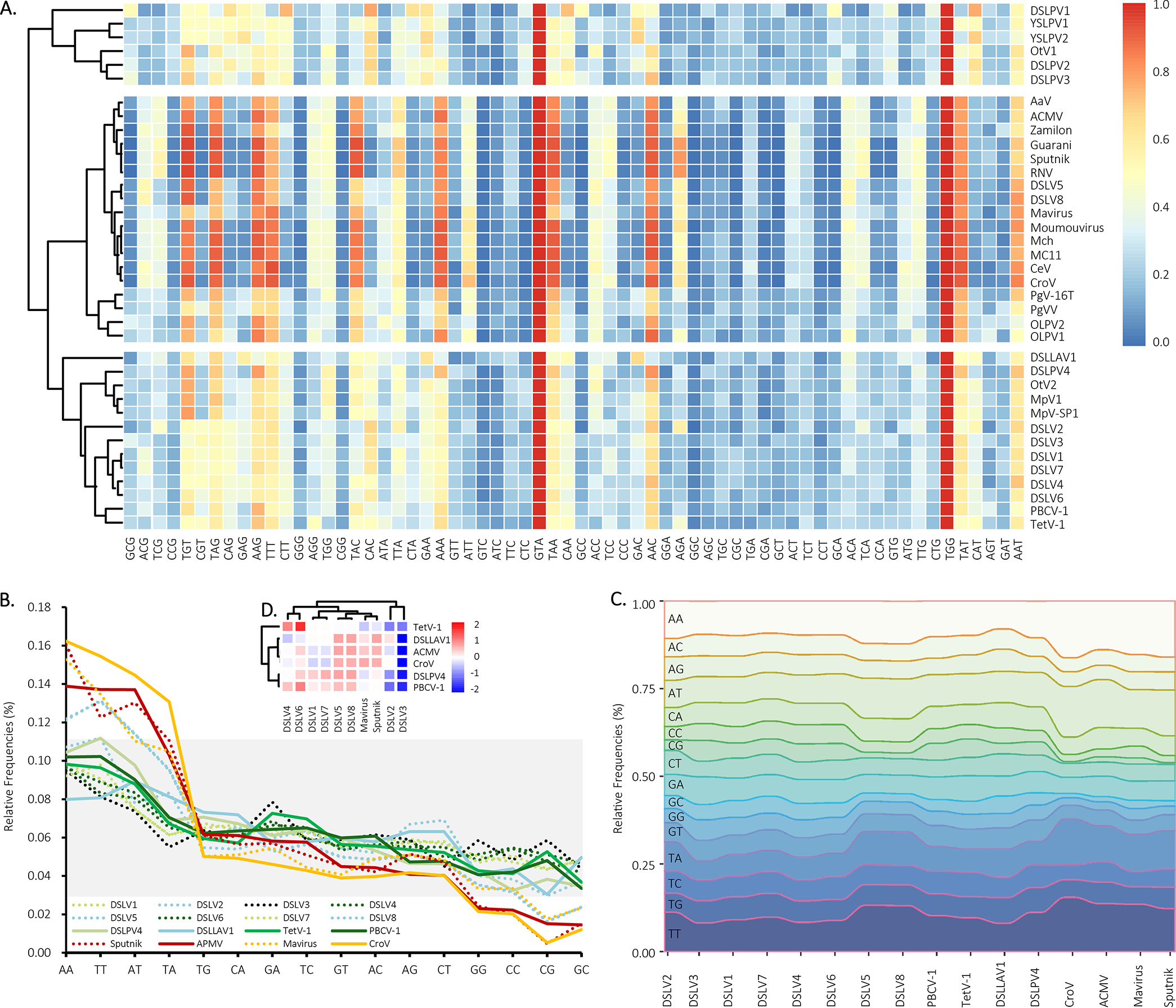
圖7 噬病毒體與巨/大病毒之間的基因組特征分析
05、滴水湖大病毒(DSLLAV1)CRISPR-Cas-like系統分析
如圖8A所示,分別來自DSLV5 ORF24和DSLV8 ORF24的長度為20 bp的核酸序列片段匹配(DSLV5一個錯配,DSLV8無錯配)到DSLLAV1基因組上(ORF142)。同時在匹配處的上游和下游存在47 bp的重復序列,且在這個重復序列中,另有8 bp在47 bp中重復了4次,在20bp中重復了2次。此外,在DSLLAV1的ORF142上游分別發現了可編碼類Cas4和Cas9蛋白的基因。綜上所述,本研究認為,DSLLAV1擁有一個可以防御噬病毒體DSLV5和DSLV8侵染的類CRISPR-Cas的系統,并將其命名為藻類大病毒噬病毒體防御系統LAVVIRE(Large algae virus virophage resistance element)。
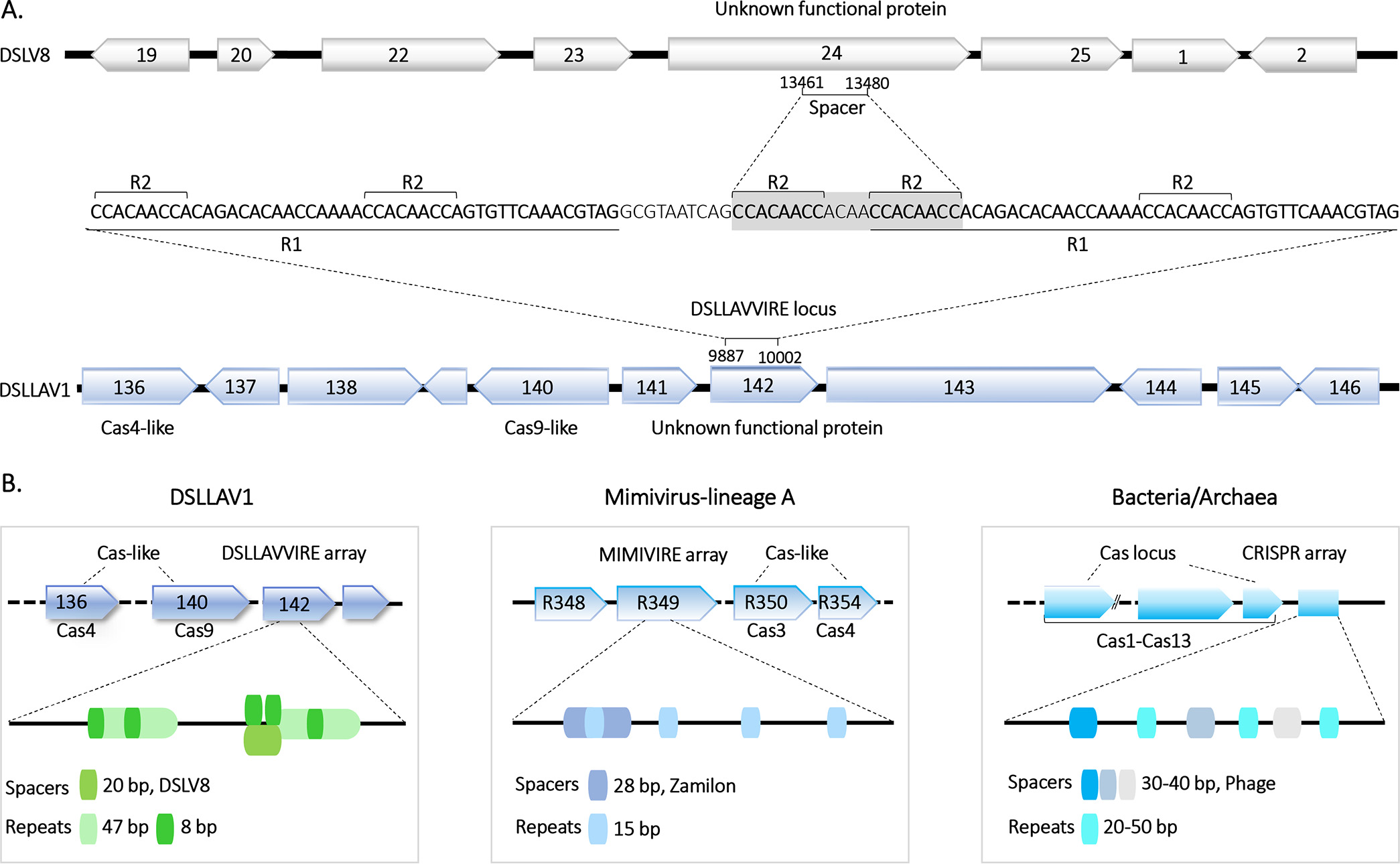
圖8 DSLLAV1中的CRISPR-Cas-like系統
06、滴水湖中新型噬病毒體—綠藻大病毒—單細胞真核綠藻三相互作系統
目前已分離的噬病毒體(如Sputnik、Zamilon等)必須依賴宿主巨病毒才能進行正常增殖,并且噬病毒體與其宿主巨病毒共享相似度較高的同源基因。本文通過比較基因組學分析發現,滴水湖中噬病毒體與綠藻大病毒共享相似度較高的同源基因,并進一步通過基因組特征分析揭示了滴水湖噬病毒體與綠藻大病毒之間存在的共同進化關系。在DSLLAV1基因組中發現的抵抗噬病毒DSLV5/8侵染的CRISPR-Cas-like系統再次證實了噬病毒體與綠藻大病毒由來已久的互作關系,所有分析表明滴水湖中噬病毒體與綠藻大病毒、單細胞真核綠藻共同構成了新型Cell-Virus-Virophage三相互作系統。
總結
本研究通過水體宏基因組學的分析方法,探究了上海市滴水湖表層湖水中噬病毒體和藻類大病毒的多樣性及其互作關系。通過病毒序列的調取及基因組序列拼接,共獲得了多條新型噬病毒體和藻類大病毒的基因組序列,并綜合運用基因關聯分析(Genetic link)、基因組特征分析(Genomic signature)和CRISPR-Cas系統(CRISPR-Cas system)分析等方法,揭示了滴水湖中新型噬病毒體與綠藻大病毒、單細胞真核綠藻之間的三相互作侵染系統。
本研究的測序工作由上海派森諾生物科技股份有限公司完成。
文章索引:
Xu S, Zhou L, Liang X, et al. Novel Cell-Virus-Virophage Tripartite Infection Systems Discovered in the Freshwater Lake Dishui Lake in Shanghai, China[J]. Journal of Virology, 2020, 94(11).
原文鏈接:https://jvi.asm.org/content/94/11/e00149-20