2019-04-09
2019年2月,派森諾生物與中國科學院海洋研究所合作發表了石鯛魚基因組測序組裝與注釋的文章。該研究使用二、三代測序結合Hi-C技術組裝出石鯛科染色體水平的條石鯛魚基因組,并利用二代轉錄組測序,完成基因組的結構注釋。文章發表在《GIGASCIENCE》,影響因子7.267。

研究背景
研究背景
條石鯛(Oplegnathus fasciatus),中脊目中脊魚科的一員,是原產于東亞的一種具有重要商業價值的礁巖魚。在中國以及日韓等國家,條石鯛已成為海洋網箱養殖和海洋牧場魚類儲備的重要漁業資源。近年來,新性染色發育過程中的性二態性現象和廣泛存在的生物病害引起了人們越來越多的關注,然而,目前還缺乏完善的基因組資源來深入了解性別決定機制和建立生殖抗性育種體系。
研究方法

研究方法
研究結果
研究結果
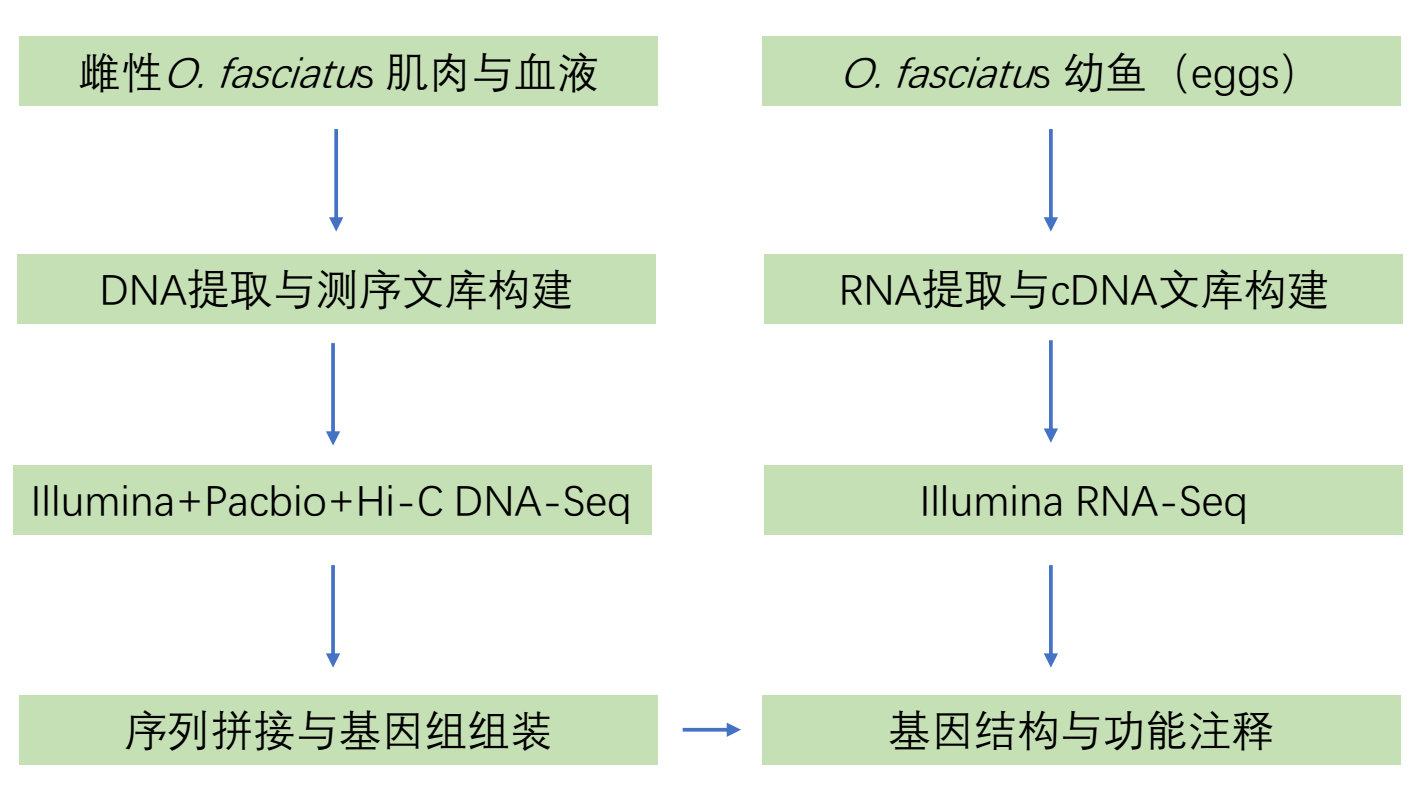
? Part 1 基因組測序
利用Illumina二代測序平臺進行DNA測序以評估基因組的大小,獲得80.8Gb的clean data。最終拼接得到744.5Mb大小的基因組,GC含量為41%。獲得的基因組質量較低,因為得到的基因組有大量的重復內容。
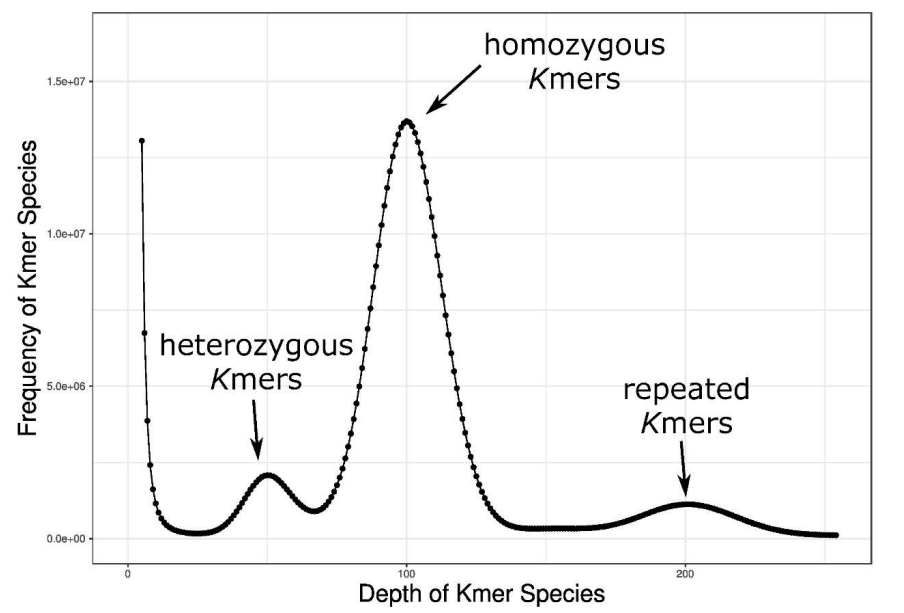
圖1:O.fasciatus基因組的K-mer分布
基于Pacbio平臺的三代DNA測序共得到62.9Gb的raw reads,并組裝得到875.9Mb的條石鯛魚基因組,比二代測序評估的基因組稍大。考慮到基因組序列的復雜性,在利用Redundans v0.13c去除冗余序列之后,得到778Mb大小的基因組。再經過二代測序結果的校正完善之后,最終獲得778.7Mb大小的石鯛魚基因組草圖。
繼二代與三代DNA測序之后,實施Hi-C技術,并將Hi-C數據比對到石鯛魚基因組草圖上,組裝獲得24條染色體序列,最終的確定了768.8Mb的基因組,占基因組草圖大小的98.7%。
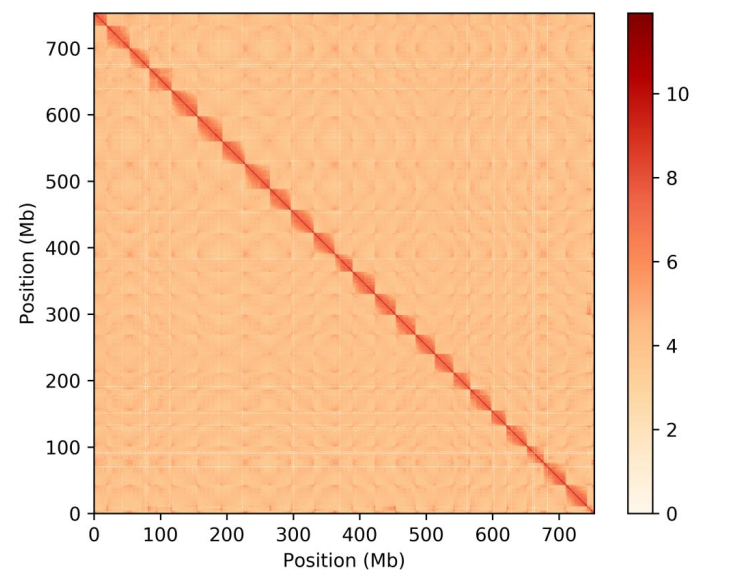
圖2:O.fasciatus 基因組的Hi-C染色體間相互作用
? Part 2 轉錄組測序
利用Illumina二代測序平臺進行轉錄組測序,以獲得基因組的注釋信息。RNA-Seq共獲得42.2Gb的高質量序列。基于同源性以及轉錄組測序結果,利用de novo 對基因組進行注釋。使用TopHat1.2軟件將二代轉錄組數據比對到基因組,并利用Cufflinks預測基因結構,進行基因的結構注釋,最終的總基因集由24003個基因組成,平均每個基因有10.1個外顯子。
基因組的功能注釋通過利用BLASTX以及BLASTN與Swiss-prot、NR、GO與KEGG等數據庫比對獲得,24003個基因中有97.3% (23364個基因)被至少一個數據庫注釋。另外,使用tRNAscan-SE (tRNAscan-SE, RRID:SCR_010835)和Rfam數據庫對四種非編碼RNA (microRNAs、轉運RNA、核糖體RNA和小核RNA)進行了注釋。
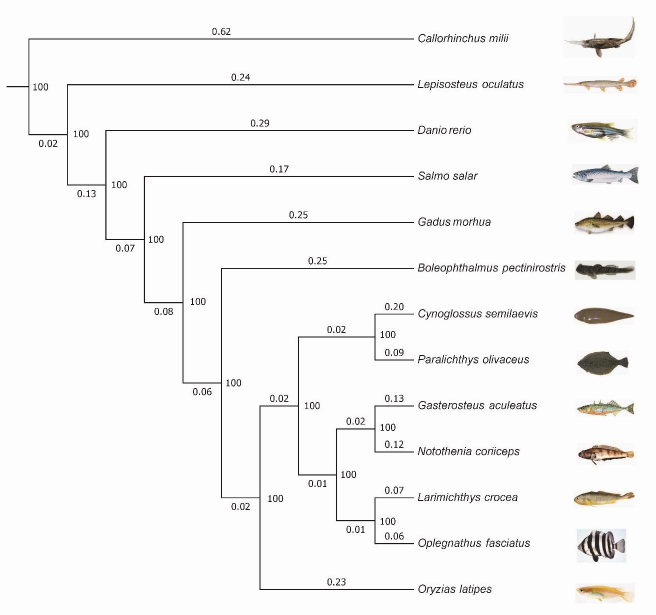
圖3:O.fasciatus 與其他魚類的系統發育關系
研究結論
研究結論
本研究成功組裝了染色體水平的O.fasciatus 基因組,這是基于三代測序平臺的條石碉魚的基因組的測序、組裝與注釋的研究。本研究中獲得的染色體水平的基因組以及為雌魚生成的基因注釋數據,將為進一步研究性別決定機制,特別是獲得雄魚Y染色體的組裝提供有價值的資源。
參考文獻
參考文獻
Xiao Y, Xiao Z, Ma D, Liu J, Li J. Genome sequence of the barred knifejaw Oplegnathus fasciatus (Temminck& Schlegel, 1844): the first chromosome-level draft genome in the family Oplegnathidae.Gigascience. 2019;8(3):giz013. doi:10.1093/gigascience/giz013


