2017-03-31

派森諾生物與華東理工大學合作,應用宏基因組學的方法,挖掘棉花降解中新的糖苷水解酶代謝路徑,其研究成果于2016年發表于《Journal of Biotechnology》上(影響因子:2.667)。
1 研究背景
糖苷水解酶是水解酶家族中的一大類,隸屬于糖基酶,是所有可水解糖苷鍵的酶類總稱。近年來,隨著糖類物質在生物學領域內的重要性越發凸顯,關于糖苷酶的研究與應用也變得越發重要。在工業生產過程中,需要更高性能的糖苷水解酶,以便使生物質原料有效的降解為可發酵糖類。本文通過宏基因組學方法結合基因克隆、蛋白表達等手段,挖掘土壤微生物群落中,可以提高生物質轉化和生物制品生產的新的生物質降解酶。
2 研究方法
樣本來源:棉花種植地土壤樣本
測序平臺:Illumina MiSeq+HiSeq
首先通過MiSeq平臺分別測序細菌16S V4區以及真菌ITS1區,獲取樣本中相應的菌群組成多樣性及豐度等信息;然后利用HiSeq平臺對樣本進行宏基因組鳥槍法測序,并根據測序結果在CAZy數據庫中比對獲得相應糖苷水解酶信息;最后通過基因克隆,蛋白表達以及酶活性測定等方式挖掘新的糖苷水解酶。
3 研究結果
3.1 土壤微生物群落組成分析
MiSeq平臺共產出57,883 條序列,根據97%相似度閾值歸并劃分出214個OTU,并發現在門水平優勢菌群為Proteobacteria(72%)、Bacteroidetes(17%)、Firmicutes(10%),其中Proteobacteria中以Gamma-Proteobacteria(41%)與Beta-Proteobacteria(26.1%)為主;Bacteroidetes中以Cytophagales(14.3%)為主;Firmicutes中以Bacillus(9.99%)為主。
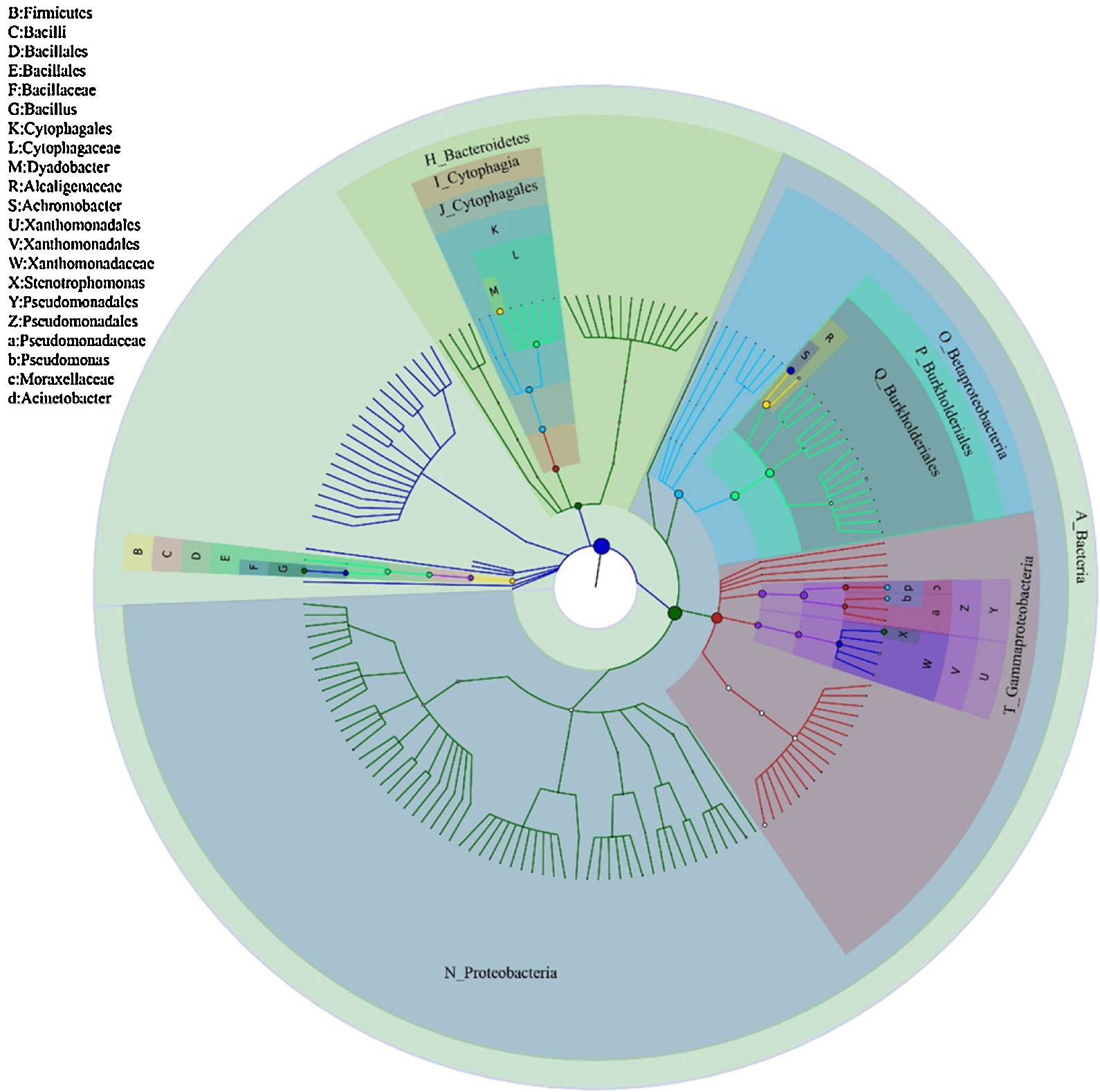
利用MetaPhlAn構建菌群結構分類等級樹,以不同顏色區分各分類單元,并通過節點大小反映它們的豐度分布情況,從復雜的群落數據中快速發現優勢的微生物類群。
3.2 宏基因組數據分析
HiSeq平臺共產出3.3Gb宏基因組數據,拼接組裝后的Contigs數據提交到MG-RAST進行分析;共406,305條序列通過質量控制,并預測得到321,789個蛋白編碼區進行后續分析;將這些蛋白通過BLASTX在CAZy數據庫中進行比對,確定了32個主要的糖苷水解酶類家族成員,包含2058個候選基因,其中豐度高的三個家族為GH2、GH3以及GH43,并最終從這些基因中挑選24個作為克隆及蛋白表達分析。
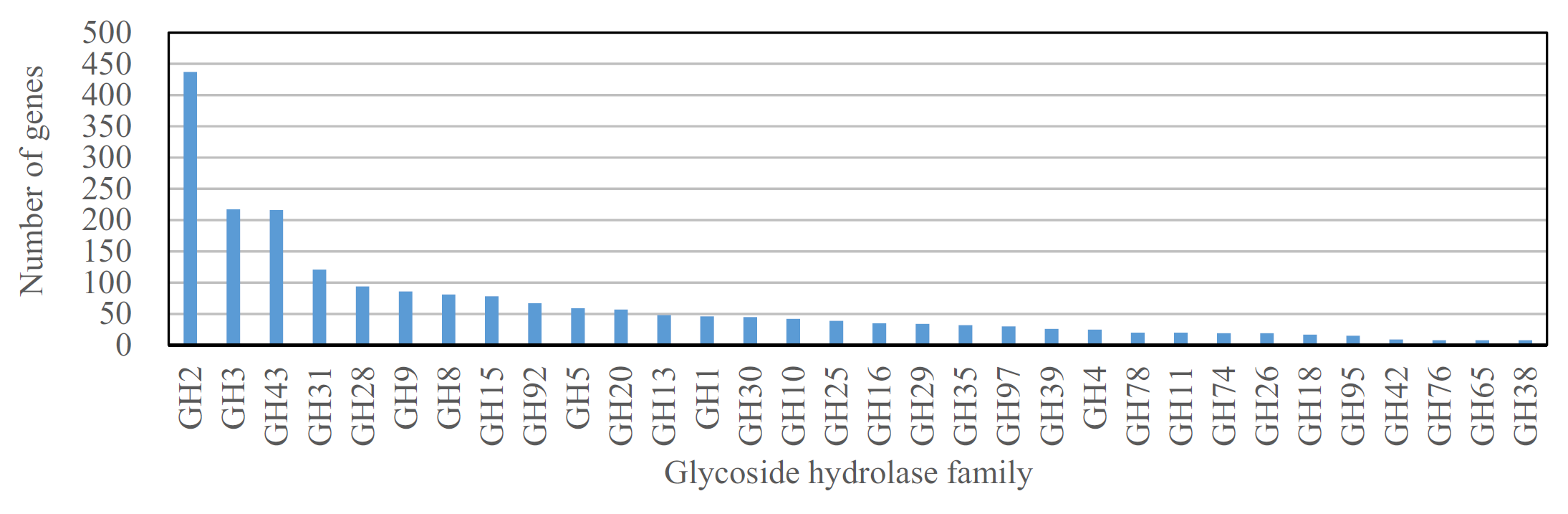
32個主要的糖苷水解酶家族,共包含2058個候選基因
3.3 糖苷水解酶基因的克隆表達及特性研究
被選定的糖苷水解酶基因通過PCR擴增及克隆到表達載體的方式引入到大腸桿菌等宿主中進行蛋白表達,并對細胞裂解液進行SDS-PAGE電泳,后續進行酶活性檢測;最終挑選出16個在生物質轉化及生物制品生產中存在潛在應用價值的糖苷水解酶基因克隆。
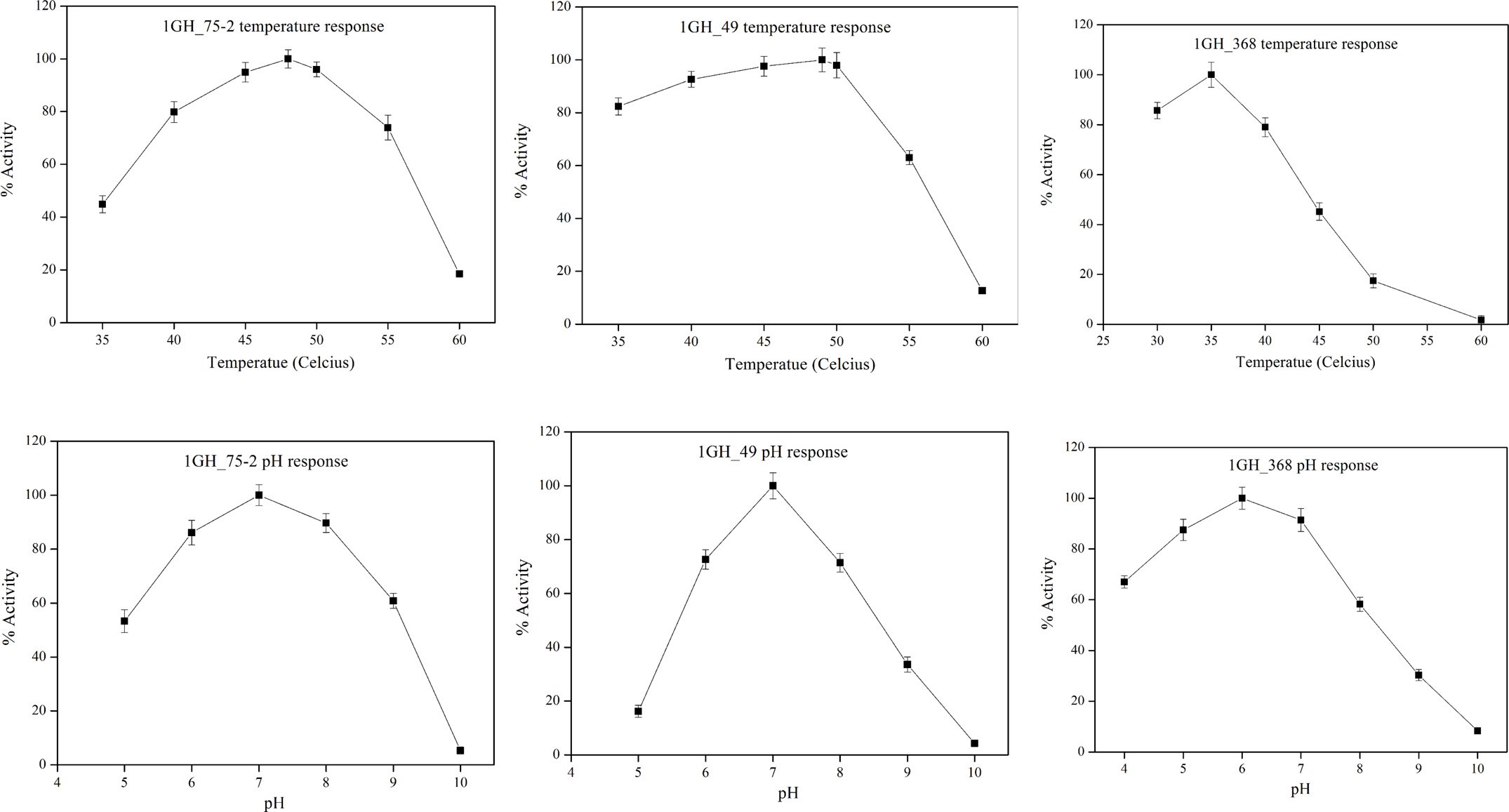
對于1GH_75-2,1GH_49以及1GH_368酶的溫度和pH調查
4 研究結論
本研究描述了從棉花種植地土壤中鑒定的微生物群落參與粗棉生物降解的功能特性,通過宏基因組學的研究方法結合基因克隆、蛋白表達及酶活性測定等手段,成功篩選出16個在生物質轉化及生物制品生產存在潛在應用價值的糖苷水解酶類基因,為研究生物質降解酶并結合生產實際提供了理論基礎與研究思路。
參考文獻
Zhang G, Liu P, Zhang L, et al. Bioprospecting metagenomics of a microbial community on cotton degradation: Mining for new glycoside hydrolases[J]. Journal of Biotechnology, 2016, 234:35-42.


