干貨!IGV快速入門手冊
2021-05-25
IGV(Integrative Genomics Viewer)是一款本地即可使用的可視化工具,安裝簡單,操作便捷,支持?jǐn)?shù)據(jù)多元且非常實用!只需導(dǎo)入?yún)⒖蓟蚪M文件以及bam文件(IGV支持多種文件類型,這里我們主要以轉(zhuǎn)錄組結(jié)果提供給老師的bam文件為例),即可對轉(zhuǎn)錄組的比對結(jié)果進(jìn)行可視化瀏覽!
什么?你還不會操作?別怕,小派已經(jīng)準(zhǔn)備好了IGV保姆級操作指南,帶你輕松玩轉(zhuǎn)IGV!
除了備好IGV軟件(注意要在安裝java的前提下),還要對應(yīng)的參考基因組文件,也就是本地的基因組fa文件,注釋文件gtf。這里每個文件都需要有對應(yīng)的index,fa文件需要有對應(yīng)的index文件fai,bam則需要對應(yīng)的bai文件:
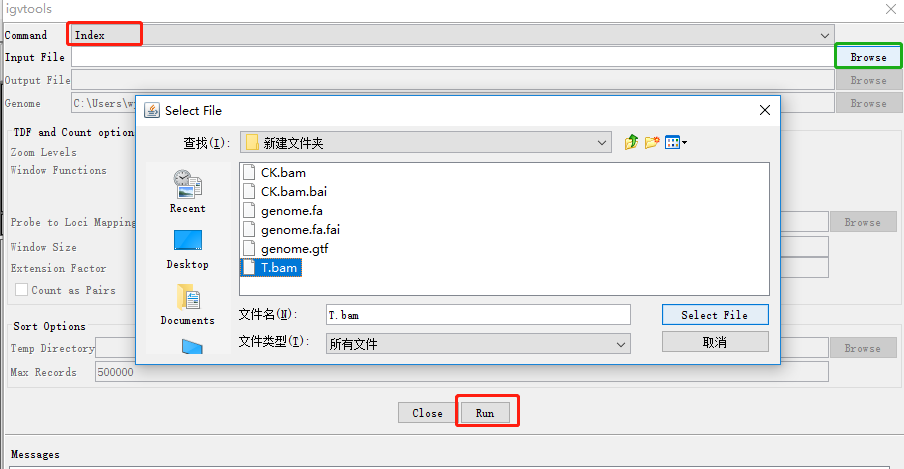
缺少index文件也可使用IGV來生成,點擊Tools→Run igvtools:
Command選擇Index,Input File選擇需要生成index的文件,點擊Run,即可生成需要的index啦!
點擊Genomes→Load Genomes From Files導(dǎo)入基因組文件:
點擊File→Load from File, 導(dǎo)入gtf和bam文件,即可進(jìn)入如下界面:
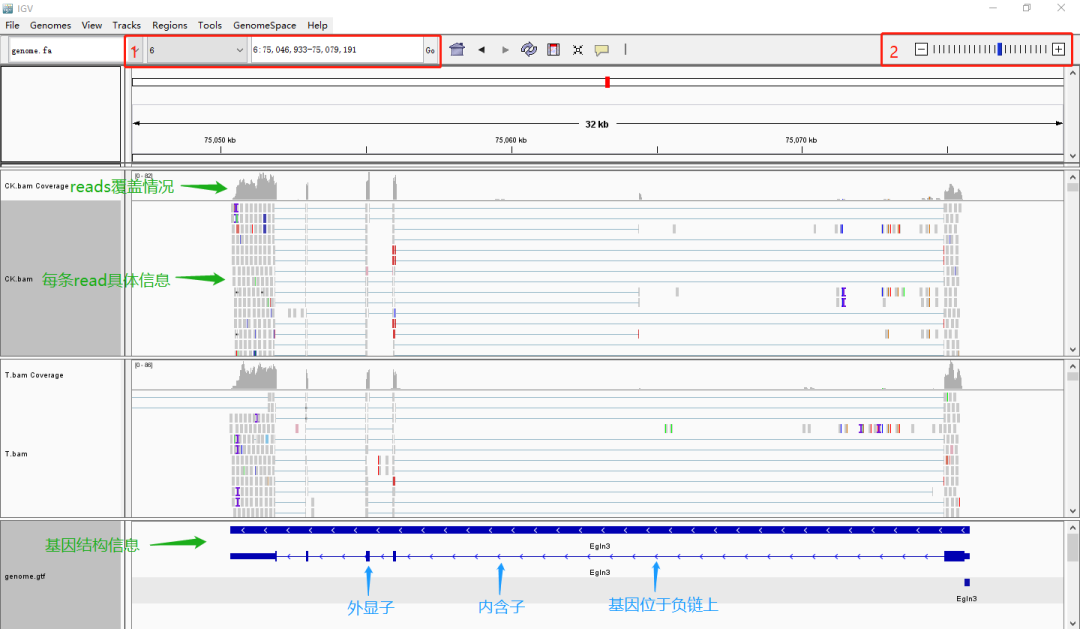
可以在“1”的位置輸入需要查看的基因所在的染色體及位置(在轉(zhuǎn)錄組結(jié)果中的Annotation表均有提供對應(yīng)信息)即可快速查看到該基因的具體情況,包括基因結(jié)構(gòu)、轉(zhuǎn)錄組每個樣本中該基因的reads覆蓋情況。可以通過“2”的位置可以放大或縮小視野范圍,放大到一定程度可以查看到序列信息,同時也可以查看發(fā)生的插入或突變的具體情況。每個堿基擁有特定的顏色,可以看出下圖中突變的位置在參考基因組上該位點堿基為A,所測到的該條read在該位置堿基突變?yōu)镃:
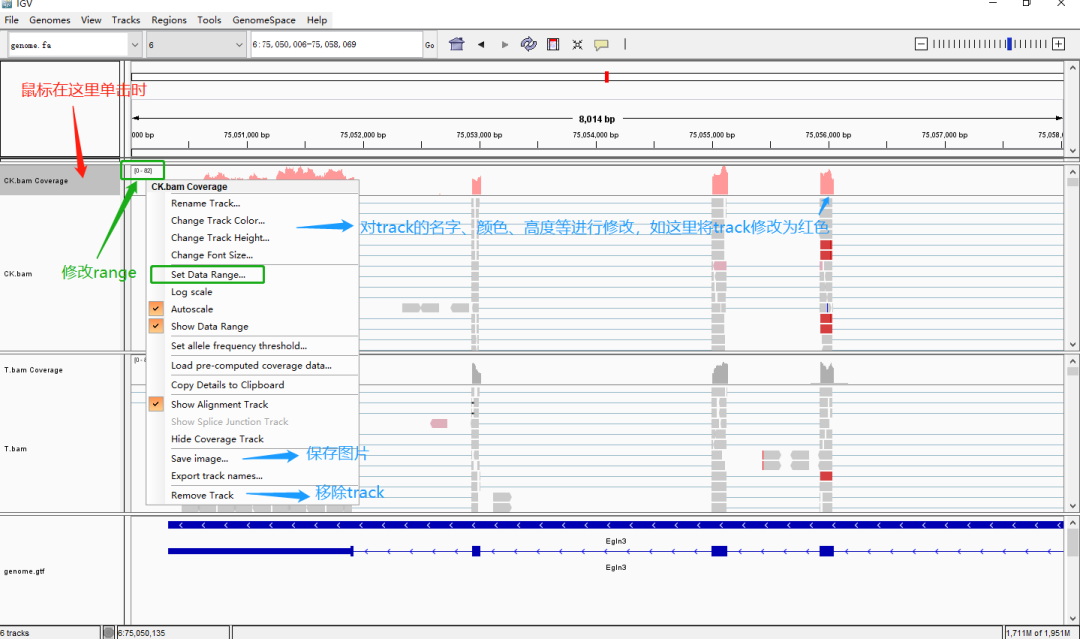
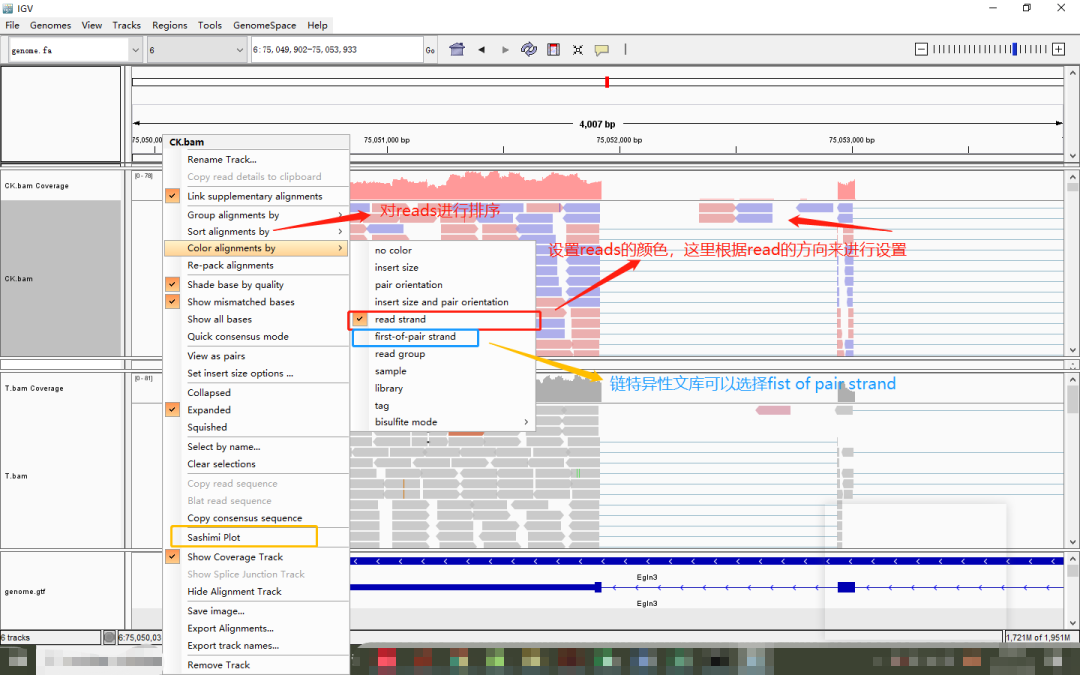
在IGV中,在不同位置單擊鼠標(biāo)右鍵會出現(xiàn)不同的菜單欄,可以進(jìn)行多種操作,這里我們只列舉一些常見設(shè)置:
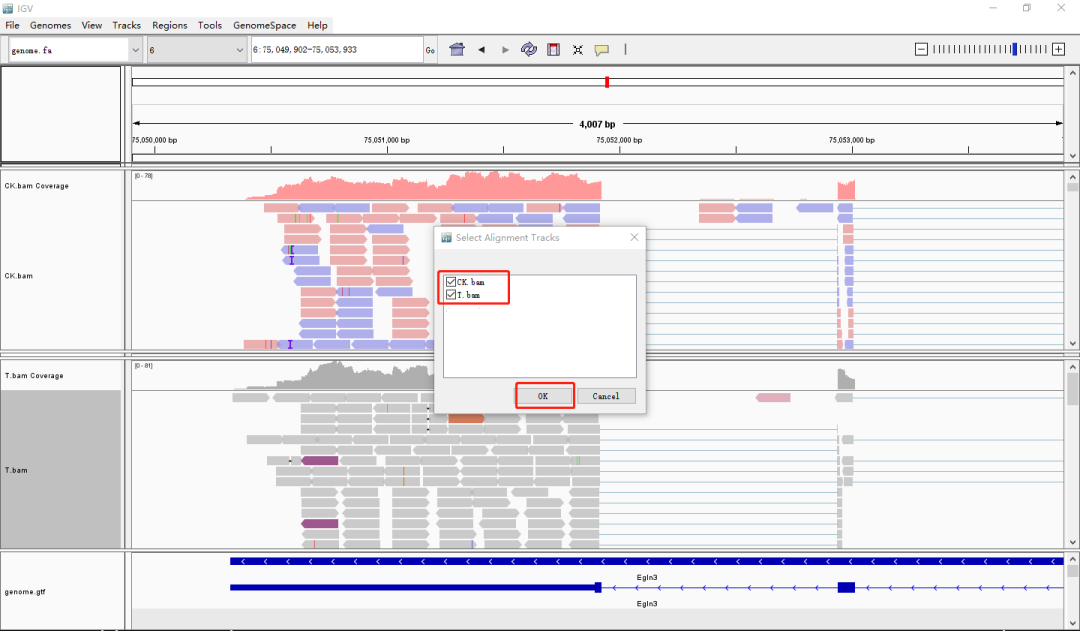
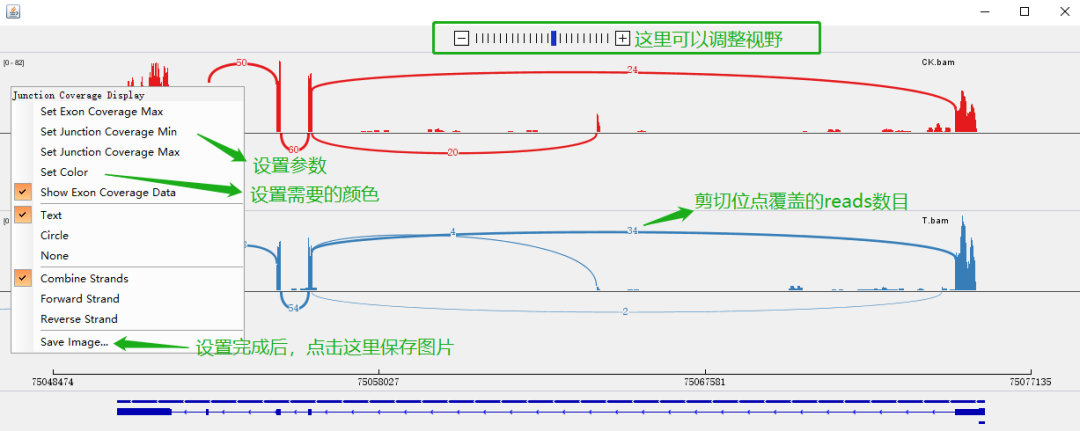
細(xì)心的同學(xué)已經(jīng)發(fā)現(xiàn)了,大名鼎鼎的sashimi也出現(xiàn)在了菜單欄中。沒錯!只要單擊sashimi plot,選擇需要進(jìn)行分析的樣本,就可以一鍵獲得文章中常用于展示可變剪切的刺身圖啦:
看過這篇使用指南,是不是發(fā)現(xiàn)IGV操作也不過如此!其實小派這里只是展示了轉(zhuǎn)錄組數(shù)據(jù)可視化的一些常規(guī)操作,IGV還擁有非常豐富的功能和用途。
更多詳細(xì)咨詢請關(guān)注派森諾官網(wǎng)