
2021-05-20
今天結合nature medicine中的一篇文章,和大家分享下熱圖的繪制,主要亮點功能是:
(1)名稱太多看不清,如何只展示特定的名稱?
(2)數據太密集,如何快速調整單元格的寬和高?
論文頁面:

文章鏈接:https://www.nature.com/articles/s41591-020-0944-y
代碼及數據:https://github.com/ajwilk/2020_Wilk_COVID
擬復現圖片樣式:Fig2中的熱圖樣式
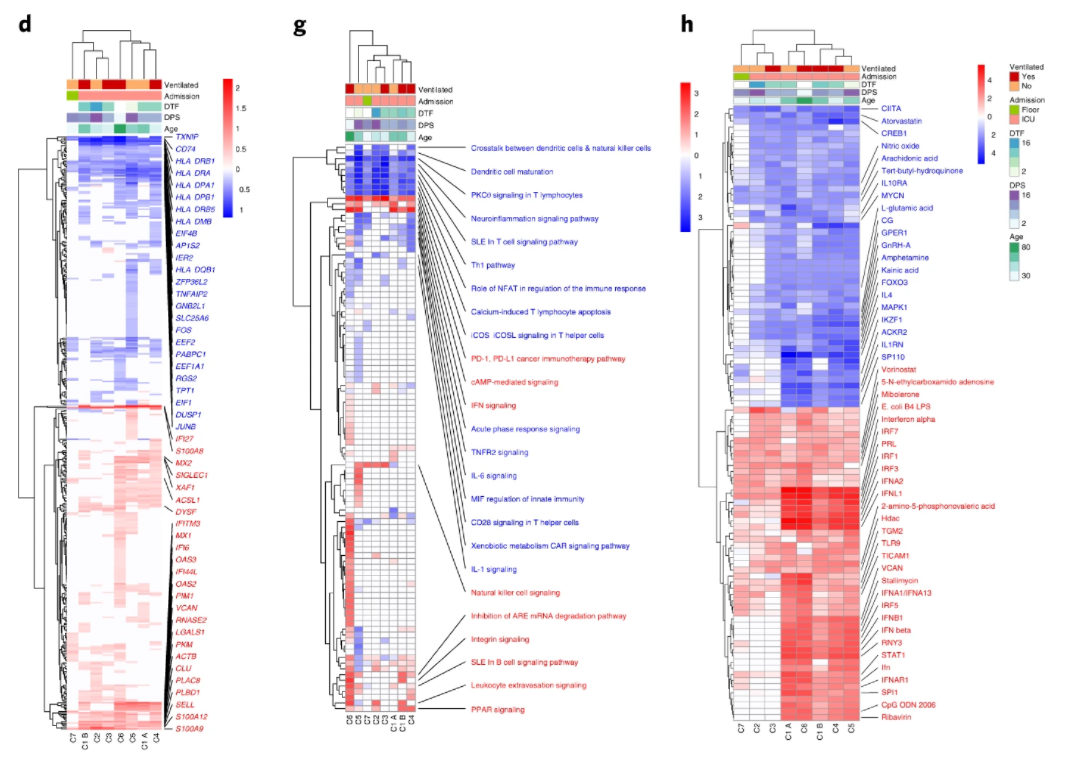
圖1 擬復現圖片樣式
使用數據:數據大家可以通過上述鏈接下載,附件是一個rds文件(1.5G,一般電腦慎加載會卡死的), 我們已經下載處理好了一個示例數據(如圖2所示)。大家可以通過基因云(https://www.genescloud.cn)的云端文件進行選擇使用, 具體可參考下圖7 云端數據選擇
name | C1A | C1B | C2 | C3 | C4 | C5 | C6 | C7 |
ACKR2 | -3.606 | -2.4 | 0 | 0 | -3.273 | -3.701 | -3.701 | 0 |
amphetamine | -2.491 | -2.491 | -1.944 | -2.303 | -2.491 | -2.664 | -2.094 | 0 |
anisomycin | -2.218 | -2.218 | -1.243 | -2.218 | -2.218 | -2.433 | -1.074 | -0.506 |
APEX1 | -2.236 | -2.236 | 0 | -2 | -2.236 | -2.236 | -2 | 0 |
arachidonic acid | -2.403 | -2.063 | -1.679 | -1.806 | -2.19 | -2.19 | -0.993 | -1.894 |
atorvastatin | -2.967 | -3.13 | -2.236 | -1.569 | -3.13 | -2.828 | -1.906 | -2 |
bicuculline | -2.942 | -2.469 | -0.728 | -1.107 | -2.469 | -1.709 | -0.397 | 0 |
bucladesine | -1.792 | -1.611 | -0.733 | -1.392 | -2.718 | -2.385 | -1.239 | -0.179 |
圖2 示例數據
按照慣例,我們先畫一個基本的熱圖。
library(pheatmap) library(grid) mat <- read.delim("heatmap.txt",sep="\t",row.names=1) pheatmap(mat)
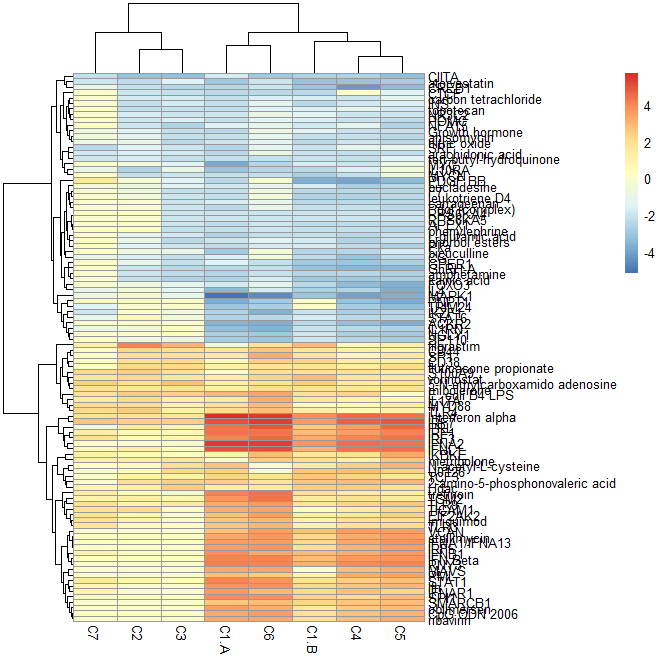
圖3 初始熱圖
上圖樣式不是很好看,存在以下幾點需要完善:①顏色不是很好看,且有灰色邊框線條;②行名有很多重疊無法識別;③ 熱圖缺少分組信息, 接下來我們通過代碼繼續完善。
# 設置顏色 color <- c("blue", "white", "red") myColor <- colorRampPalette(color)(100) # 添加分組信息 annotation_col <- data.frame(Group = factor(rep(c("T", "C"),4))) rownames(annotation_col) <- colnames(mat) # 繪制熱圖 p1 <- pheatmap(mat,color = myColor, border_color=NA, annotation_col = annotation_col)

圖4 美化后熱圖一
接下來通過調整單元格高度,使得文字錯開。
# 調整單元格高度,避免文字重疊 p1 <- pheatmap(mat,color = myColor, border_color=NA, annotation_col = annotation_col, cellheight=10)
圖5 美化后熱圖二
上圖通過調整單元格高度調整,文字是清晰可分辨了,但是圖片的整體高度會被拉長,放在文章里面不太方便查看。那么我們是否可以只展示特定的行名呢? 首先我們來看下文中提及的,可以實現只展示特定行名的函數:
# 展示特定行名函數 add.flag <- function(pheatmap, kept.labels, repel.degree) { heatmap <- pheatmap$gtable new.label <- heatmap$grobs[[which(heatmap$layout$name == "row_names")]] # keep only labels in kept.labels, replace the rest with "" new.label$label <- ifelse(new.label$label %in% kept.labels, new.label$label, "") # calculate evenly spaced out y-axis positions repelled.y <- function(d, d.select, k = repel.degree){ # d = vector of distances for labels # d.select = vector of T/F for which labels are significant # recursive function to get current label positions # (note the unit is "npc" for all components of each distance) strip.npc <- function(dd){ if(!"unit.arithmetic" %in% class(dd)) { return(as.numeric(dd)) } d1 <- strip.npc(dd$arg1) d2 <- strip.npc(dd$arg2) fn <- dd$fname return(lazyeval::lazy_eval(paste(d1, fn, d2))) } full.range <- sapply(seq_along(d), function(i) strip.npc(d[i])) selected.range <- sapply(seq_along(d[d.select]), function(i) strip.npc(d[d.select][i])) return(unit(seq(from = max(selected.range) + k*(max(full.range) - max(selected.range)), to = min(selected.range) - k*(min(selected.range) - min(full.range)), length.out = sum(d.select)), "npc")) } new.y.positions <- repelled.y(new.label$y, d.select = new.label$label != "") new.flag <- segmentsGrob(x0 = new.label$x, x1 = new.label$x + unit(0.15, "npc"), y0 = new.label$y[new.label$label != ""], y1 = new.y.positions) # shift position for selected labels new.label$x <- new.label$x + unit(0.2, "npc") new.label$y[new.label$label != ""] <- new.y.positions # add flag to heatmap heatmap <- gtable::gtable_add_grob(x = heatmap, grobs = new.flag, t = 4, l = 4 ) # replace label positions in heatmap heatmap$grobs[[which(heatmap$layout$name == "row_names")]] <- new.label # plot result grid.newpage() grid.draw(heatmap) # return a copy of the heatmap invisibly invisible(heatmap) }
函數寫好了,接下來我們看看具體效果。本示例隨機抽取20個行名,添加到原來的熱圖中。具提代碼如下,最終效果圖如圖6所示。
# 這里隨機抽取20個基因進行展示
gene_name<-sample(rownames(mat),20)
add.flag(p1,kept.labels = gene_name,repel.degree = 0.2)
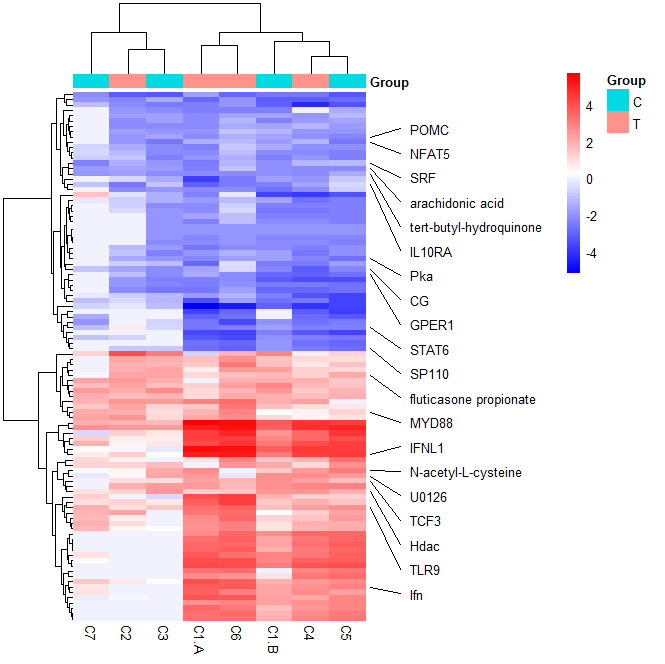
圖6 美化后熱圖三
到此我們就成功的通過代碼實現了一幅含有分組信息,只展示特定行名的熱圖,那么如何不通過代碼實現呢?接下來,給大家分享下基因云(https://www.genescloud.cn)的“交互熱圖”,幫助你“0”代碼快速制作漂亮的上述圖表,同時還提供多種樣式的在線調整。
為了方便大家學習實踐,基因云平臺已整合該文章數據,進入“交互熱圖”繪圖頁面,直接通過【文件上傳→云端文件→公共數據】按照路徑: Home>ref_data>COVID-19_data>交互熱圖,即可選擇使用。
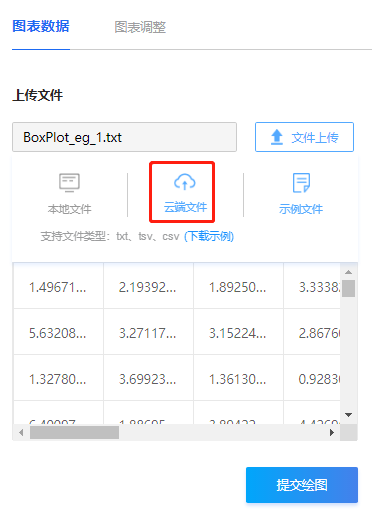
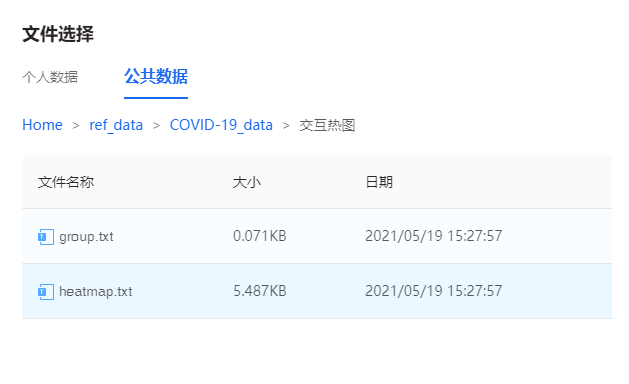
圖7 云端數據選擇
選擇好數據和分組文件后,一鍵提交繪圖。

圖8 快速提交頁面
(1)顯示特定基因名稱:在圖表調整里面,選擇【顯示名稱→行/行列】,下方會出現所有行名列表,可隨意勾選你想要展示的名稱。
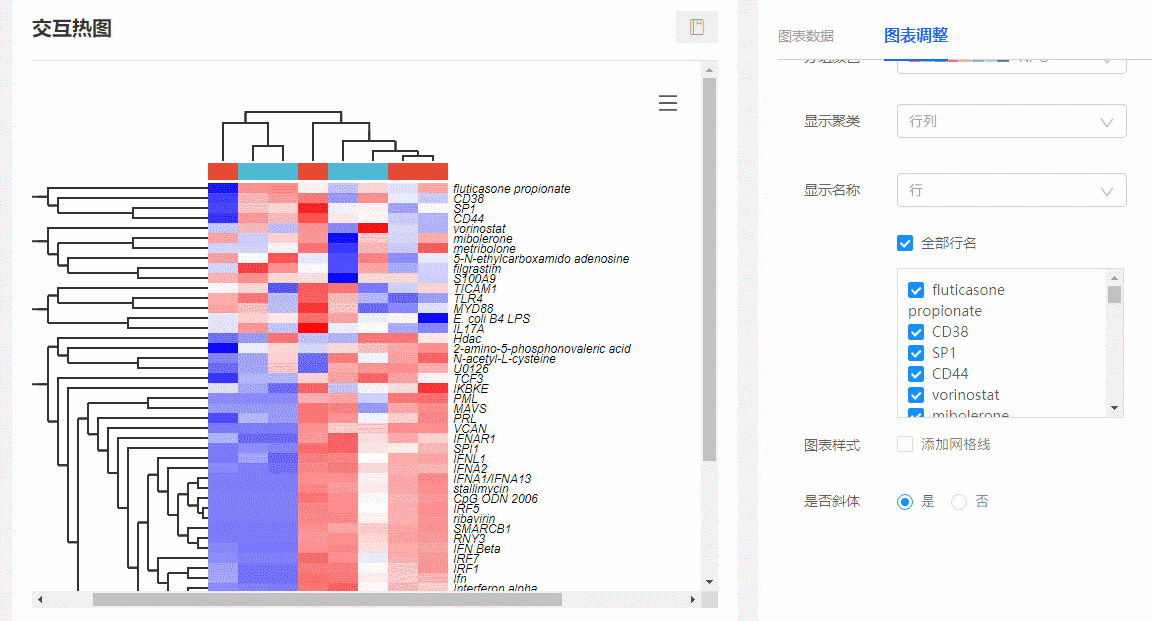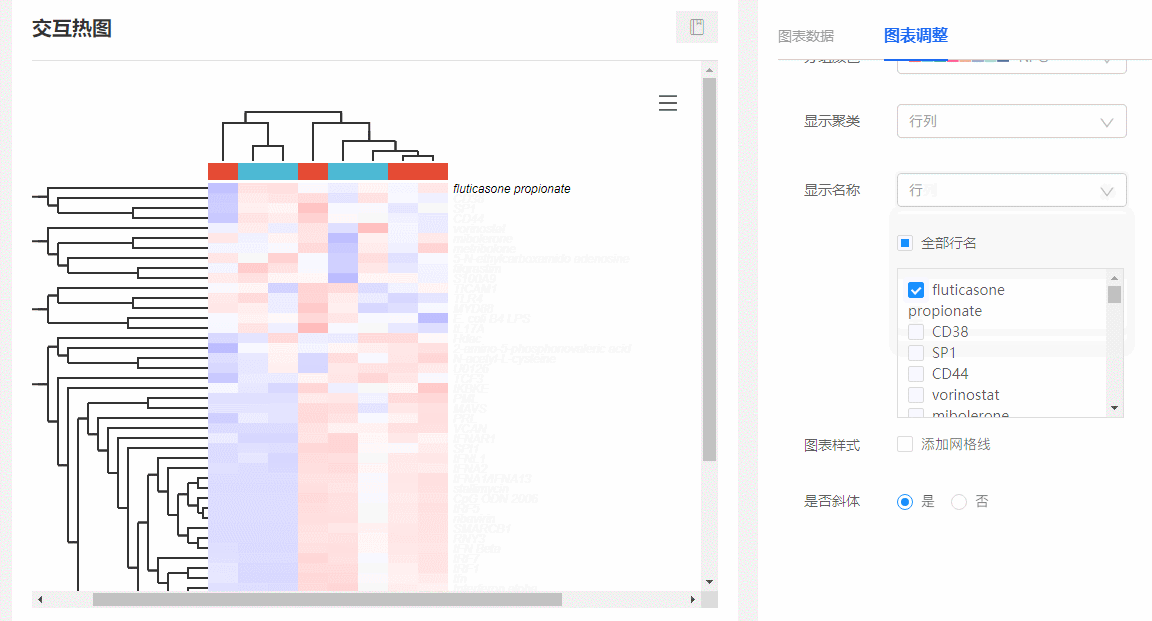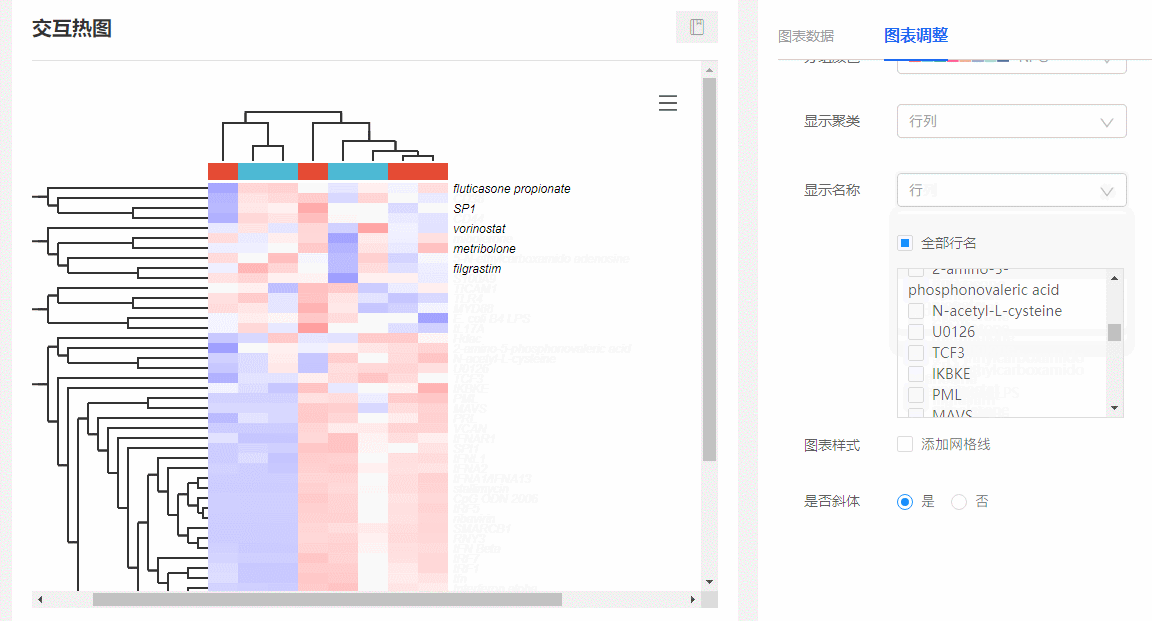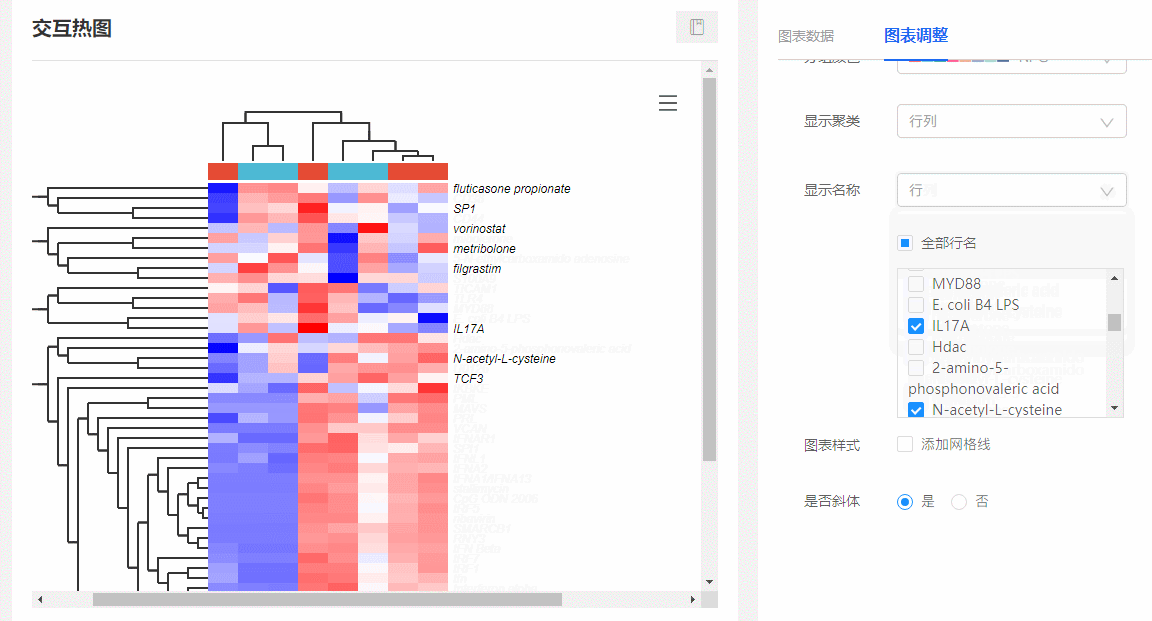
圖9 顯示特定基因名稱
(2)隨意伸縮單元格寬高:在圖表調整欄,隨意拖動【單元格寬度/高度】對應的滑動控制條,可隨意更改熱圖單元格的寬和高。
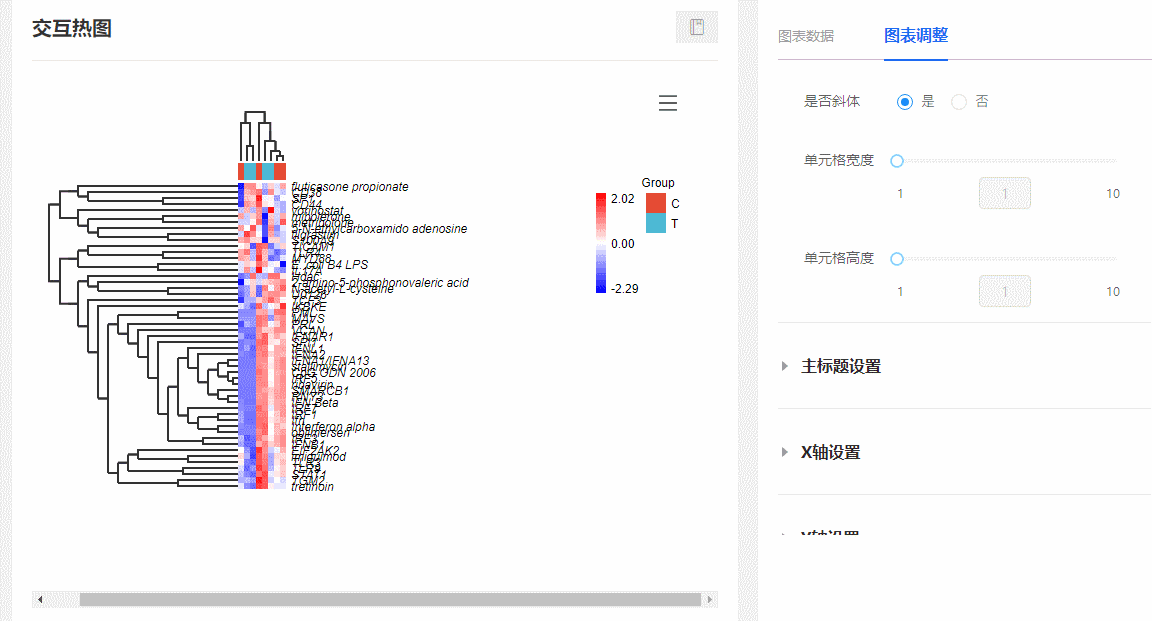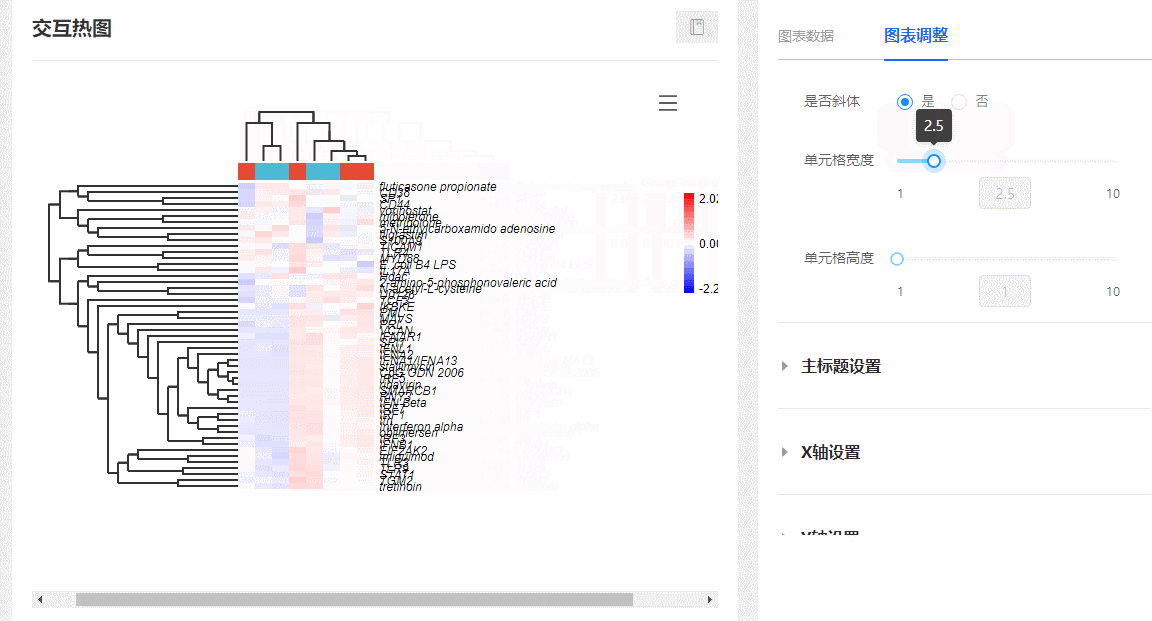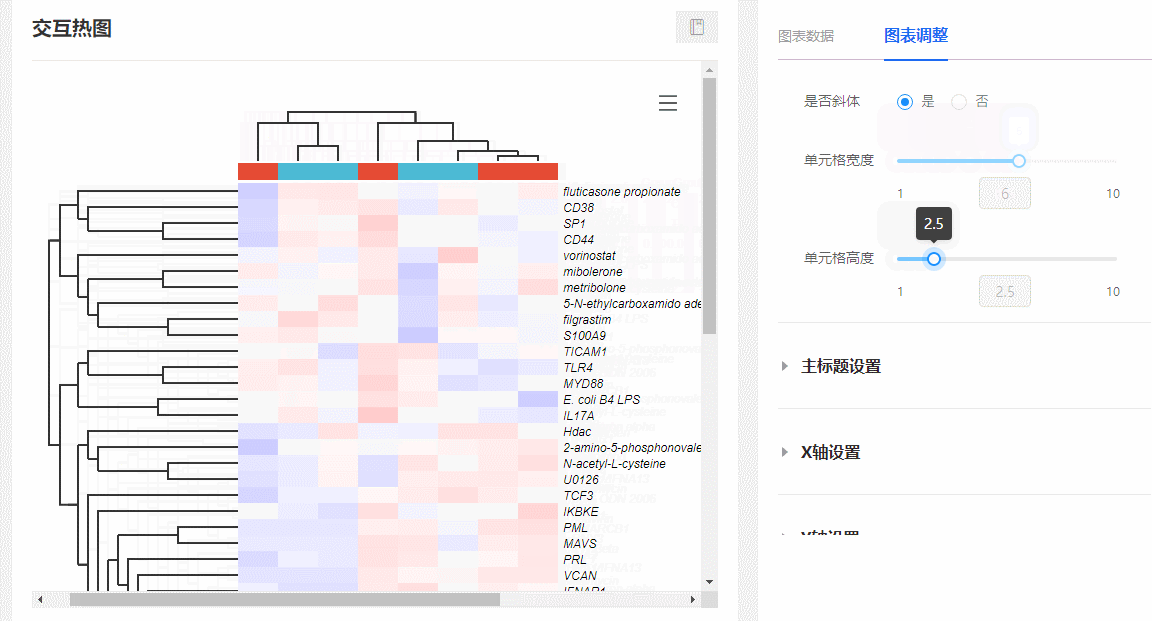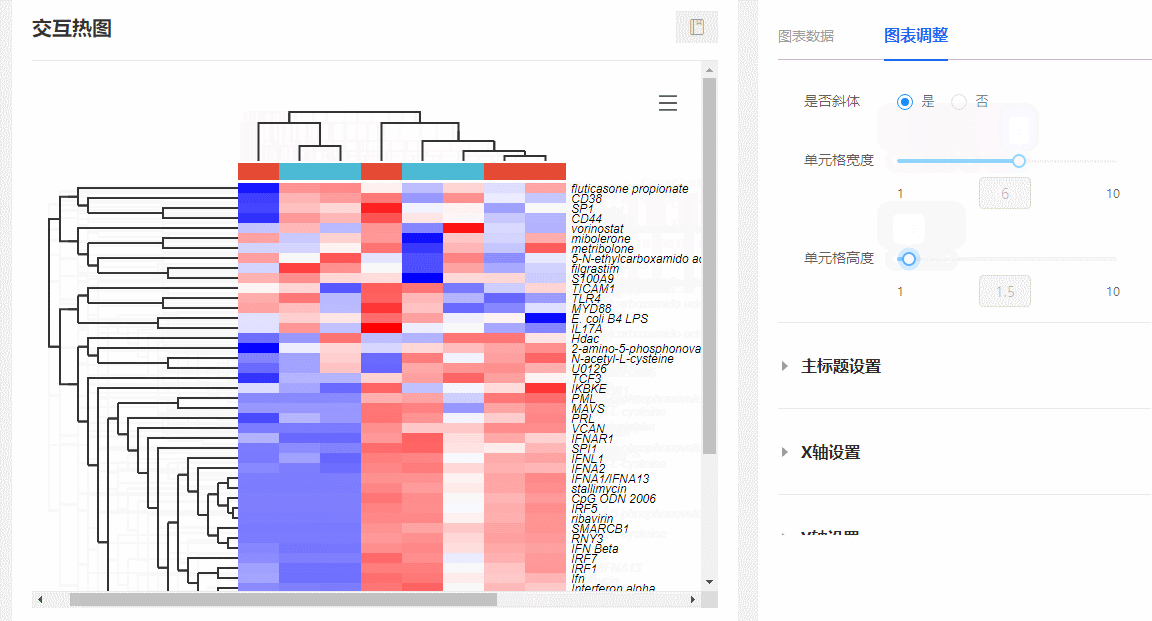
圖10 調整單元格長寬
趕緊來試一試吧,百度搜索“派森諾基因云”或者直接訪問https://www.genescloud.cn/home,進入“云圖匯”搜索“交互熱圖”嘗試體驗,并提寶貴建議至平臺消息中心-》反饋列表,或者發送到郵箱: [email protected]。"派森諾基因云" 一直持續上心上新,接下來會有更多好圖好工具陸續和大家見面,歡迎大家關注并進行體驗。