2021-01-29

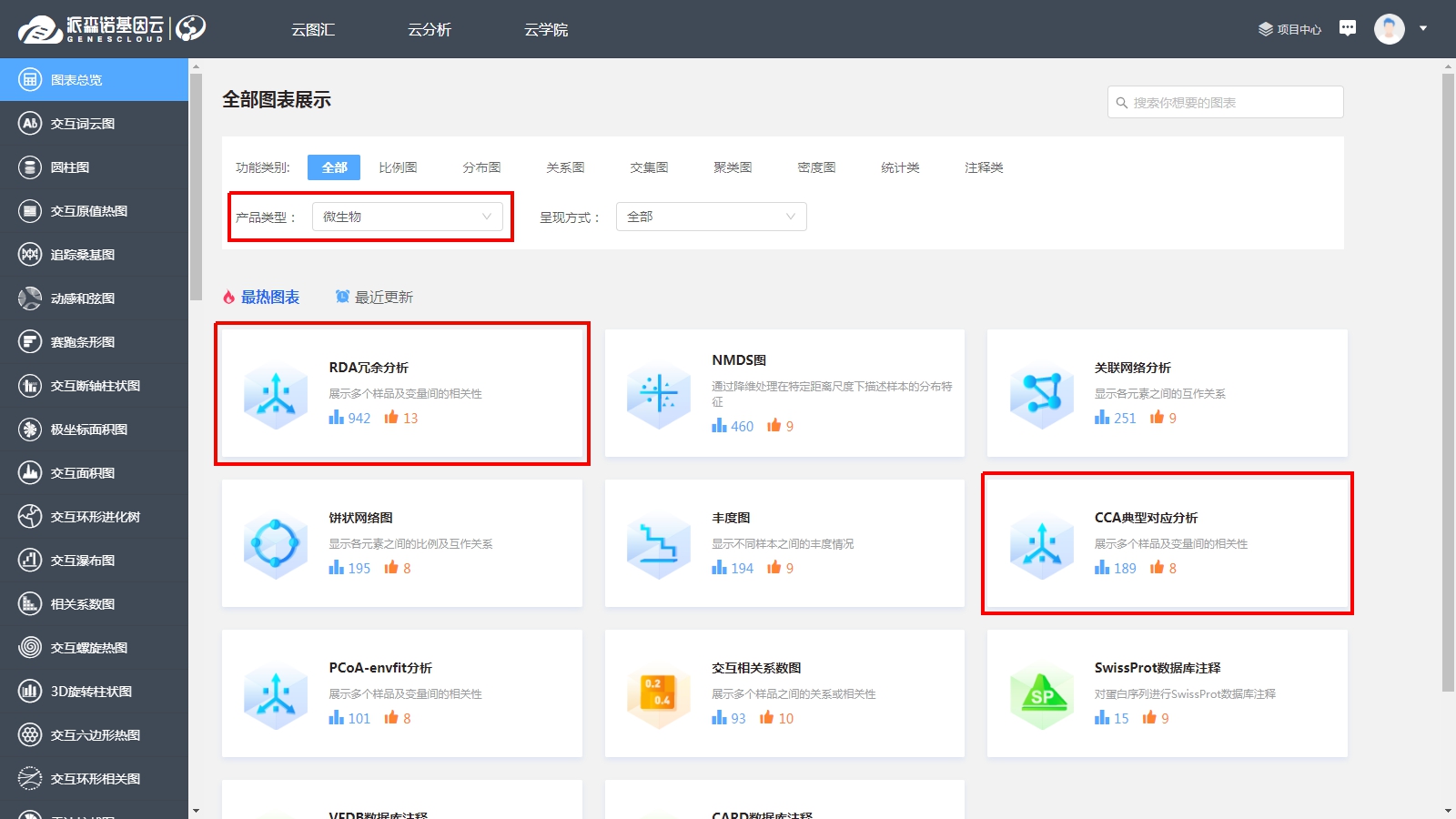
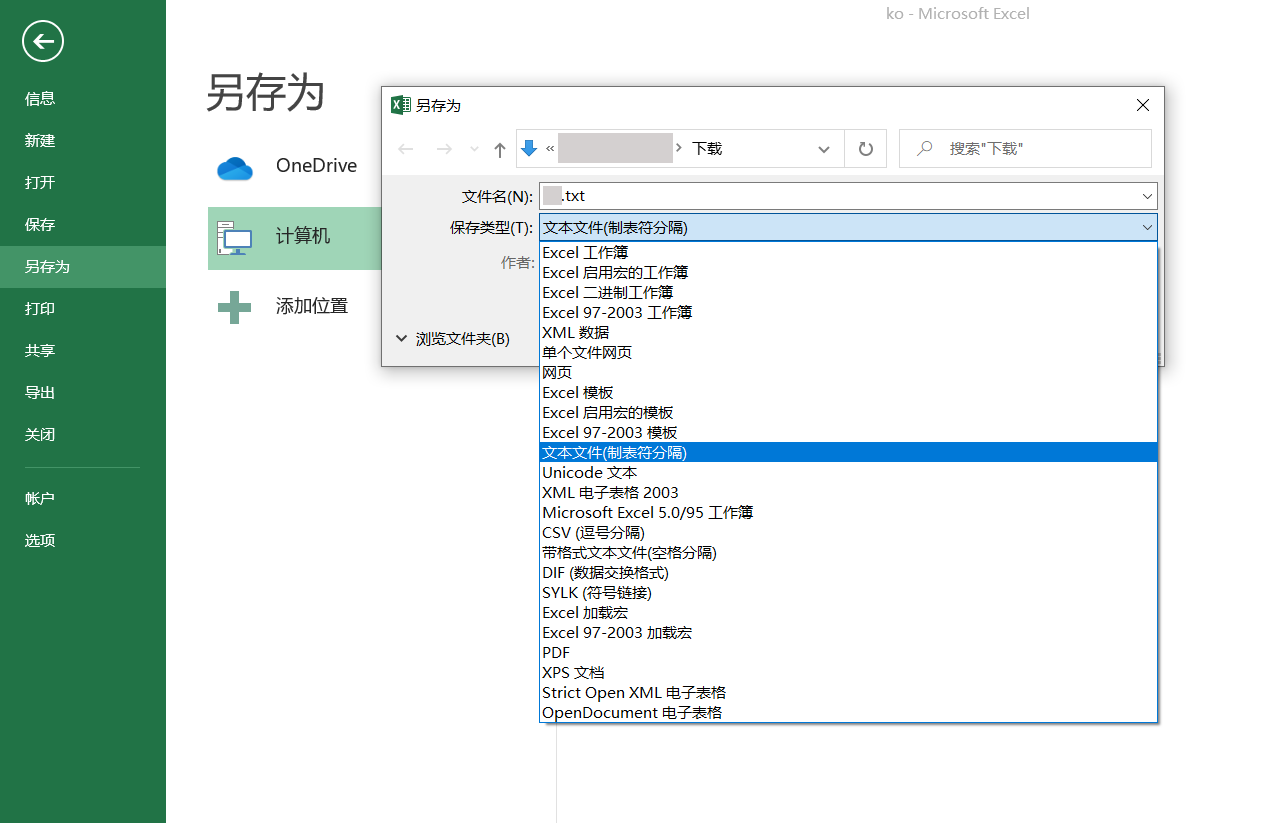
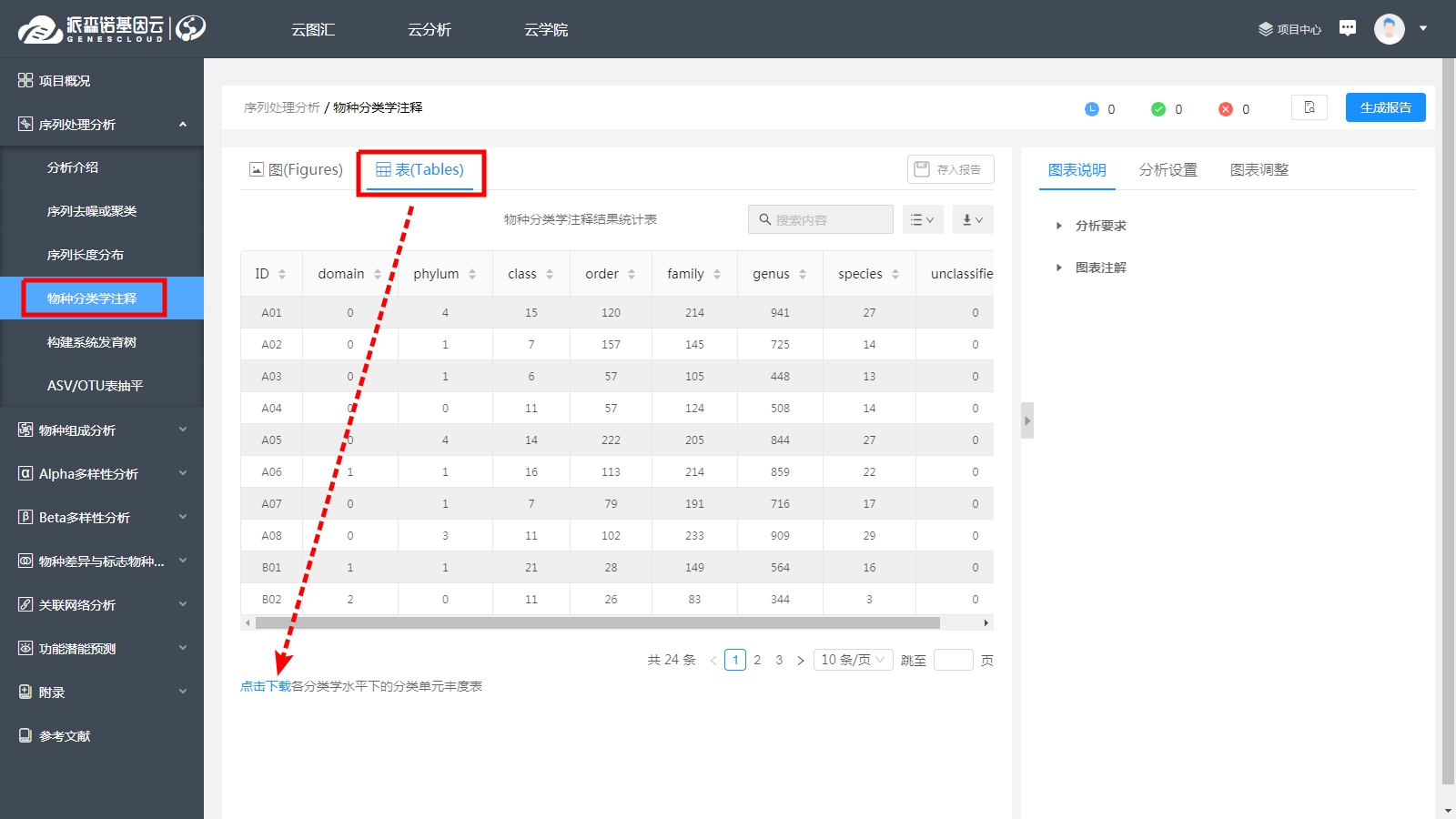
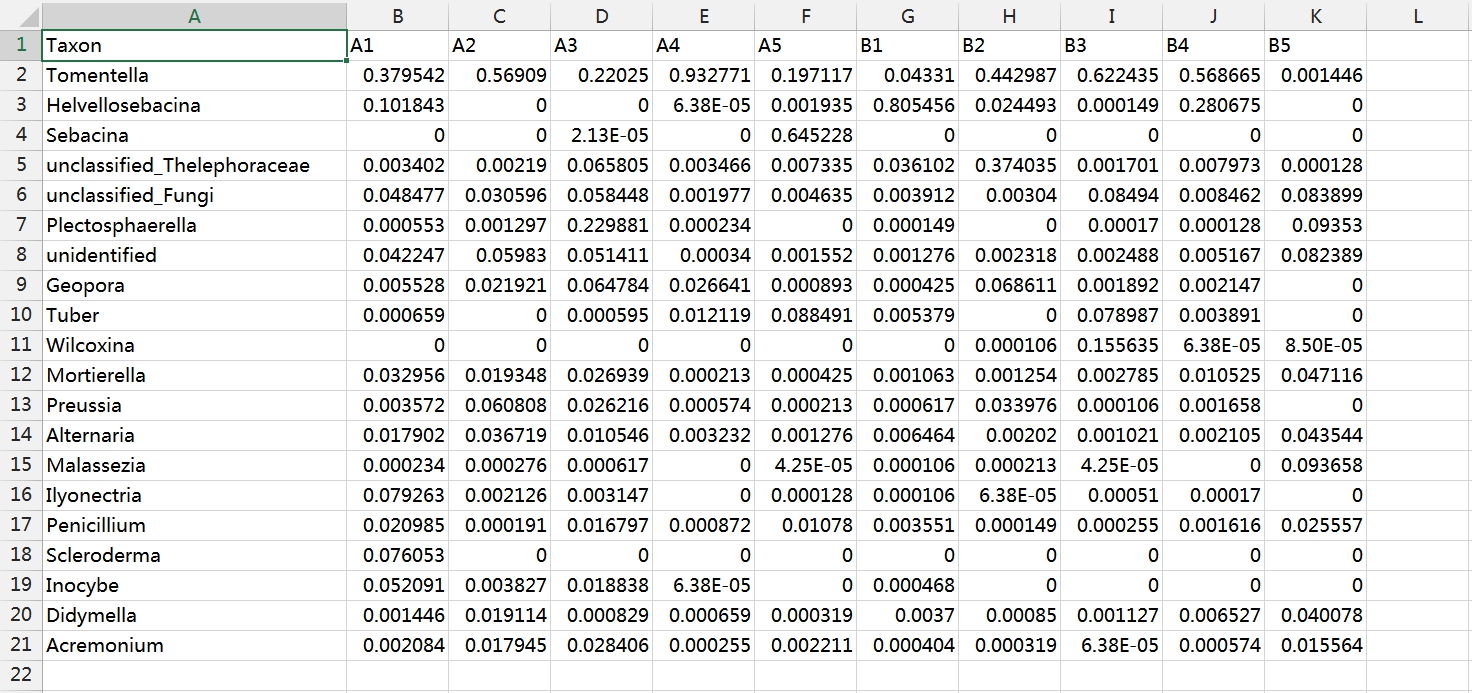
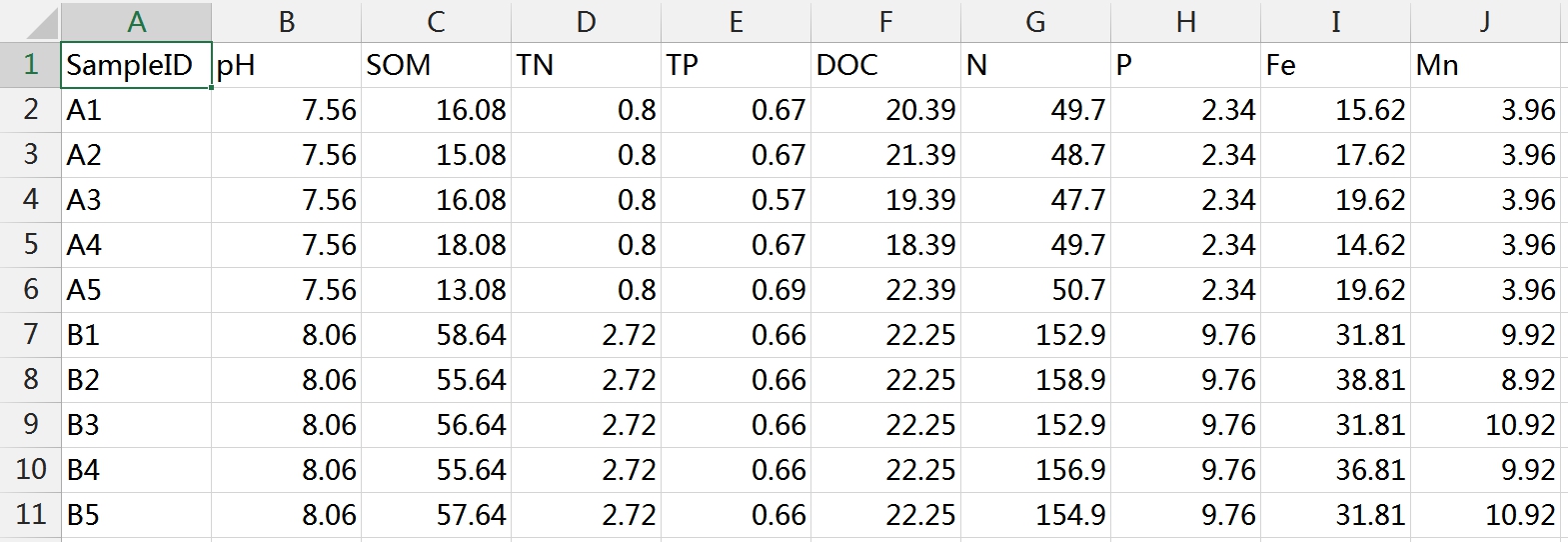
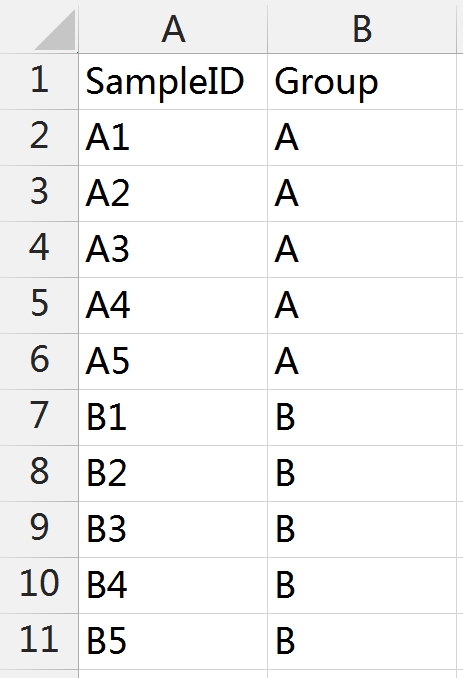
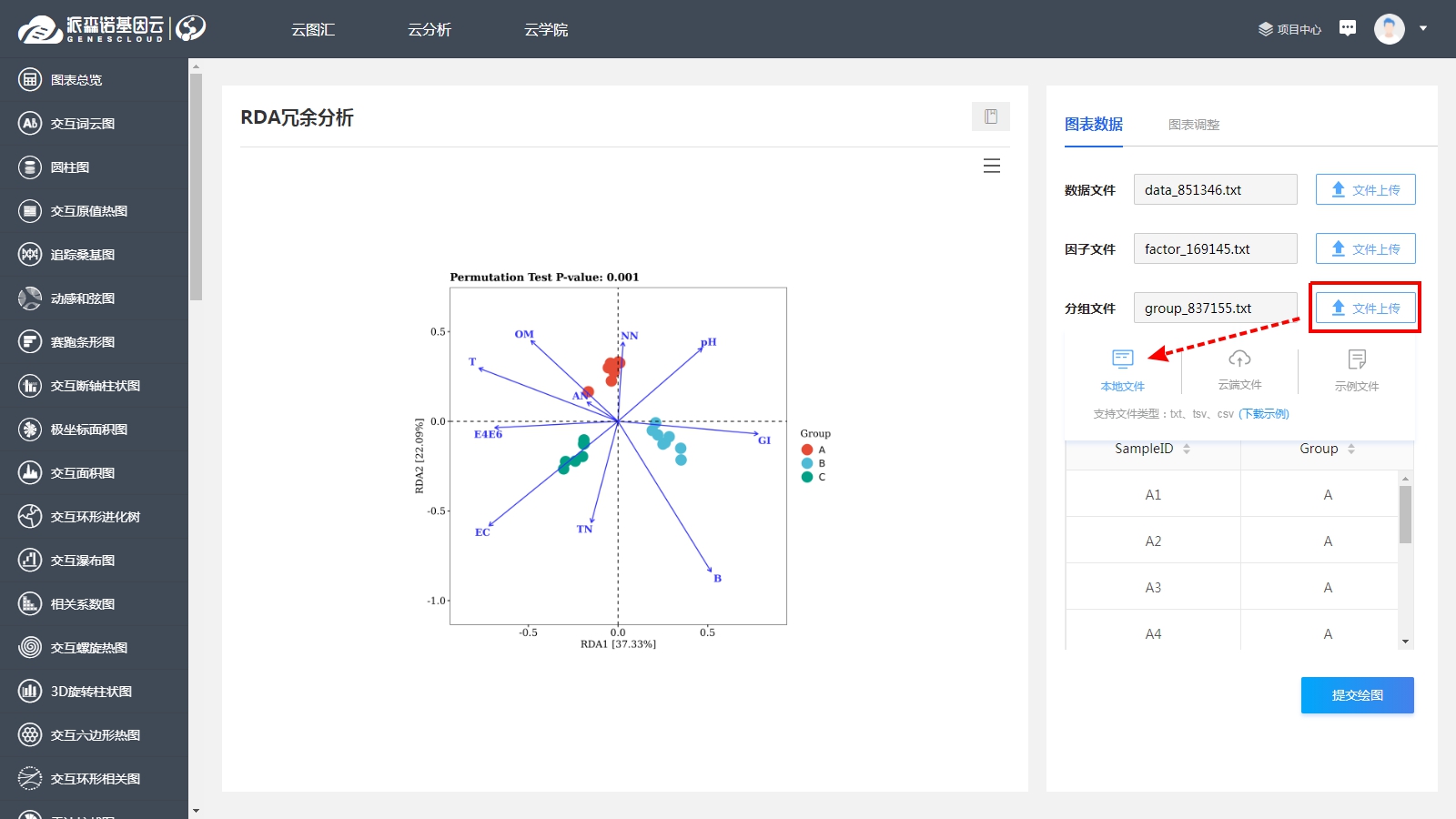
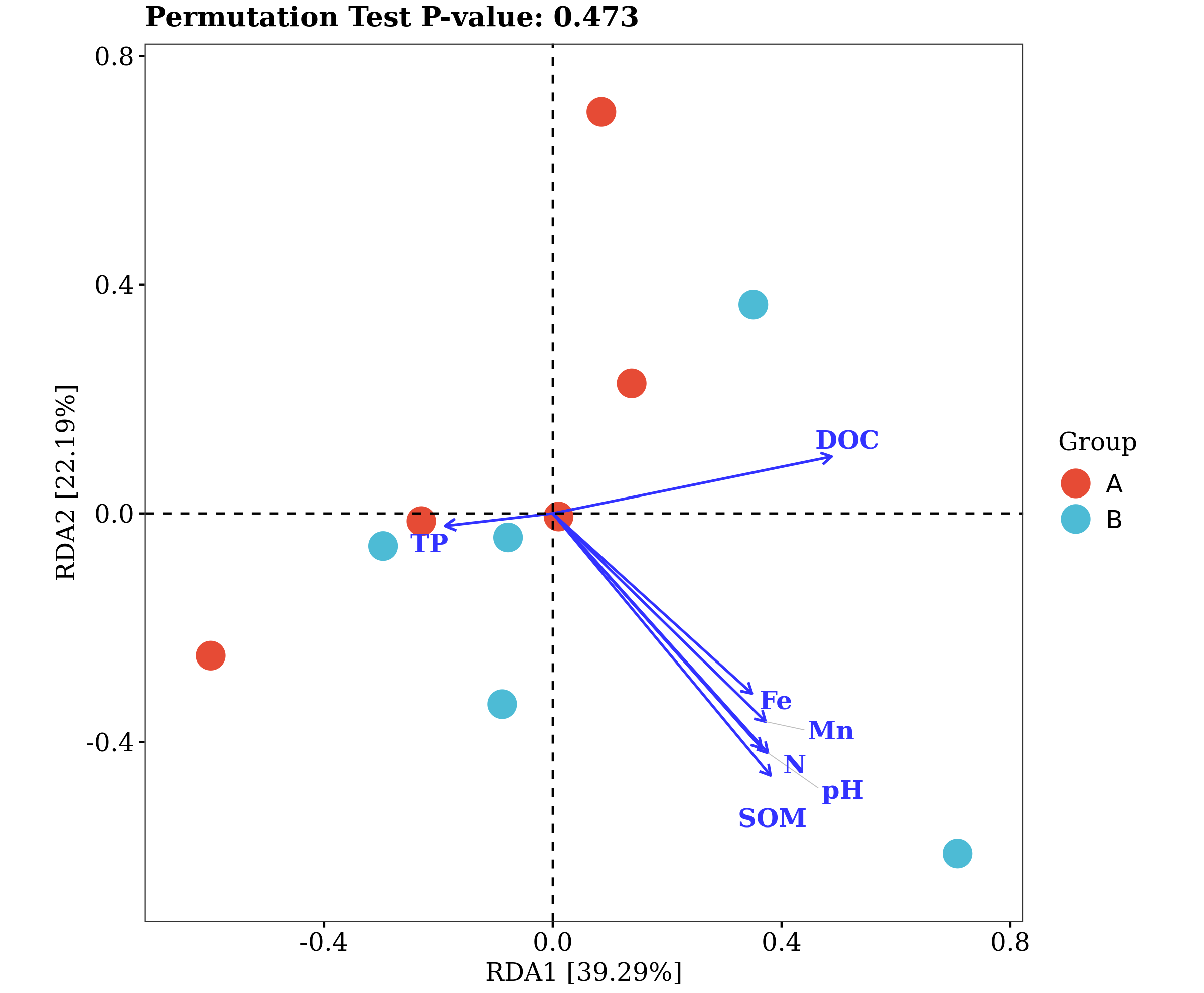
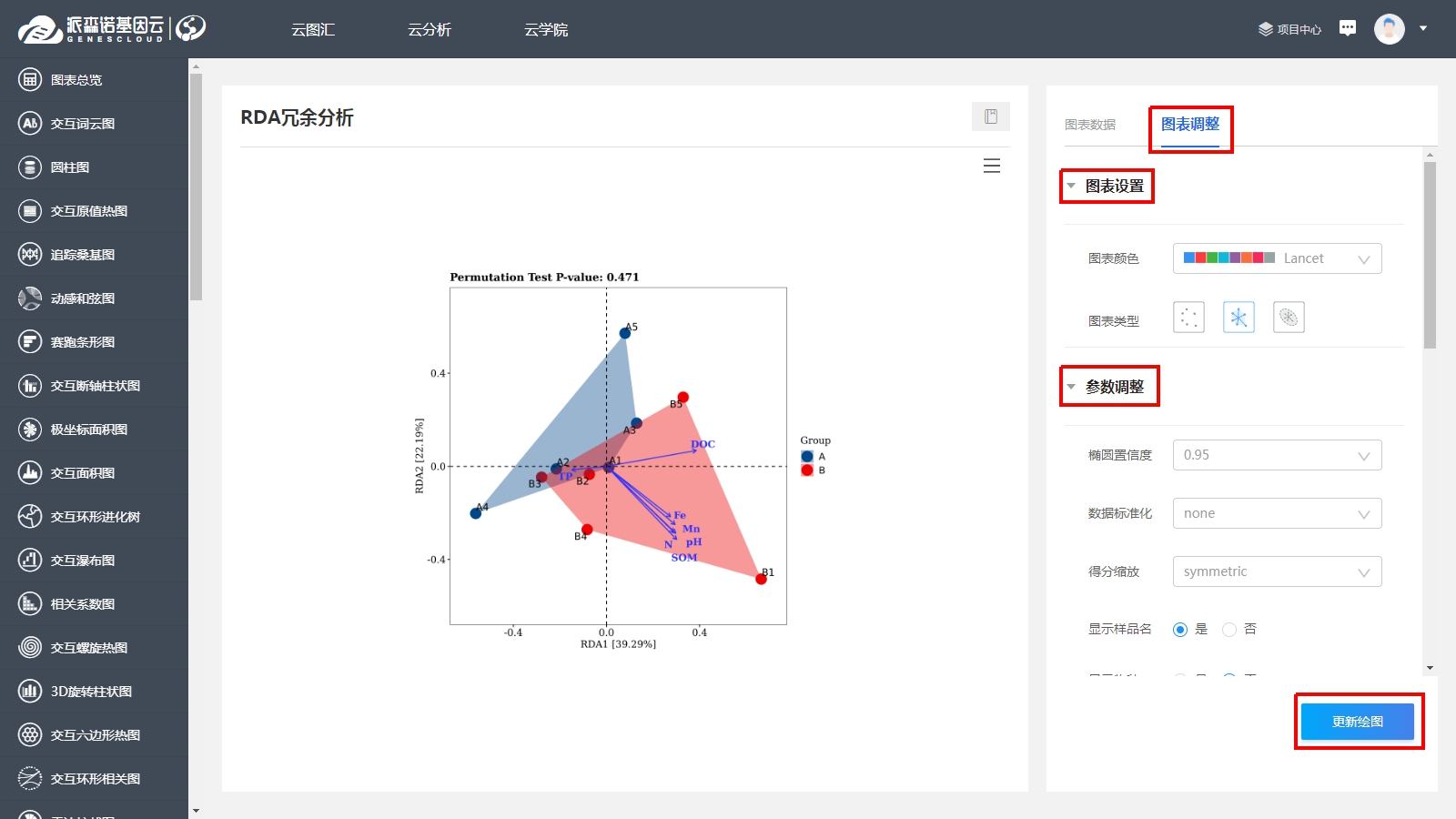
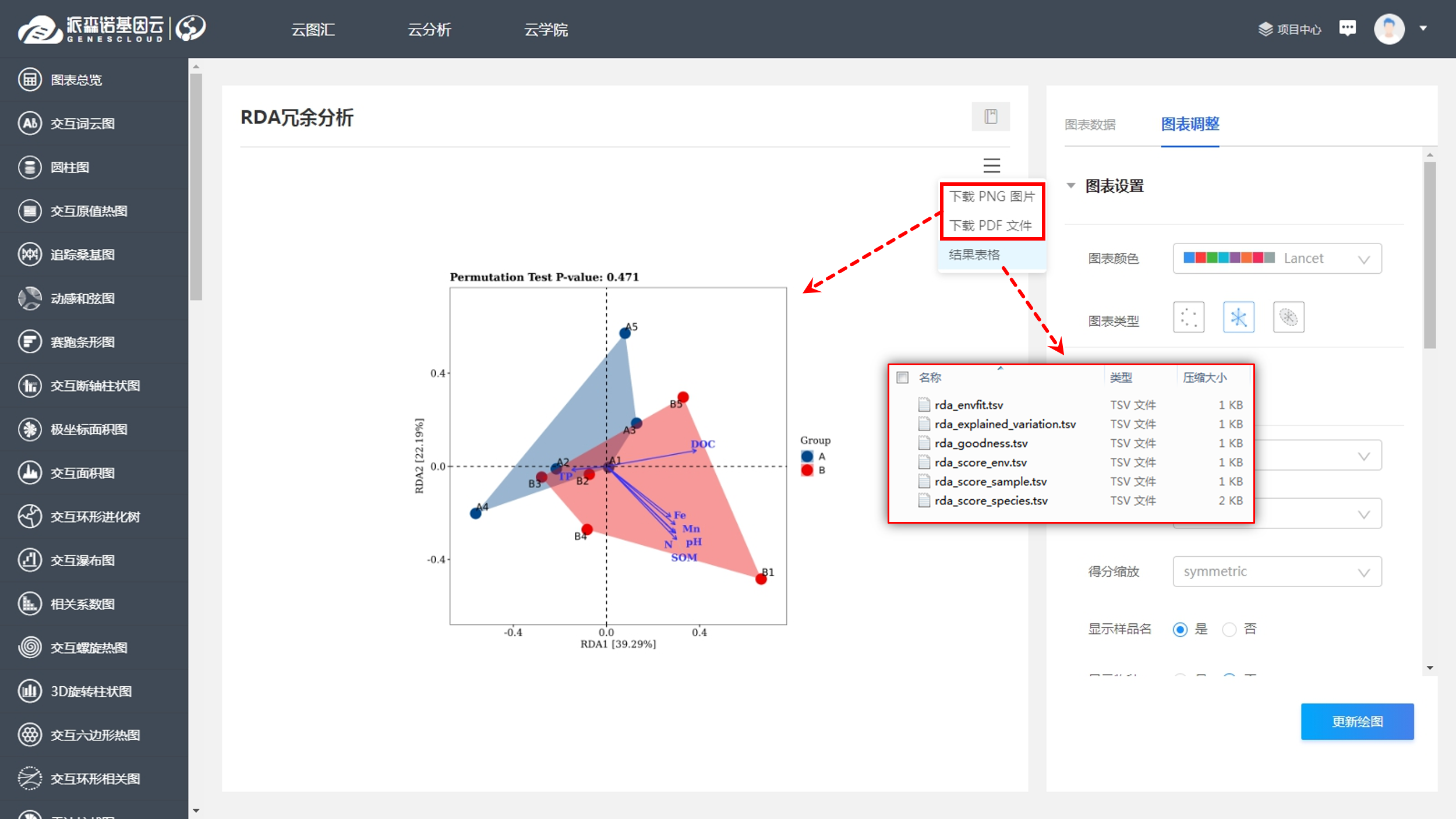
在對微生物群落進行研究時,除了分析不同群落樣本之間在微生物群落組成、功能上的差異外,往往還想了解,具體有哪些因素會影響微生物群落,使其組成和功能發生變化?其中,哪種因素又占據了主導作用? 為了回答這一問題,需要將微生物群落樣本的相關數據,與其對應的環境因子數據進行關聯分析。在眾多關聯分析的方法中,出鏡率最高的,就是RDA/CCA分析啦~ RDA/CCA分析一般需要使用CANOCO軟件或運用R語言進行分析,這對生信小白來說,挑戰還是很大滴。現在,RDA/CCA分析已經在派森諾基因云的“云圖匯”模塊中發布上線啦(網址:https://www.genescloud.cn/chart/)!我們也已做了初步介紹和解讀(點擊查看)。 簡單而言,RDA(Redundancy Analysis)和CCA(Canonical Correspondence Analysis)均屬于直接排序的分析方法,RDA基于線性模型,CCA則是基于單峰模型。一般情況下,兩者都能有效分析微生物群落與環境因子之間的變化相關性。 那么問題來了,既然兩者都可以進行,又該如何選擇呢? 在進行排序分析之前,可以先對物種群落數據進行去趨勢對應分析(Detrended correspondence analysis,DCA),根據結果中Lengths of gradient的值來進行判斷,如果最大的值大于4,則應選擇CCA,如果最大的值小于3,則選擇RDA,而如果最大的值在3~4之間,則兩種分析均可。然而,這種標準并不是100%合適,在實際的使用中,我們最好是同時進行CCA和RDA,根據結果進行選擇~ 1、數據準備 需要準備物種組成數據(data.txt)、環境因子數據(factor.txt)、分組數據(group.txt),具體如下: 注意:在準備數據時,不能出現中文、空格、特殊字符如()、?、*、[ ]、$等。最后需要把整理好的Excel文件另存為“制表符分隔”的txt格式文件。 ① 物種組成豐度數據(data.txt) ● 通過網盤下載數據的項目:Treat*\2.2.2_taxa_summary\ungrouped\otu_table_L*.xls文件,對應門綱目科屬種等不同分類水平下的物種組成豐度表(QIIME2版本); ● 通過基因云獲取文件的項目:在下載鏈接中,下載“各分類學水平分類單元豐度表”。 以屬水平上的數據為例,將表格整理成如下形式(第一列為屬名,第一行為樣本名)后,另存為txt文檔: ② 環境因子數據(factor.txt) 根據各樣本環境因子的測定結果,可將其整理成如下形式(第一列為樣本名,第一行為因子名),整理好后,同樣另存為txt文檔。 注意:環境因子的個數應小于樣本的個數。 ③ 樣本分組文件(group.txt) 根據分組將樣本整理成如下形式(第一列為樣本名稱,第二列為分組情況),整理好后,同樣另存為txt文檔。 2、將整理好的三個文件上傳至“云圖匯” ●以RDA為例(https://www.genescloud.cn/chart/RDA),在“圖表數據”標簽中,點擊“文件上傳→本地文件”,即可選擇剛才我們已經整理好的文件進行分析。 ● 待3個文件均上傳成功后,點擊“提交繪圖”,就可以輕松得到分析結果啦!是不是很簡單呢? ● 隨后,我們還可以在“圖表調整”模塊中,對樣本顏色、分析參數等進行進一步的調整優化哦,調整完點擊“更新繪圖”,就可以看到結果啦~ ● 最后,我們可以點擊下載結果圖片及表格文件哦~ 以上就是今天給大家介紹的云圖匯RDA/CCA操作指南了,當然,作圖效果的好壞,很大程度上還是取決于分析結果是否理想、微生物群落是否和環境因子間真的存在關聯(例如,上圖中的P值是不顯著的)。 歡迎大家進入基因云嘗試使用哦~也歡迎大家在討論區留言或者發郵件給我們喲(郵箱地址:[email protected])!