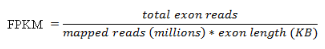2021-01-27
很多小伙伴拿到轉錄組結果,都會有一個大大的疑問,明明上一步已經計算了FPKM了,為什么差異分析還是用readcount來做,我想用FPKM行不行?
這里,鄭重地回答你:不可以且沒必要!你熟悉的DESeq,DESeq2和EdgeR都表示不同意!他們都只認readcount!
首先,先讓你和老朋友FPKM再重新熟悉一下:由于不同樣品過濾后獲得的數據量是不可能完全一致的,不同基因長度也有很大差異。因此為了能夠在樣品內比較基因的表達量,需要采用FPKM 對表達量進行標準化(Normalization):FPKM(Fragments Per Kilobase Million),為每百萬 Reads 中來自某一基因每千堿基長度的 Reads 數目,是一種普遍采用的基因表達量標準化方法,這種方法同時考慮了測序深度和基因長度對基因表達量計數的影響。其計算公式如下:
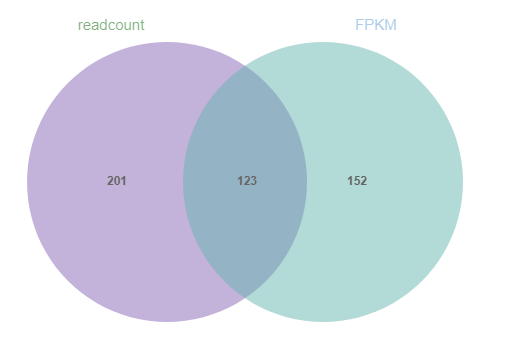
敲黑板!FPKM考慮到基因長度對基因表達量計數的影響,但是在進行差異分析時,同一個基因在不同樣本中的表達差異根本不需要考慮這條基因的長度!!!差異分析需要均一化的是不同處理中的基因,FPKM均一化的則是這個樣本中的所有基因,硬要讓FPKM去做不屬于他的工作,小心出現大問題!口說無憑,這里我們隨機抽取了同一個項目的兩組幸運數據,每組3個重復,直接來驗證一下: 同樣的兩組數據,我們分別用readcount和FPKM進行差異分析,分別得到了324個和275個“差異基因”,光看數目是不是好像差別不大?可是這些基因中,只有123個是相同的,所以如果你用FPKM強行做了差異分析,那么得出的結果,可與用readcount的差異分析結果大相徑庭。你以為得到的是孫悟空,其實根本就是披著孫悟空馬甲的六耳獼猴! 這里可能小伙伴又疑惑了起來,FPKM又不能做差異,我要他有什么用??? No No No,想要比較同一個樣本中所有基因誰的表達量更高更強,還是要FPKM出馬。以及你熟悉的樣品相關性分析、熱圖和WGCNA,他們通通都需要FPKM的支持! 因此,分析中無論是選用FPKM,還是readcount,都是經過統計學家,軟件開發人員和分析人員的反復驗證,深思熟慮選用的,所以如果你的結果還是不太滿意,是不是也想想實驗? 以上結果僅供參考!