2020-12-18

12月2日,派森諾基因云“云圖匯”模塊中上線了一批精品分析工具,不知各位小伙伴是否有嘗鮮呢?【12月的第一批高效精美作圖小工具熱血上線,你get了嗎?】(點擊查看)今天我們先來了解下RDA/CCA/PCoA_envfit這幾個分析內容。
對于微生態領域(多樣性組成譜/宏基因組/宏轉錄組等)的測序數據,除了通過差異統計分析來篩選各組樣本中的微生物標志物種或基因(Biomarker)的信息外,我們還需要將其它各種檢測手段獲得的數據,比如代謝組、蛋白組、或檢測獲得的理化指標、臨床指標等各類數據,與微生物組的海量數據進行關聯分析,以期找出與各類指標變化相關聯的具體微生物物種及其基因。這種研究思路,統稱為全微生物組關聯分析(Microbiome-wide association study,MWAS)。目前已有多種算法,可以幫助我們進行上述多組學數據的聯合分析。
RDA冗余分析(Redundancy analysis)是一種典型的約束排序方法,可以理解為“有約束條件”的PCA分析,通過多元線性回歸(Multiple linear regression,MLR),將菌群結構數據與某一種或多種給定的影響因素互相擬合,并通過置換檢驗來判斷這些因素對于菌群結構的影響是否顯著。
CCA典型關聯分析(Canonical Correlation Analysis)與RDA分析相似,亦是最常用的挖掘數據關聯關系的算法之一。其中RDA分析是基于線性模型,CCA分析則是基于單峰模型(通常可以兩種分析模型都做下嘗試;有動手分析能力的小伙伴,還可以通過R-Vegan包中的decorana函數,進行DCA分析,如果DCA排序前4個軸中最大值超過4,選擇單峰模型;如果是小于3,則選擇線性模型;如果介于3與4之間,兩者都可以哦~)。
但當我們利用約束排序方法(如RDA或CCA分析等),發現環境變量的解釋程度較低、物種與環境變量間的排序結果較為混亂、組間差異不明顯時,說明約束排序模型并不適用;這時我們可以考慮通過非約束排序模型(PCoA、NMDS、PCA分析等)擬合環境變量的方式來解釋“環境—個體—菌群”間的關系,即這里提到的PCoA_envfit分析。該分析是基于樣品間距離矩陣(bray_curtis或unifrac等),通過排序分析獲得樣本在排序軸上的分布,接著利用回歸函數envfit進行環境因子與排序軸之間的擬合分析,最后通過置換檢驗(permutation test)進行顯著性分析,一般默認選擇重復999次,獲取相關數據及繪圖。
我們在派森諾基因云的“云圖匯”模塊中,已經發布了這三種分析作圖的工具。同時,我們也在“云學院”的“云文檔”中,提供了具體操作方法,小伙伴可以按照提示,來操作嘗鮮哦!
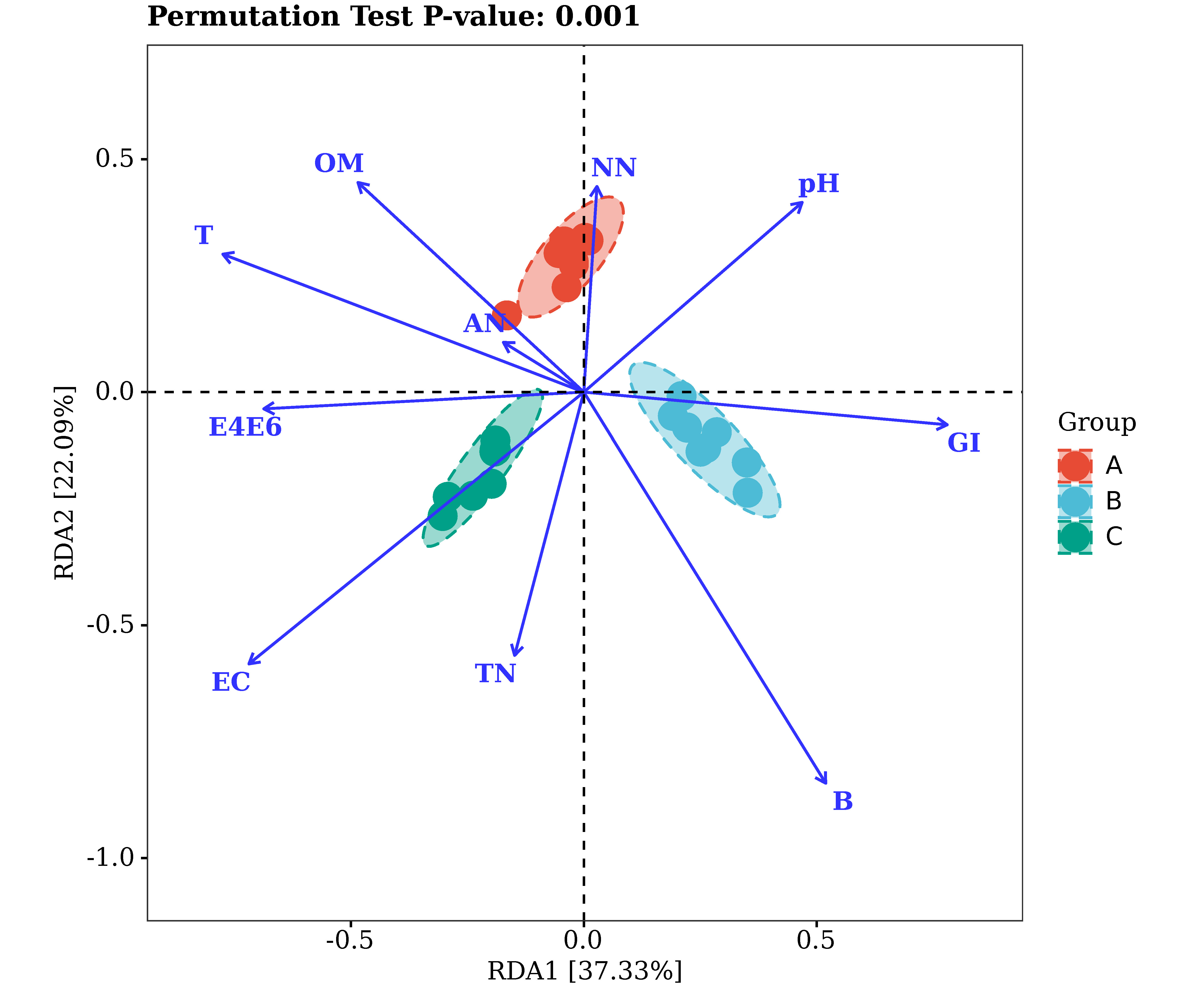
派森諾基因云RDA分析
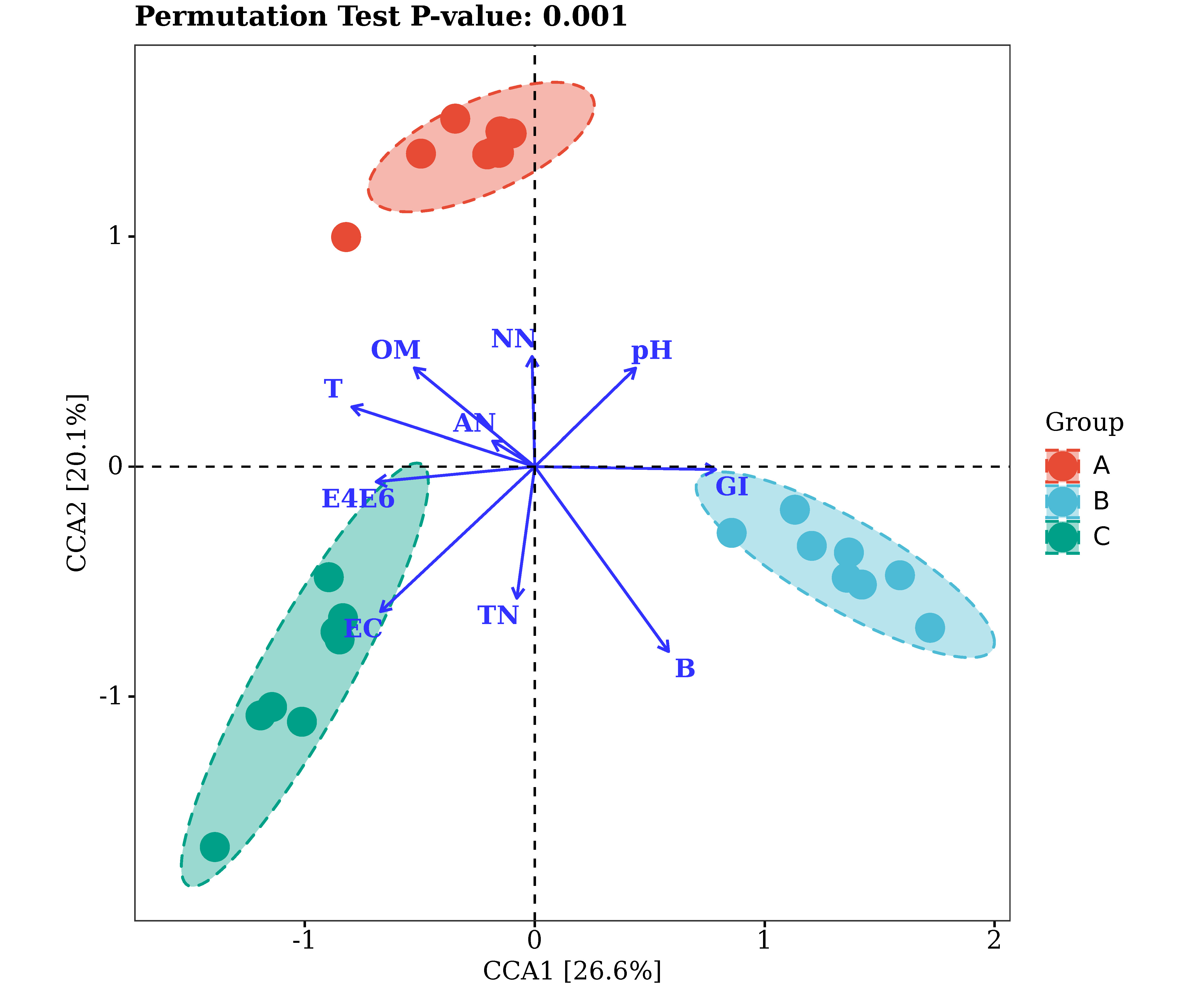
派森諾基因云CCA分析
注:圖中,每個點代表一個樣本,不同顏色的點屬于不同分組,兩點之間的距離越接近,說明兩個樣本的菌群組成/功能相似度越高。藍色箭頭分別代表不同的影響因素,影響因素之間的夾角代表它們之間相關性的大小,銳角表示兩個因素正相關,直角為不相關,鈍角時為負相關,射線越長,表明該因素對菌群組成/功能的影響作用越大;藍色箭頭射線和坐標軸的夾角代表某個環境因子與坐標軸的相關性大小,夾角越小,相關性越高。樣本在藍色箭頭上投影點的位置,近似代表該因素在對應樣本中的數值大小;排序圖上方的P值代表采用蒙特卡洛置換檢驗獲得的P值,P值越小,影響因素對于菌群組成/功能的作用越顯著。坐標軸括號中的百分比代表了對應的坐標軸所能解釋的原始數據中差異的比例。
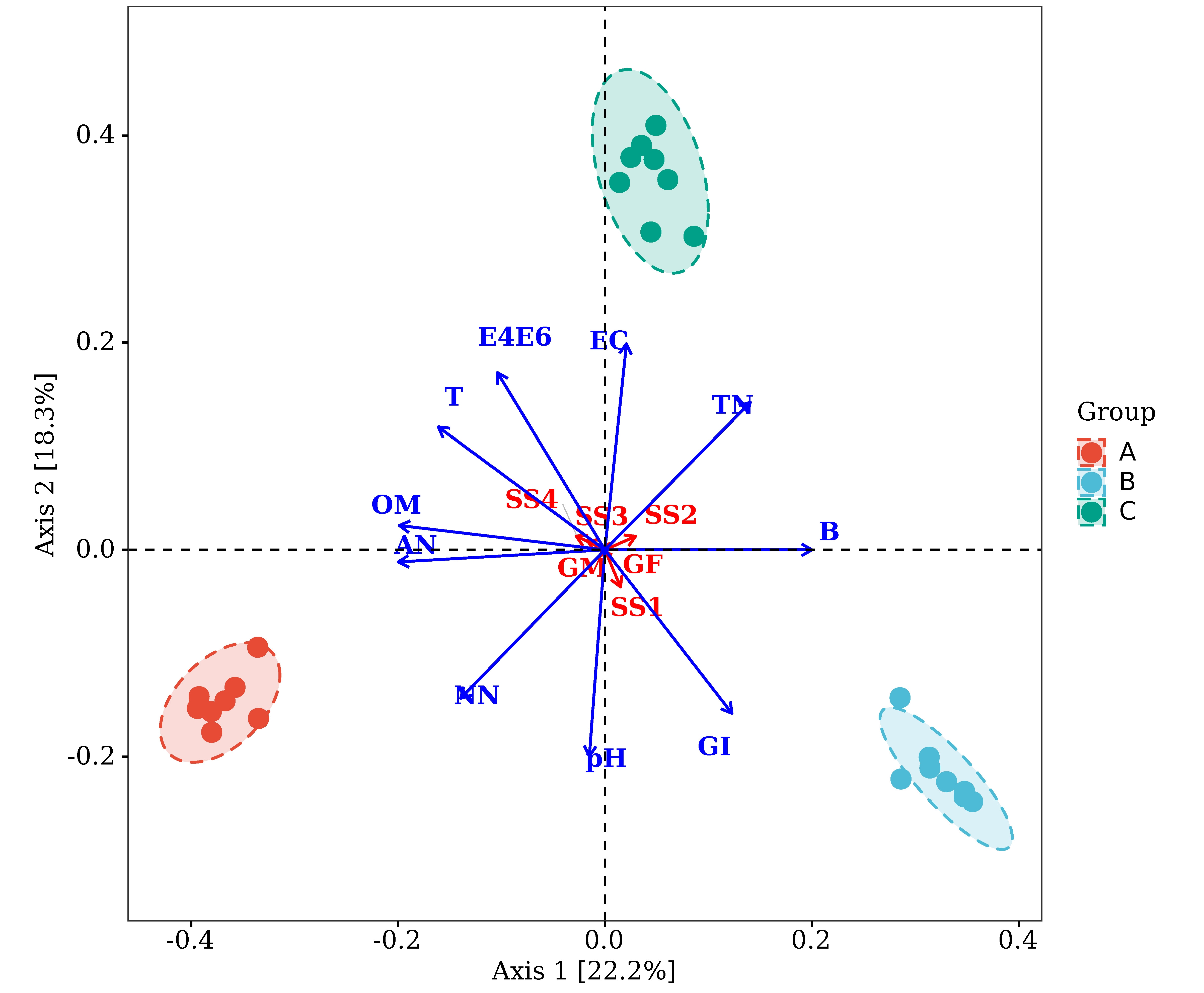
派森諾基因云PCoA_envfit分析
注:圖中,每個點代表一個樣本,不同顏色的點屬于不同分組,兩點之間的距離越接近,說明兩個樣本的菌群組成/功能相似度越高。藍色箭頭(數值變量:如pH、濃度等)與紅色箭頭(性狀變量:如性別、部位、季節等)分別代表連續型和離散型的影響因素,影響因素之間的夾角代表它們之間相關性的大小,銳角表示兩個因素正相關,直角為不相關,鈍角時為負相關,射線越長,表明該因素對菌群組成/功能的影響作用越大;箭頭射線和坐標軸的夾角代表某個環境因子與坐標軸的相關性大小,夾角越小,相關性越高。樣本在箭頭上投影點的位置,近似代表該因素在對應樣本中的數值大小;坐標軸括號中的百分比代表了對應的坐標軸所能解釋的原始數據中差異的比例。
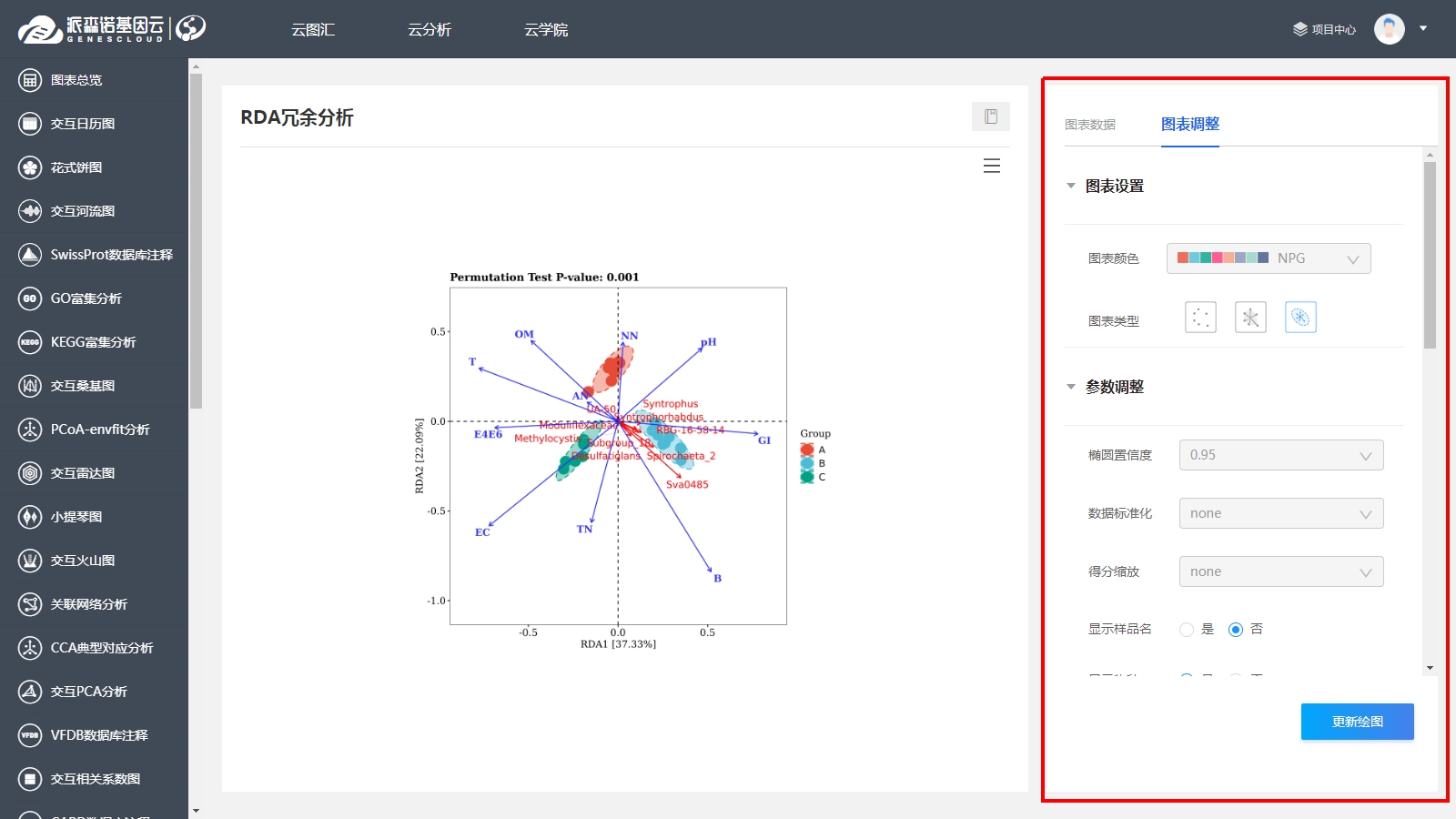
作圖完成后,還可以通過右邊側欄的“圖表調整”,進一步優化作圖效果哦~
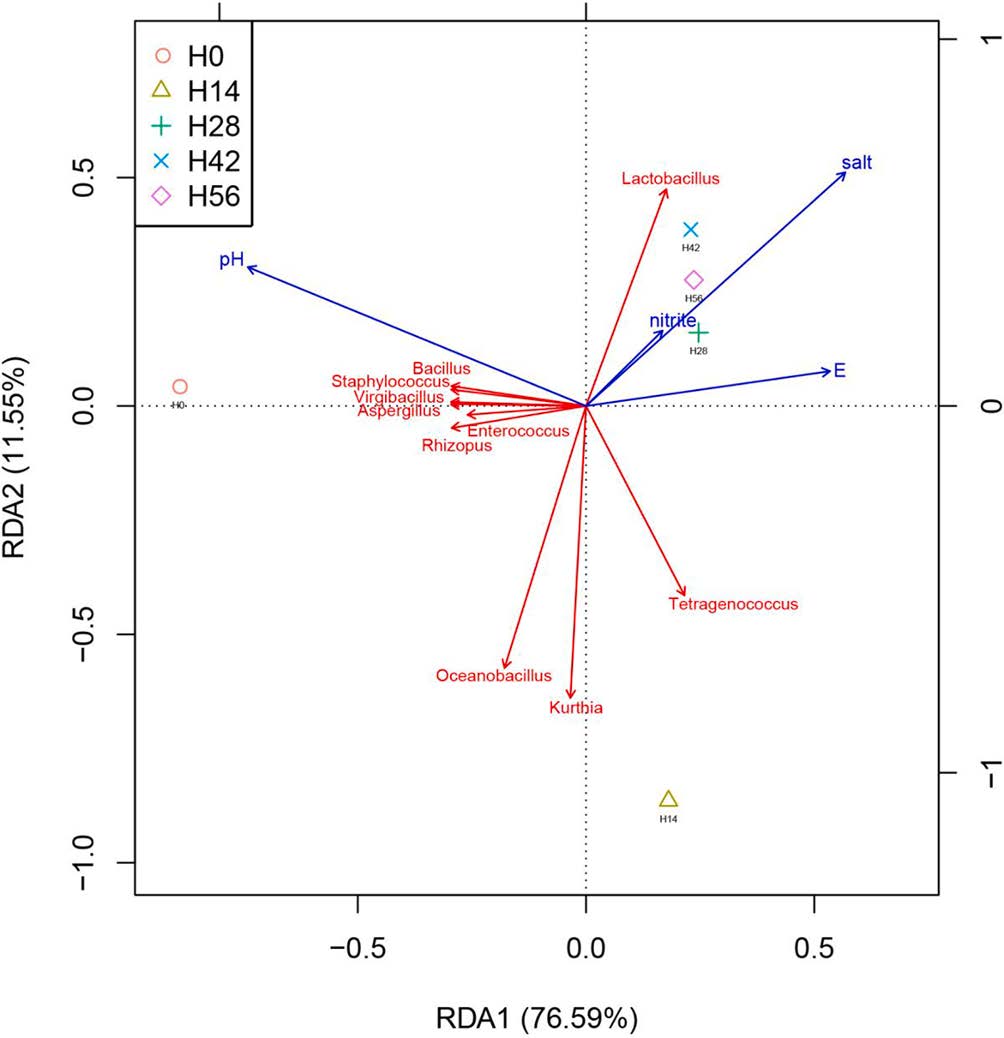
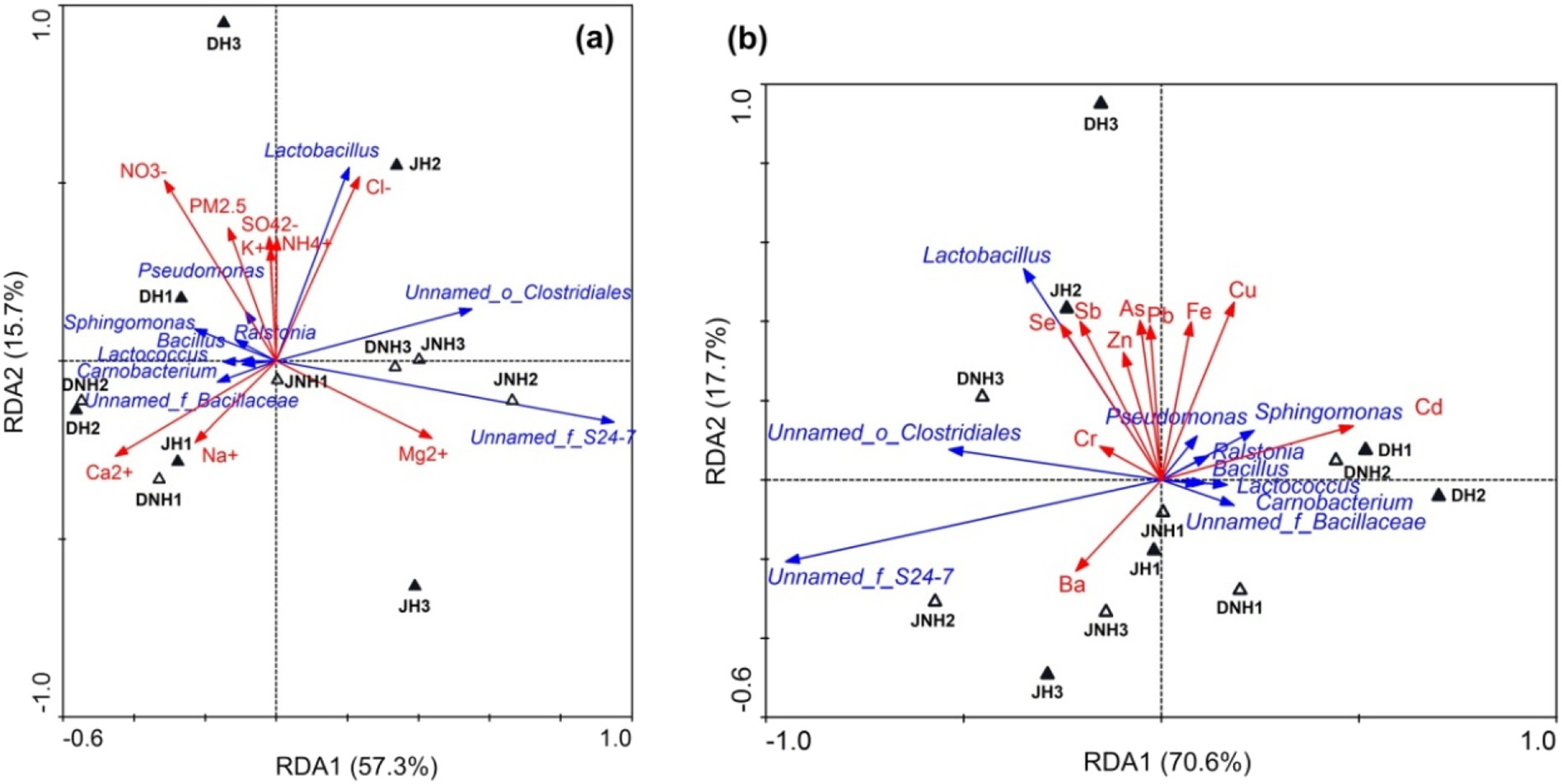
RDA/CCA/PCoA_envfit作為最常見的“環境—個體—菌群”的關聯分析方法,已經在諸多研究中廣泛應用,我們在這里也拋磚引玉,列舉一二,供大家參考~


對于以上的分析內容,小伙伴們是否心動了呢?心動不如行動,快來派森諾基因云(https://www.genescloud.cn/)體驗吧!


