
2020-10-12
近年來,隨著DNA/RNA、蛋白質(zhì)以及代謝物檢測分析技術的迅速發(fā)展,以及計算能力的飛速提升,聚焦于微生物組的研究正以指數(shù)級的速度迅猛增長。作為目前當紅的研究熱點,微生物組學可謂“集萬千寵愛于一身”,已廣泛應用于人體/動物、自然/工業(yè)等各種環(huán)境體系的微生物群落研究中。
微生物組、微生物群、宏基因組、代謝組……面對一長串令人眼花繚亂的概念術語、研究技術、分析方法,您是不是也會感到困惑,不知從何入手呢?本期的微生物組問答集錦,就將為您答疑解惑,避免混淆和誤解,助您快速入門微生物組!
1、微生物組和微生物群,傻傻分不清楚?
微生物組(Microbiome),指生態(tài)群落中的全體微生物(細菌、古細菌、低等或高等真核生物和病毒等各種生命體形式)的基因組(基因集合),及其在所處生境內(nèi)所產(chǎn)生的全部物質(zhì)信息。這個定義是基于Biome(生物群落)而形成的,囊括了生態(tài)環(huán)境中的所有生物和非生物因素。
與Microbiome相對的是,Microbiota常用來指代“微生物群”,指的是特定環(huán)境中存在的微生物群體的集合。早期有研究使用Microflora來表述“微生物群”的概念,但這一詞匯已經(jīng)不適用于微生物組研究領域,被時代淘汰了。
2、微生物組研究主要使用哪些高通量測序技術?
目前普遍采用二代Illumina和三代PacBio測序技術開展微生物組研究。其中,二代Illumina測序技術根據(jù)通量、讀長等技術指標,有MiSeq、NovaSeq等測序系統(tǒng),采用的是雙末端(Paired-end,PE)測序,即在待測DNA片段的正反兩條鏈上,各自從5’端開始,測定相同堿基數(shù)的序列,目前常見的雙末端測序模式有PE150、PE250和PE300(也可表示成2×150、2×250和2×300,150/250/300是指雙末端各自測定的堿基數(shù));而三代PacBio測序則通過單分子實時測序技術(Single molecule real-time sequencing,SMRT),對DNA序列實現(xiàn)單分子級別的超長讀長測序,并通過環(huán)形一致性測序模式(Circular-consensus sequence,CCS),極大地提高單堿基測序的準確率,因而具有無需PCR擴增富集、超長讀長、堿基檢測無偏好性和系統(tǒng)性偏差、一致性準確率高、原位實時直接檢測DNA修飾等優(yōu)勢。
目前,二代Illumina測序技術可用于多樣性組成譜測序(以PE250/PE300測序模式為主)和宏基因組/宏轉錄組測序(以PE150測序模式為主),而三代PacBio測序技術主要應用于多樣性組成譜測序(通量不及二代Illumina測序,但能測定目標基因的全長信息,因而對于優(yōu)勢物種的注釋而言更為精準)。
3、什么是16S rRNA基因?ITS序列又是什么?
16S rRNA基因指原核微生物(細菌和古菌)染色體上編碼核糖體(Ribosome)RNA的16S(S是沉降系數(shù))小亞基(Subunit)的基因序列。該基因存在于所有原核微生物的基因組中,相當于一個“分子時鐘”,序列會隨著物種的多樣化而發(fā)生進化。它既含有高度保守的序列區(qū)域,又有中度保守和高度可變的序列區(qū)域。利用保守區(qū)序列設計引物,從而能夠?qū)?6S rRNA基因片段進行PCR擴增并測序,這樣就能根據(jù)可變區(qū)序列的差異,對不同種屬的微生物進行物種鑒定和分類。

細菌/古菌16S rRNA基因常用測序引物及其對應區(qū)域

三代PacBio平臺和二代Illumina平臺的測序模式示意圖
ITS序列全稱為Internal transcribed spacer,即內(nèi)轉錄間隔區(qū)序列,是真核生物核糖體RNA(rRNA)基因非轉錄區(qū)的一部分,常用于真菌類群的物種鑒定和群落組成分析。用于真菌物種鑒定的ITS包括ITS1和ITS2兩個區(qū)域。其中,ITS1位于真核生物18S rRNA基因和5.8S rRNA基因兩個亞基之間,ITS2則位于真核生物5.8S rRNA基因和28S rRNA基因兩個亞基之間。
真菌ITS及真核生物18S rRNA基因常用測序引物及其對應區(qū)域
(https://unite.ut.ee/primers.php)
4、什么是宏基因組學?
宏基因組(Metagenome)是指微生物群落中,全體成員的基因組和遺傳信息。而宏基因組學(Metagenomics),就是描述宏基因組特征的方法手段,從中我們可以獲得與微生物群落潛在功能相關的信息。通常,我們對微生物組DNA片段化后進行鳥槍法測序,收集所得序列數(shù)據(jù)并進行序列組裝和基因注釋,獲得群落中各種微生物(包括但不限于:細菌、真菌、病毒等)的基因組序列,從而解析該生態(tài)系統(tǒng)下所有微生物的物種分布特征和功能特性信息。因此,宏基因組學研究能全面精細地展示微生物群落整體性的功能代謝譜和物種精細組成譜,從原理上闡明微生物群落在生態(tài)系統(tǒng)中發(fā)揮作用的根本機制。
5、什么是宏轉錄組學?
宏轉錄組學(Metatranscriptomics),的研究對象是微生物組mRNA,通過對特定時間、空間和條件狀態(tài)下,采集的樣本中所有微生物群落的轉錄本進行大規(guī)模高通量測序,可以直接獲得樣本中可培養(yǎng)和不可培養(yǎng)的各種微生物的轉錄組及其表達量信息,從而能精確定量整個菌群中具有活性的物種精細組成及其對應功能的表達水平,進而鎖定菌群中的關鍵生物標記物、闡明其生物學意義。因此,宏轉錄組研究可以檢測微生物群落中活性物種的分布特征及其表達轉錄的功能特性信息,同時可以比較不同時間、空間和條件狀態(tài)下的差異表達基因和差異功能途徑,從而揭示微生物群落在不同環(huán)境壓力下的調(diào)節(jié)適應機制,探索“微生物組——環(huán)境/宿主”之間的互作機理。
與宏基因組學相比,宏轉錄組學是在RNA水平研究微生物組的表達譜信息,因而能提供微生物組在環(huán)境響應以及表達調(diào)控層面的更為真實的數(shù)據(jù)結果,能有效避免群落中死亡或休眠的微生物的影響,還能避免微生物基因組上未真正發(fā)揮作用的基因的干擾。
6、對于微生物組測序研究,生物學重復應該如何設計?
生物學重復的總體設計原則就是:多多益善。一般而言,微生物群落樣本總是存在一定的隨機性和不均一性,而設置生物學重復將有助于評估樣本間差異程度,同時增強結果的可靠性。測序的樣本數(shù)越多,越能夠降低組內(nèi)樣本差異而引入的影響。此外,足夠數(shù)量的生物學重復,還有助于檢測離群樣本,因為異常樣本的存在,會嚴重影響檢測結果的準確性。在生物學重復數(shù)量足夠的情況下,通過分析菌群組成、Alpha和Beta多樣性指數(shù),可以發(fā)現(xiàn)異常樣本,將其剔除。
如果樣本的類型是動植物相關樣本,如腸道內(nèi)容物或糞便樣本、組織樣本等,個體間差異較大,建議至少10~20個生物學重復;如果樣本的類型是環(huán)境樣本,如土壤、水體、空氣等,建議至少6~8個生物學重復;提高生物學重復的數(shù)量可以大大減小生物個體間存在的誤差。
7、有生物學重復和無生物學重復,對于微生物組分析有哪些差異?
對于微生物組研究而言,有無重復,判斷差異物種或差異基因的統(tǒng)計檢驗方法是不同的。一般而言,在有生物學重復的情況下,差異的評估是根據(jù)重復樣本來評估,統(tǒng)計檢驗的方法更為多樣,分析手段更為豐富(如常見的Adonis分析、LEfSe分析、隨機森林分析等);而在無生物學重復的情況下,差異只能通過單獨樣本的菌群組成豐度或基因豐度來估計,容易產(chǎn)生偏差,分析方法的選擇很少。
8、微生物組學研究實驗設計,主要有哪些關注點?
一般而言,微生物組研究可以分為橫向和縱向兩種主要的研究思路。橫向研究主要是指多個不同的平行處理之間的相互比較,而縱向研究一般是指多個不同時期/階段之間的動態(tài)變化。在設計實驗時,首先需要確定采取的研究思路,當然也可以橫向、縱向研究相結合。
此外,微生物組學研究實驗設計還應遵循以下原則:
1)取樣有代表性。不同類型的微生物群落,其特征差異很大,可能樣本采集所需的方式、位置和質(zhì)量體積都不一樣。由于環(huán)境條件的變化對微生物生長的影響很大,因此要求不同樣本的采集條件、方法保持高度一致,比如對于溫濕度、光照條件、取樣深度的控制(尤其是多時間分批次收集的樣本)。另外,大部分微生物群落的本身分布存在一定的不均一性,需采集合適的樣點來盡可能反映樣本的總體真實面貌;
2)盡可能排除干擾微生物組檢測的潛在影響因素,如人體、動物等群體研究中,混雜的遺傳背景、分籠飼養(yǎng)等隱含于實驗設計因素外的干擾因素。取樣過程中,也要避免發(fā)生污染,包括如宿主污染、環(huán)境污染等,取樣時需要什么,就只取什么。同時,取樣所用的工具需經(jīng)過無菌處理;
3)盡可能多地收集各種影響微生物組的內(nèi)在/外在因素,比如樣本本身的理化特征數(shù)據(jù)/離子或氣體濃度等、人體/動物相關的生化數(shù)據(jù)、量化的表型數(shù)據(jù)等,以便與微生物組檢測數(shù)據(jù)進行全面的關聯(lián)分析,因而這些“元數(shù)據(jù)(Metadata)”收集得越全面越好,多多益善;
4)不同處理條件的各組樣本,包含盡可能多的生物學重復。
9、微生物組學研究流程,一般包括哪些環(huán)節(jié)?
微生物組學研究流程,主要包括研究設計、實驗檢測、數(shù)據(jù)分析和實驗驗證四大環(huán)節(jié)。
對于實驗測序環(huán)節(jié)而言,又可細分為:微生物組核酸提取(DNA或RNA)→測序文庫制備(對于多樣性組成譜測序,需要在PCR擴增后方可構建文庫)→上機測序等步驟;
數(shù)據(jù)分析環(huán)節(jié)可細分為:數(shù)據(jù)質(zhì)控和預處理→注釋分析(根據(jù)檢測方法的不同,可能涉及物種注釋、功能注釋和代謝注釋等)→生態(tài)學分析(如Alpha和Beta多樣性分析等)→多變量統(tǒng)計分析→標志物識別等步驟;
基于上述研究設計和數(shù)據(jù)分析結果,可以通過體外實驗、無菌動物模型等體內(nèi)實驗、熒光定量qPCR等策略,對候選得到的生物標志物進行進一步的實驗驗證,以明確特定微生物和宿主、環(huán)境之間的互作關系和因果鏈。
(https://www.nature.com/articles/d42473-020-00214-9)
10、什么是MWAS?
Microbiome-wide association study,即全微生物組關聯(lián)分析。對于微生物組研究,除了通過測序和差異統(tǒng)計分析來篩選各組樣本中的微生物標志物種或基因(Biomarker)的信息外,我們還需要將其它各種檢測手段獲得的數(shù)據(jù),比如代謝組、蛋白組、或檢測獲得的理化指標、臨床指標等各類數(shù)據(jù),與微生物組的海量數(shù)據(jù)進行關聯(lián)分析,以期找出與各類指標變化相關聯(lián)的具體微生物物種及其基因。這種研究思路,統(tǒng)稱為全微生物組關聯(lián)分析。
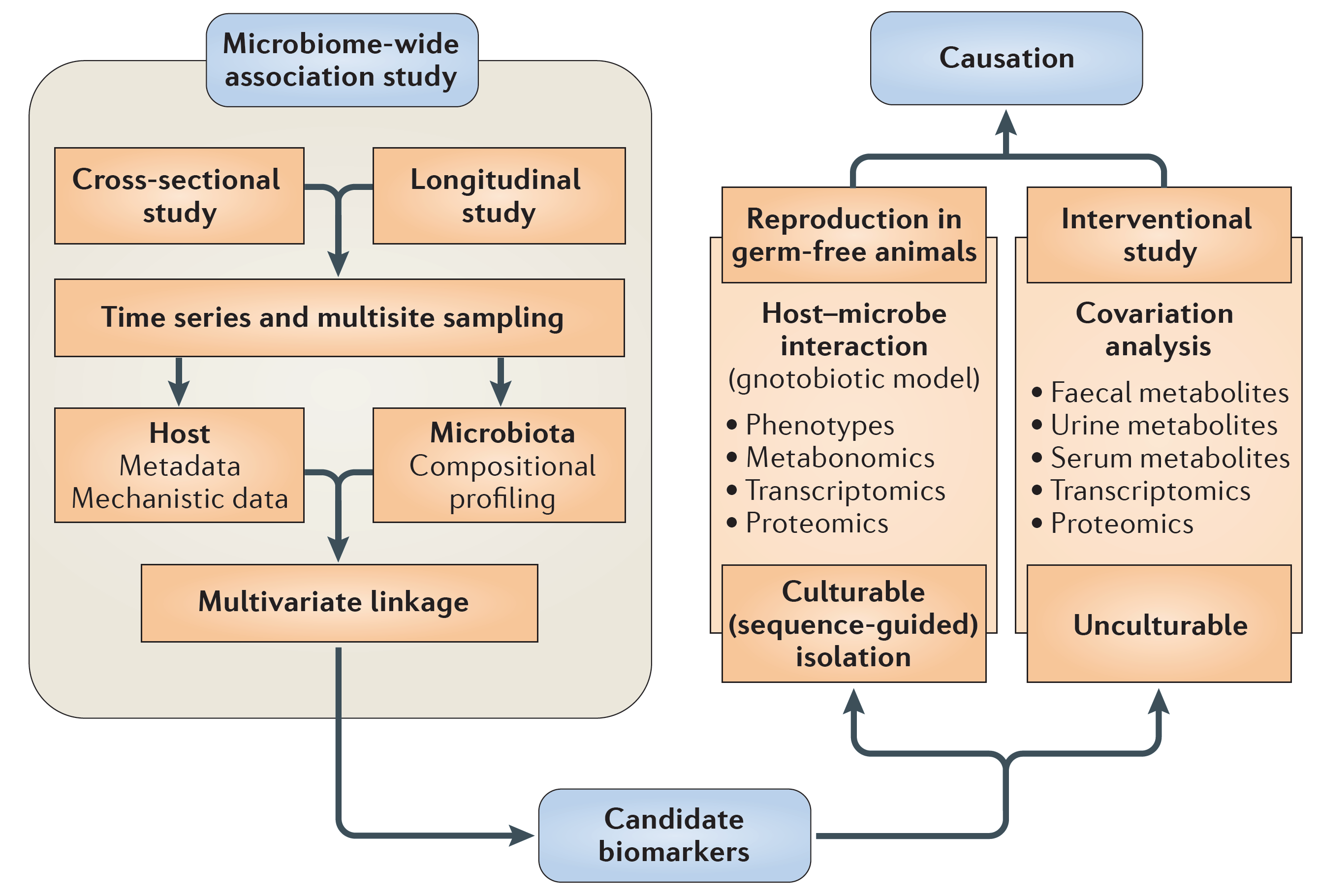
(Zhao, 2013, Nat Rev Microbiol 11, 639-647)
以上就是我們精心整理的微生物組學問答集錦,希望能夠成為大家開啟微生物組研究的敲門磚!我們后續(xù)還將帶來更多知識的梳理和分享,敬請期待哦!任何疑問,都歡迎反饋至[email protected],我們一定知無不言,言無不盡哦!