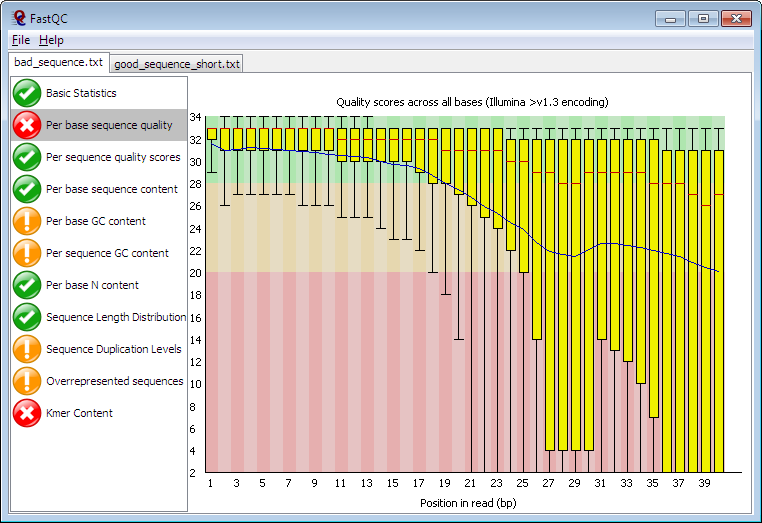
FastQC能全方位評(píng)估測序產(chǎn)生的原始數(shù)據(jù)的質(zhì)量,并通過網(wǎng)頁形式展示圖形化報(bào)告,是測序數(shù)據(jù)質(zhì)控的經(jīng)典工具。
https://www.bioinformatics.babraham.ac.uk/projects/fastqc/
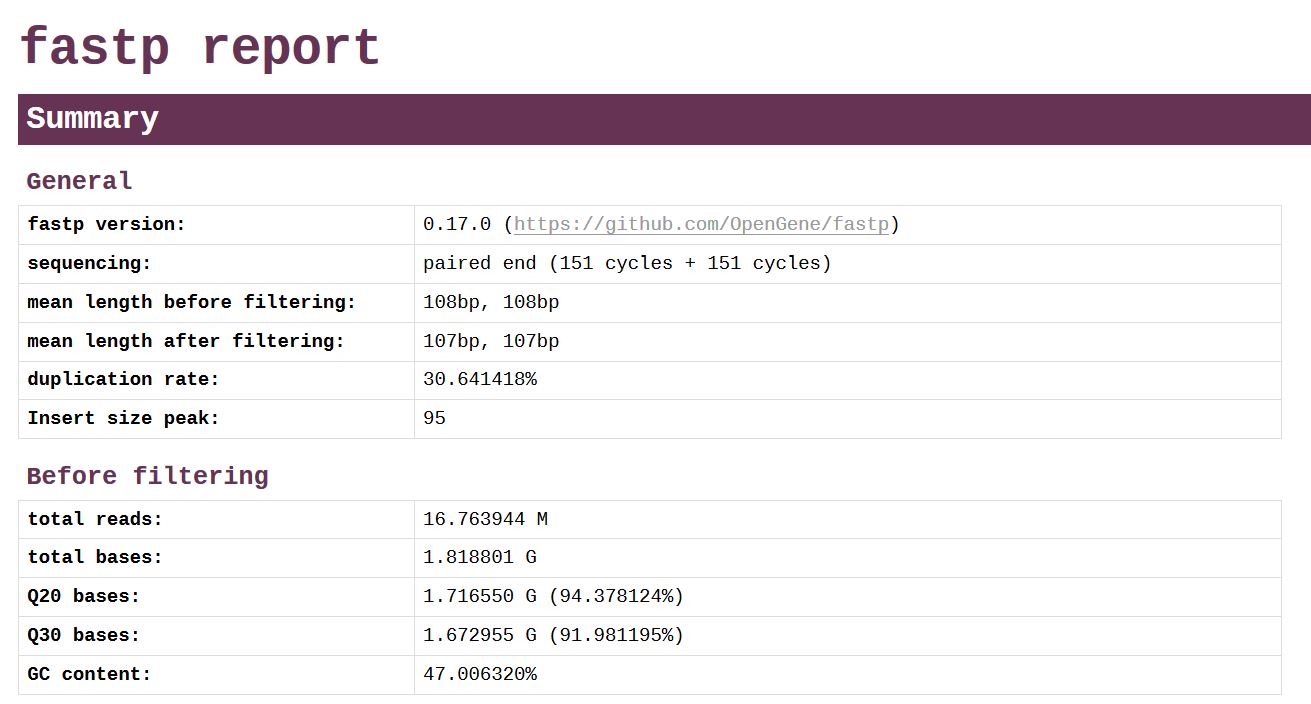
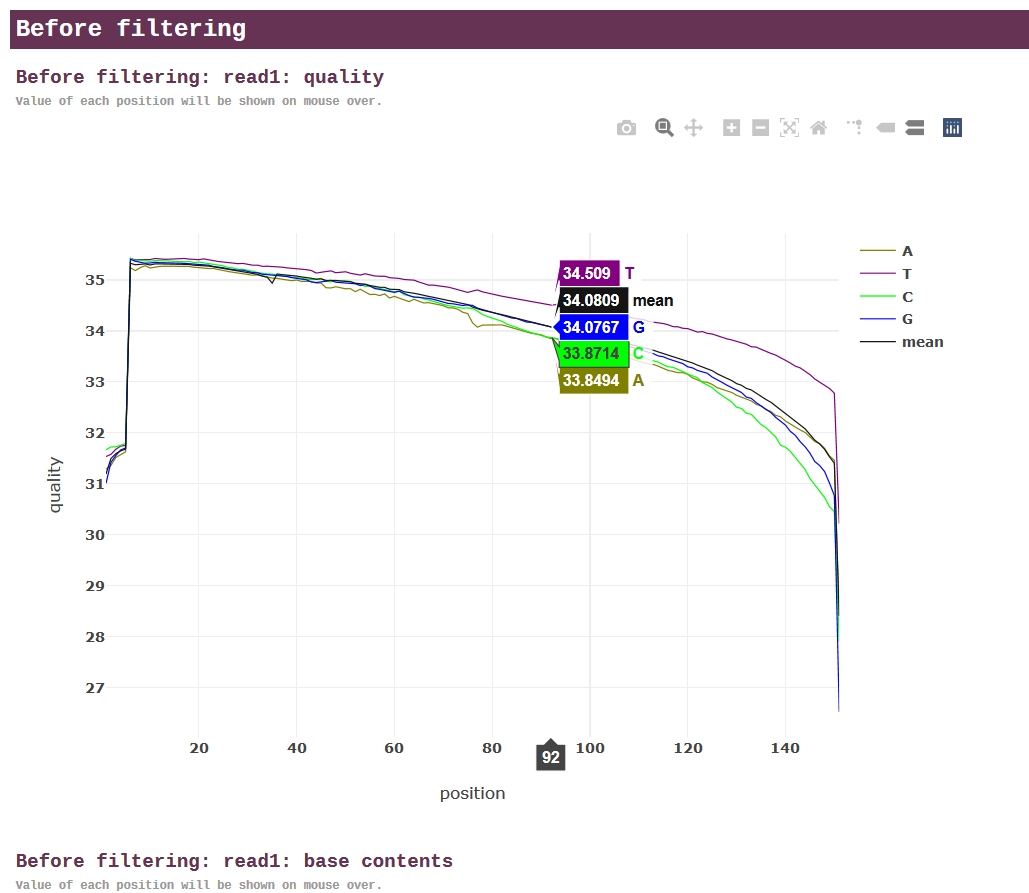
fastp是新近發(fā)表的測序質(zhì)控軟件,能對數(shù)據(jù)自動(dòng)進(jìn)行全方位質(zhì)控,并生成人性化的報(bào)告。與FastQC相比,fastp運(yùn)行速度快,功能多,號(hào)稱“all in one, one for all”。生成的報(bào)告中,所有的圖表都是使用JavaScript動(dòng)態(tài)繪制,交互功能非常強(qiáng)大。
https://github.com/OpenGene/fastp
對于存在宿主等污染的宏基因組/宏轉(zhuǎn)錄組測序數(shù)據(jù),可以使用BMTagger軟件將質(zhì)控后的高質(zhì)量序列與宿主基因組參考序列比對,并舍棄匹配上宿主的序列,以此來盡可能地去除宿主污染序列。
ftp://ftp.ncbi.nlm.nih.gov/pub/agarwala/bmtagger/
對于宏轉(zhuǎn)錄組或RNA類的宏病毒組數(shù)據(jù),SortMeRNA是去除測序結(jié)果中殘余的核糖體RNA(rRNA)序列的不二之選。
http://bioinfo.lifl.fr/RNA/sortmerna/
Kraken2是Kraken的升級(jí)版本,可以通過使用精準(zhǔn)k-mer匹配,對非拼接序列實(shí)現(xiàn)高精度且快速的物種注釋。Kraken2可同時(shí)適用于原核與真核微生物的識(shí)別,因而具有廣泛的應(yīng)用。
http://ccb.jhu.edu/software/kraken2/
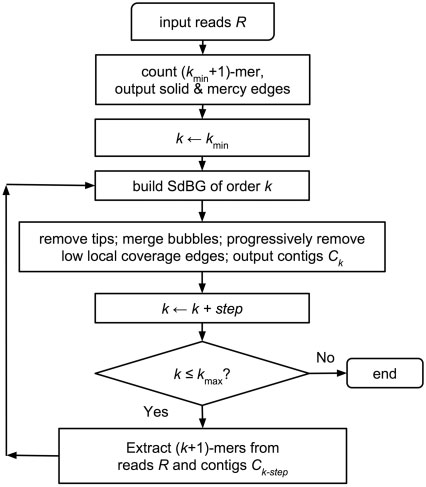
MEGAHIT是一款基于無損壓縮的Succinct de Bruijn graphs(SdBG)的快速組裝工具,因其具有內(nèi)存消耗小、計(jì)算速度快、低錯(cuò)誤率、安裝便捷、較長的重疊群長度等優(yōu)點(diǎn),在來源較復(fù)雜、數(shù)據(jù)規(guī)模較大的宏基因組序列的快速組裝上優(yōu)勢明顯,是一款應(yīng)用廣泛的主流組裝軟件。拼接組裝時(shí),可以根據(jù)樣品和數(shù)據(jù)特性,選擇meta-large或meta-sensitive等組裝模式。
https://hku-bal.github.io/megabox
DNA/mRNA序列快速比對神器,相較于BWA-MEM、Bowtie2等常用比對工具比對速度更快,內(nèi)存占用更少,可適用于二代、三代測序數(shù)據(jù)。
https://lh3.github.io/minimap2/
目前除了BLASTP之外最流行的蛋白比對軟件,比對速度快、內(nèi)存占用少、靈敏度高,可謂集萬千優(yōu)點(diǎn)于一身。
http://github.com/bbuchfink/diamond
對于基于拼接序列的物種注釋,由于并非所有Contigs序列都具有足夠的長度和特異性,它們可能會(huì)在注釋比對時(shí),同時(shí)匹配上多條參考序列,而這些匹配的參考序列又可能分屬于不同的物種分類單元;為使分析嚴(yán)謹(jǐn)可靠,同時(shí)又不丟失生物學(xué)意義,可以采用Blast2lca軟件的“最近共同祖先(Lowest Common Ancestor,LCA)”算法,將參考序列分化為不同物種分枝前的最后一級(jí)共同分類,作為目標(biāo)序列的物種分類注釋信息。該算法的原理與MEGAN軟件相同,是物種注釋的主流方法。
https://github.com/emepyc/Blast2lca
MetaGeneMark是專門用于預(yù)測原核微生物和宏基因組基因序列的老牌軟件,可以識(shí)別組裝得到的Contigs序列中的開放閱讀框,并預(yù)測其中的編碼區(qū)域,從而獲得對應(yīng)的基因和蛋白序列,運(yùn)算速度快。
http://exon.gatech.edu/GeneMark/
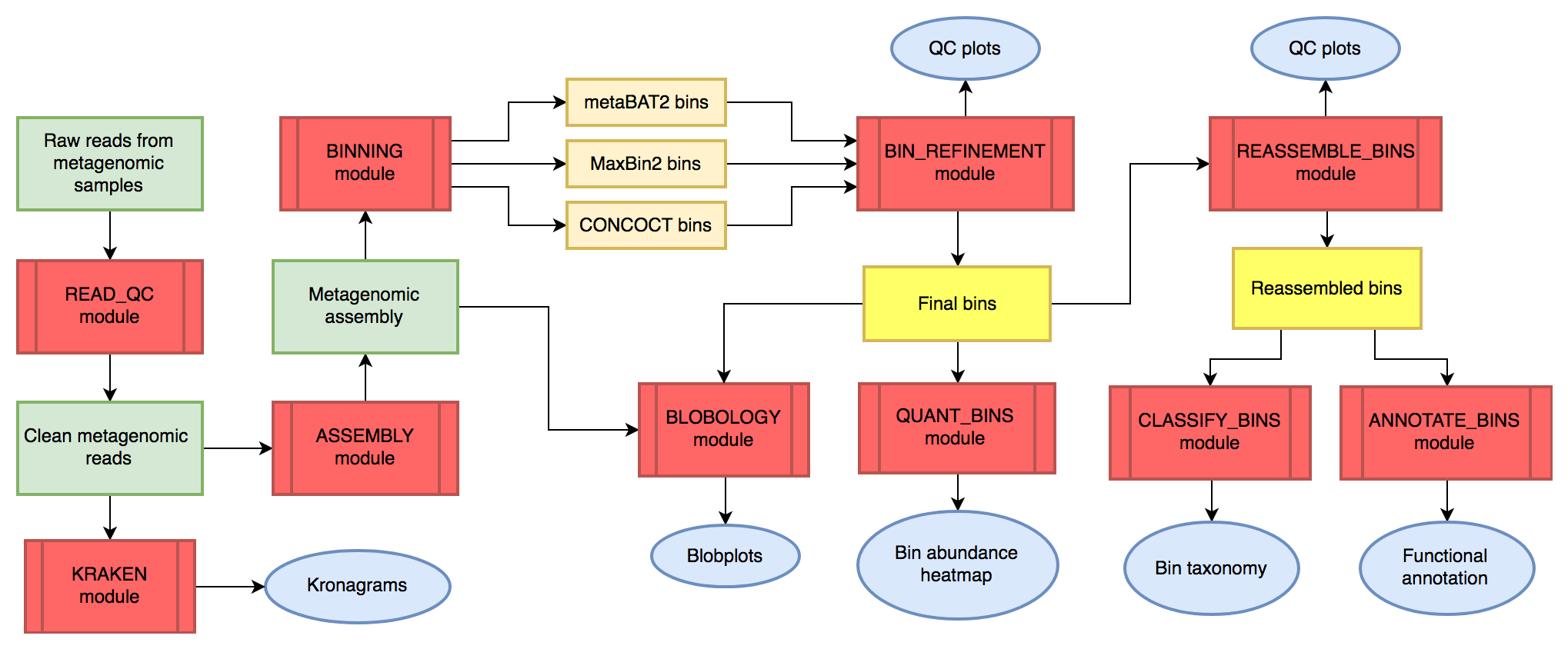
MetaWRAP是一款整合了質(zhì)控、拼接、分箱(Binning)、提純、評(píng)估、物種注釋、豐度估計(jì)、功能注釋和可視化的分析流程,納入超140個(gè)工具軟件,可一鍵安裝。MetaWRAP流程整合了CONCOCT、MaxBin、MetaBAT等三款分箱工具以及提純和重組裝算法,能提供出色的分箱組裝效果,從而有助于從宏基因組數(shù)據(jù)挖掘單菌基因組信息。
https://github.com/bxlab/metaWRAP