2020-08-13
遺傳圖譜又稱連鎖圖譜、遺傳連鎖圖譜。如果說物理圖譜指的是堿基在DNA上的線性排列,那么遺傳圖譜則指基因或DNA分子標記在染色體上的排列順序了,根據選定的基因或分子標記,以及這些標記之間的重組率為“圖距”確定不同多態性標記位點在每條連鎖群上排列的順序和遺傳距離所得到的圖譜就稱為遺傳圖譜。
遺傳圖譜的單位為厘摩(cM),即基因或DNA片段在染色體交換過程中的重組頻率。距離越遠,發生重組的概率越高。
遺傳圖譜主要有四個方向的應用:數量性狀位點(Quantitative trait loci , QTLs)定位、輔助基因組組裝、比較基因組分析、分子標記輔助育種等[1]。遺傳圖譜構通過遺傳重組分析將各類標記根據重組率定位在染色體上。
因此,開發分子標記在遺傳圖譜的構建中是非常重要的環節。傳統的遺傳標記通過表型間接的反映基因的差異,而分子標記以DNA分子的多態性為基礎,能夠更加直接的反映DNA水平的遺傳變異[1]。
隨著分子標記的不斷發展,目前已經有幾十種不同的分子標記技術,這些標記技術各有優缺點,隨著標記技術的不斷發展,目前常用的標記主要有:RFLP (Restriction Fragment Length Polymorphism)即限制性片段長度多態性、RAPD(Random Amplified Polymorphism DNA)即隨機擴增多態性 DNA、 AFLP(Amplified Fragment Length Polymorphism)即擴增片段長度多態性 、 SCAR(Sequence Characterized Amplified Regions)即序列特異擴增區域、CAPs(Cleaved Amplified Polymorohic Sequence)即切斷擴增多態性序列、SSR(Simple Sequence Repeat)即簡單重復序列或微衛星 DNA 標記和SNP(Single Nucleotide Polymorphism)即單個核苷酸多態性標記等[1,2]。
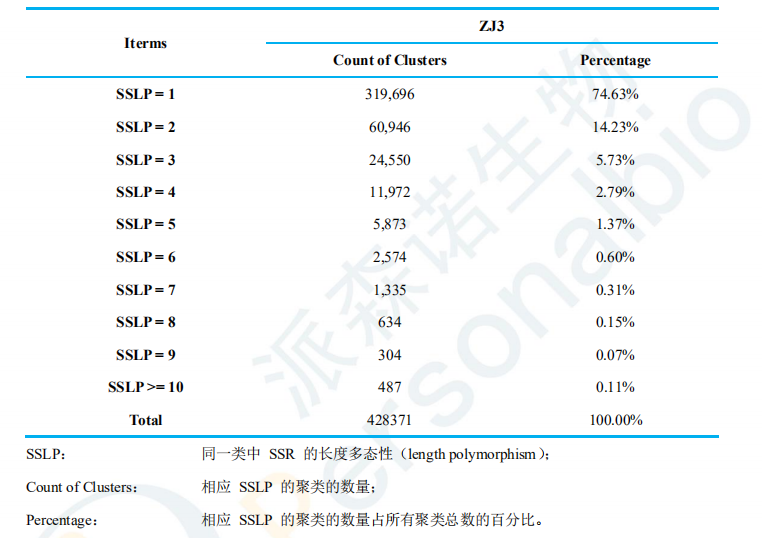
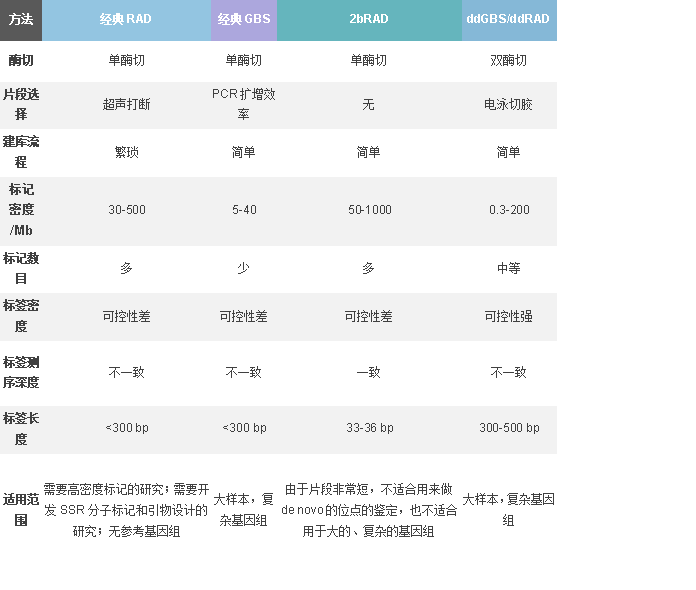
重復堿基類的分子標記 當前,測序技術不斷發展,將高通量測序技術應用于分子標記的開發,能夠更加快速的得到更加豐富的分子標記。將高通量測序技術用于分子標記開發能夠節約大量的時間和成本,常用于開發SSR、SNP等標記方法。 真核生物基因組包含大量由1 - 6 bp的重復 DNA 片段構成的特定序列,長度在1000bp以內,這段序列就被稱為SSR序列[4]。SSR序列的突變率高,且對于不同的等位基因,其重復單元的重復次數差異很大;但其兩端一般為高度保守的單拷貝序列。在DNA修復或復制時,DNA序列發生錯配錯位導致重復序列數目發生變化;在減數分裂、有絲分裂中姐妹染色單體交換的不對等性也會產生SSR序列[5]。我們可以根據其兩端保守序列設計引物擴增,其重復區域拷貝數不同導致最終片段長度也不同,根據分離片段的大小判斷基因型,并計算等位基因發生頻率[6]。 SSR是PCR技術類的分子標記代表,其被廣泛應用與各類物種的遺傳作圖研究中[5,7,8,9]。此外,還有報道SSR在種間也有良好的通用性[10]。 根據SSR的特點,其優點是顯而易見的: 共顯性標記檢,測單一的多等位基因位點; 鑒別雜合子和純合子; 檢測結果重復性好,穩定可靠; 所需 DNA 量少。 早期測序手段欠缺,所以SSR標記的前期開發成本較高,原因是我們必須得到擴增SSR序列兩端的保守序列;而很多時候,序列本身或其旁側序列并不清楚,這極大地限制了該技術的應用[11,12]。 傳統的開發的方法有很多,主要可分為以下三類:利用數據庫/相關文獻、利用近緣物的序列擴增或從gDNA、cDNA、EST中篩選。現在,測序技術不斷發展,我們利用高通量技術,通過構建一個SSR文庫(圖一),一次測序就能得到大量的SSR序列并統計這些序列的多態性(圖二)。通過高通量測序能夠極大地節約開發成本和時間,且有無參考基因組均可,適用性較廣。 圖一 SSR分子標記開發流程 圖二 派森諾SSR分子標記開發結果展示 單堿基的分子標記 單個核苷酸多態性標記(Single Nucleotide Polymorphism,SNP),指DNA上單個核苷酸的變異(如突變、插入和缺失等)導致的DNA序列多態性[13,14]。 SNP位點普遍存在于生物的基因組中,目前已經發展成為最廣泛的標記[15,16]。在不同樣本之前開發SNP標記,目前常用的測序策略就是簡化基因組測序和全基因組測序兩種方案。 以往,出于成本考慮,研究者通常采取對全基因組的一部分進行測序的方案(即簡化基因組測序,Simplified genome sequencing) ,其核心策略就是利用限制性內切酶來得到全基因組的一部分,對該部分測序以達到測相同數據量獲得更高測序深度的目的。目前簡化基因組的測序方案多種多樣,常用的方法主要為RAD(Restriction site Associated DNA)和GBS(Genotyping-By-Sequencing),其余如2b-RAD,dd-RAD,SLAF等方案都是以RAD和GBS為基礎的改良方案,各簡化方案詳細比較如表一所示。RAD和GBS的主要區別在于GBS僅使用限制性內切酶對DNA片段化并測序,而RAD先用限制性內切酶酶切DNA,隨后經行超聲波隨機打斷。目前常用的簡化基因組建庫方案中最常用的就是RAD和dd-RAD方案了,圖二是兩種方案的流程,可以明顯發現RAD方案比dd-RAD方案更加繁瑣,成本更高,但是RAD方案更加適合無參考基因組的物種,所以當我們確定實驗方案時還是要根據需要來選擇合適的方案。 以往很多遺傳圖譜基于簡化基因組測序,遺傳圖譜的飽和度相對不足;另一方面,簡化基因組測序由于其自身特點導致測序結果在全基因組中覆蓋度較低,對后續實驗有一定的影響。相比于簡化基因組測序,全基因組測序可以更快更準確的開發大量適于遺傳圖譜構建的分子標記并構建出高密度的遺傳圖譜。隨著測序成本的大幅下降、對遺傳圖譜的密度要求越來越高,在遺傳圖譜構建方面,全基因組測序已經比簡化基因組測序更具優勢了。 表一 各簡化方案比較 圖二 RAD和dd-RAD方案實驗流程 1.克拉克,顧紅雅,瞿禮嘉.植物分子生物學——實驗手冊[M].北京:高等教育出版社,1998. 2.周延清,楊清香,張改娜.生物遺傳標記與應用[M].化學工業出版社,2008.6 3.徐禮羿. 茶樹SNP高密度遺傳連鎖圖譜構建與數量性狀候選基因挖掘[D]. 2019. 4.Grover A, Aishwarya V, Sharma PC. Searching microsatellites in DNA sequences:approaches used and tools developed. Physiology and Molecular Biology of Plants,2012, 18: 11-19. 5.繆體云. 結球甘藍遺傳圖譜的構建及主要農藝性狀的QTL定位[D]. 中國農業科學院, 2007. 6.羅文永,胡駿,李曉方.微衛星序列及其應用[J].遺傳,2003.25(5):615-619 7.Hearnden PR, Eckermann PJ, Mcmichael GL, Hayden MJ, Eglinton JK, Chalmers KJ.A genetic map of 1,000 SSR and DAr T markers in a wide barley cross. Theoreticaland Applied Genetics, 2007, 115: 383. 8.Isobe SN, Hirakawa H, Sato S, Maeda F, Ishikawa M, Mori T, Yamamoto Y,Shirasawa K, Kimura M, Fukami M. Construction of an integrated high densitysimple sequence repeat linkage map in cultivated strawberry (Fragaria× ananassa)and its applicability. DNA research, 2012, 20: 79-92. 9.陳冬明, 劉長清, 朱連發,等. 基于SSR分子標記的鴨茅指紋圖譜構建及遺傳多樣性分析[J]. 草學, 2020(3). 10.Zhou CP, He XD, Li FG, et al. 2014. Development of 240 novel EST-SSRs in Eucalyptus L'Hérit. Molecular Breeding, 33(1):221-225. 11.車永和.幾種代表性分子標記技術[J].江蘇農業科學,2003,2:3-5.E523 12.曹儀植.植物分子生物學[M].北京:高等教育出版社,2002 13.Sachidanandam R , Weissman D , Schmidt S C , et al. A map of human genome sequence variation containing 1.42 million single nucleotide polymorphisms[J]. 2001, 409(6822):928-933. 14.Mammadov J, Aggarwal R, Buyyarapu R, Kumpatla S. SNP markers and their impact on plant breeding. International journal of plant genomics, 2012, 3: 728398. 15.Rafalski, Antoni J. Novel genetic mapping tools in plants: SNPs and LD-based approaches. Plant Science, 2002, 162: 329-333. 16.Jiang Z, Wang H, Michal JJ, Zhou X, Liu B, Woods LCS, Fuchs RA. Genome wide sampling sequencing for SNP genotyping: methods, challenges and future development. International journal of biological sciences, 2016, 12: 100-108.