2020-07-16
16S rRNA基因是細菌系統分類研究中最常用的分子標記,在進化上具有良好的時鐘性質,其序列包含相互間隔的10個保守區和9個可變區,保守區物種間變化不大,而可變區具有屬或種的特異性。利用高通量測序技術對16S rRNA基因可變區進行測序,能全面解析樣本中的物種組成和對應的豐度信息,因此被廣泛應用于微生物群落研究。
然而需要注意的是,當前基于高通量測序的微生物群落多樣性研究,都是基于相對定量的方法,即把數據歸一化到統一數據量,以每種菌reads數占總reads數的豐度比例來進行量化分析。這種定量方式存在的問題也是顯而易見的,由于16S擴增子文庫的構建有一個無法回避的過程,那就是PCR擴增。只要存在PCR擴增,就會有重復的產生,而擴增偏好性引發的重復的不均勻性,就是相對豐度不可靠的罪魁禍首。也許你會說,我們可以在生信分析端把重復都去掉,然而現實是這些重復的來源不同,代表的意義也不同,一刀切的去重同樣也歪曲了事實,圖一列舉了微生物群落多樣性研究的測序數據中重復的主要來源:
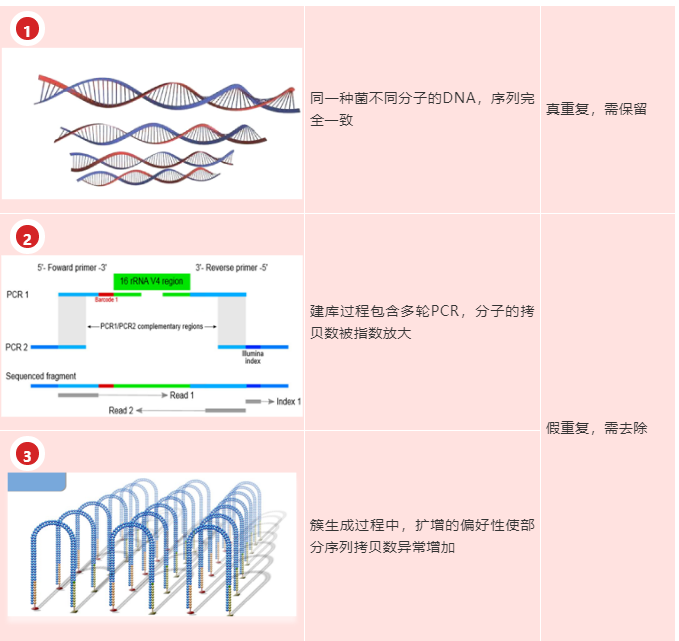
圖一:測序數據中重復的主要來源
①中重復代表的是同種菌的不同分子,是真實物種豐度的呈現,這種重復需要被保留,而②和③這些重復是由擴增產生的假重復,他的數量不能代表PCR擴增前物種的原始組成,尤其當部分序列存在PCR擴增偏好時,Reads數被人為提高,從而導致定量不準,這些重復是需要去除的。
總的來說,PCR擴增之前的重復需要保留,PCR擴增之后的重復需要去除。怎么實現呢?UMI(Unique Molecular Identifier)數字標簽技術這時候就派上用場了,只要在PCR擴增之前給每個分子加上一個特有的標簽,之后無論經過多少個循環的擴增,這個標簽都一直伴隨著同步進行復制,最后可以通過UMI的種類對真重復和假重復進行區分,從而達到去除擴增重復的目的。下圖分別展示了常規兩步法建庫和利用UMI數字標簽建庫的流程:
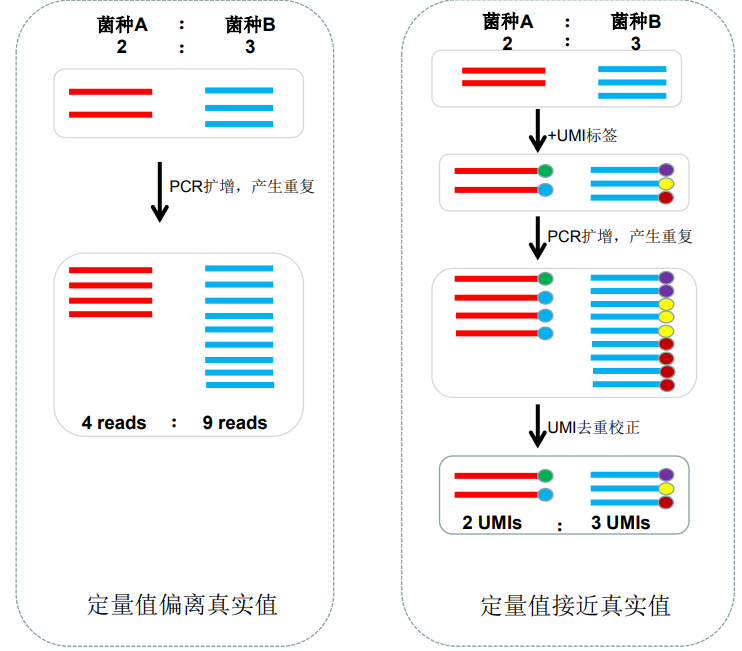
圖二:16S擴增子建庫流程 ,左,常規兩步法建庫流程,右,UMI數字標簽建庫的流程
從上圖不難看出,既往基于測序reads數進行微生物群落多樣性定量,定量值偏離真實值,在采用了UMI數字標簽之后,根據UMI標簽的種類進行定量,定量值更接近于真實值,校正了由PCR擴增偏好帶來的影響。
UMI數字標簽真就如此神奇嗎?適用于哪些微生態場景的研究呢?咱們下期再來分享。


