2020-06-29
群體進化第一期帶大家實戰了基于群體SNP的PCA分析,相信大家都已經練得很熟了,那我們今天帶大家進行實戰進化樹分析,依然含分析和繪圖代碼哦。
系統進化樹(Phylogenetic tree,又稱Evolutionary tree進化樹)是描述群體間分化順序的分支圖或樹,用來表示群體間的進化關系。根據群體的物理或遺傳學特征等方面的共同點或差異可以推斷出它們的親緣關系遠近。
實戰之前我們先熟悉一下進化樹的分類。
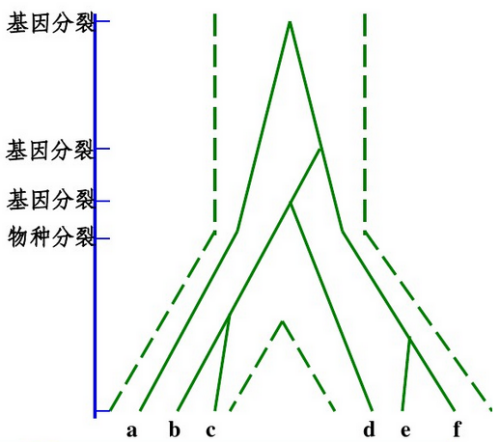
有根樹:是具有方向的樹,包含唯一的節點,將該節點作為樹中所有物種的最近共同祖先。反映時間順序。
無根樹:是沒有方向的,只指明種屬的相互關系,沒有確定共同祖先,線段的兩個演化方向都有可能,樹枝長短表示兩個物種之間進化距離的遠近。反映進化距離。
標度樹:分支的長度代表分化程度。非標度樹:分支只表示進化關系,支長無意義。
物種樹:代表一個物種或群體進化歷史的進化樹,兩個物種分歧的時間即是兩個物種發生生殖隔離的時間。
基因樹:由來自各物種的一個基因構建的進化樹,分歧時間代表基因分離的時間。
舉例來說:對于某一被研究的基因,可能存在種內多態性,即在物種分化之前,該基因可能已經開始分化。兩物種間該基因的分化時間可能早于這兩個物種的分化的時間,根據這個基因計算而來的分支長度(分歧時間)可能偏離。
群體進化分析中的進化樹構建是基于群體SNP的VCF文件進行的,如果樣本數目比較多,VCF文件也會比較大,通常在構樹前都要對原始的VCF文件進行過濾,過濾的標準有:SNP在樣本中的缺失率、質量值、多態位點、深度、次等位基因頻率等。SNP過濾后得到用于構樹的VCF文件,接下來就是進化樹構建的實戰部分了!
以下為實戰數據鏈接:https://pan.baidu.com/s/1flth4p-ORJakQbQqwkXdxQ 提取碼:k6tr
VCF文件格式轉換為fasta格式
下載一個python腳本:(https://github.com/edgardomortiz/vcf2phylip),然后用vcf2phylip.py將VCF文件轉成fasta格式(也可以選擇轉為phylip、nexus格式,如果有外群也可以指定外群),該腳本用法如下:
usage: vcf2phylip.py [-h] -i FILENAME [-m MIN_SAMPLES_LOCUS] [-o OUTGROUP] [-p] [-f] [-n] [-b]
-h, --help show this help message and exit
-i --input 輸入vcf文件,甚至是可以壓縮后的vcf文件
-m --min-samples-locus 缺失的個數,每個位點至少有多少個SNP,默認值為4
-o --outgroup 輸入外群樣本ID,這樣外群樣本會放在第一個。
-p, --phylip-disable 輸出格式為phy
-f, --fasta 輸出格式為fasta
-n, --nexus 輸出格式為nexus
-b, --nexus-binary 輸出格式為二進制
具體使用命令:
./vcf2phylip.py -i test.vcf -f
得到的結果文件test.fasta。
MEGA進行進化樹構建
用于構建進化樹的軟件有很多(比如:MEGA、Phylip、RaxML、MrBayes等),這里我們使用大家常用的MEGA軟件來進行進化樹構建。具體步驟是:打開MEGA軟件,File--導入文件test.fasta--Analysis,Data Type選擇Nucleotide Sequences,然后建樹方法選擇neighbor-joining(NJ),點擊“PHYLOGENY”→“Construct/Test Neighbor”計算模型可以選擇p-distance,最終將構建的樹保存為test.nwk。
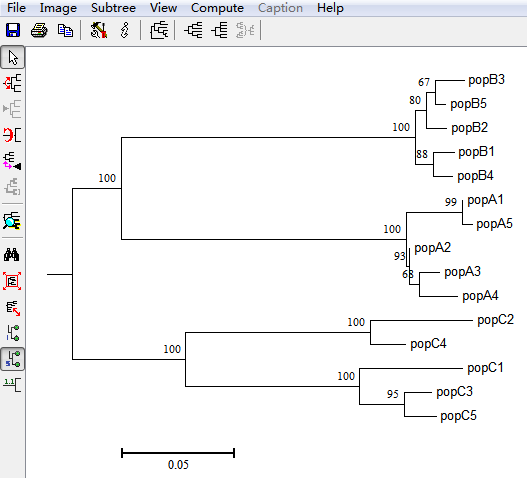
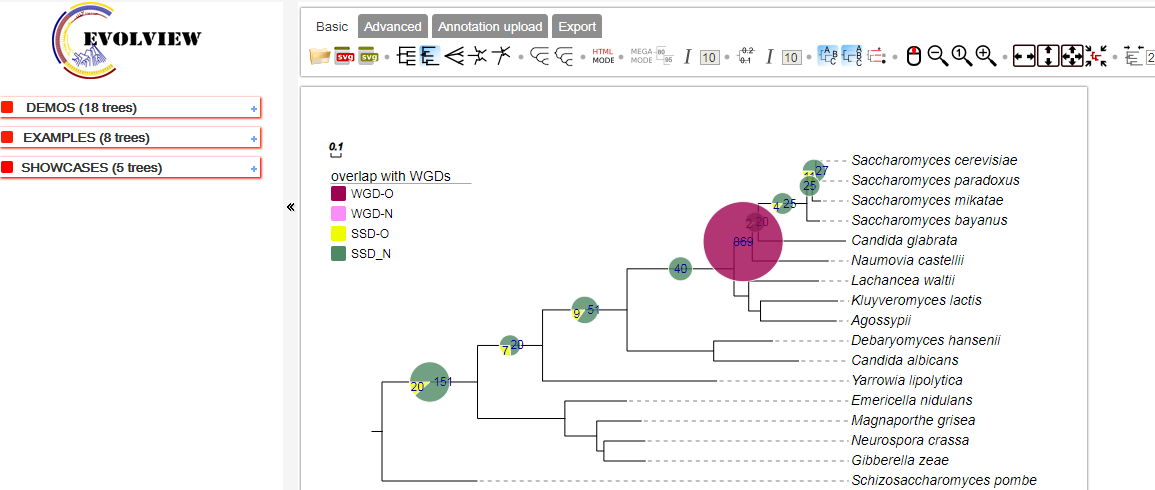
因為MEGA顯示的進化樹不是很美觀,我們需要借助一些軟件來美化一下。這里給大家推薦一款在線軟件Evolview:
(https://evolgenius.info//evolview-v2/)。該軟件不支持IE和360瀏覽器,建議用谷歌打開。下面是登錄注冊后,打開的頁面如下:
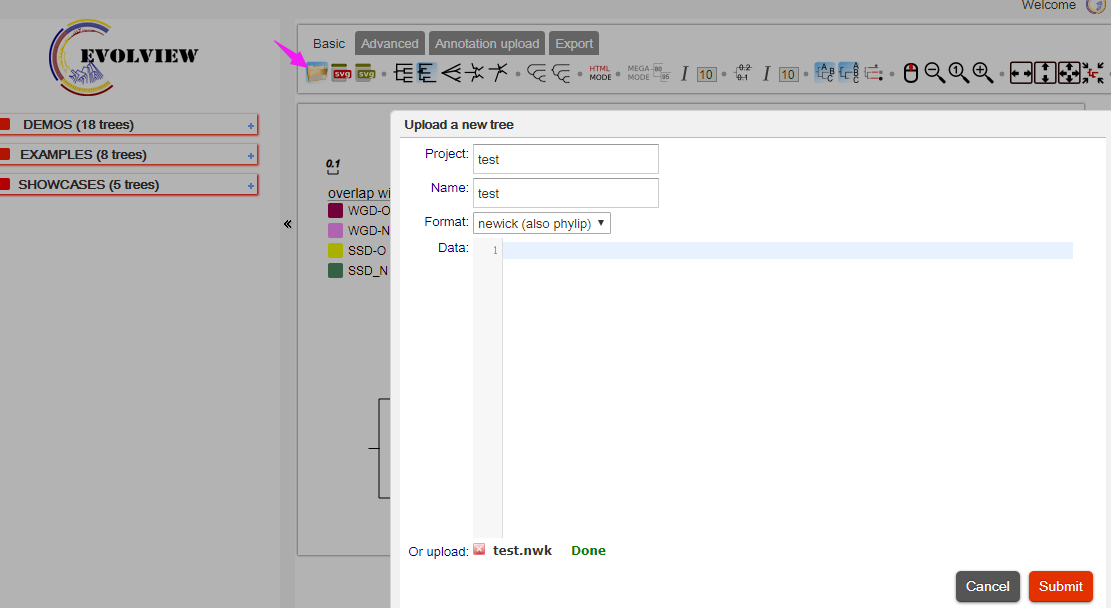
Step1:點擊“Use without an account”→“upload tree file”→“test.nwk”(##點擊下圖箭頭位置,導入進化樹文件,點擊“Submit”上傳樹文件)。
Step2:點擊箭頭所指的位置,修改樹的形狀為圓形。
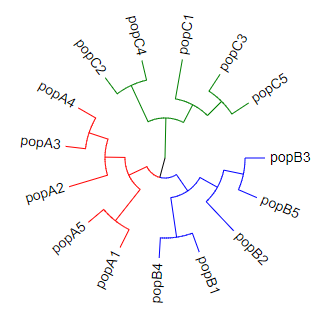
Step3:對連續的樹枝進行上色。點擊“Annotation upload”→“unload data for coloring branches(help)”→“branch.txt”,配置文件branch.txt中列與列之間采用Tab鍵進行分割,具體文件格式如下:

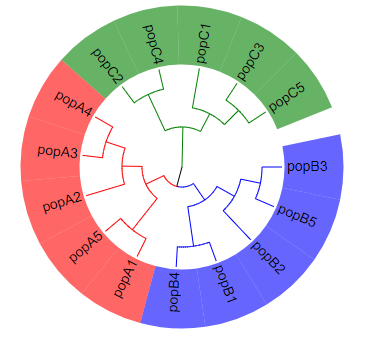
Step4:對連續的樹枝背景進行上色。點擊“Annotation upload”→“unload data for leaf label(help)”→“branch.txt”,為了保持群體分支和背景顏色一致所以依舊用branch.txt。
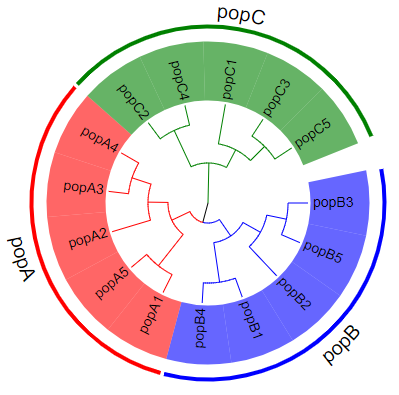
Step5:按照樣本分組指定群體。點擊“Annotation upload”→“unload data for group label(help)”→“group.txt”。
以上就是基于群體SNP進行進化樹分析和圖形美化的方法,如此簡單,趕快用自己的數據試試吧!
如果大家實戰分析的過程中有任何疑問,可在文末留言或者郵件交流([email protected]),歡迎和我們一起探討。下面是動植物產品線的產品類型,如果對其他產品比較感興趣的,也可以郵箱反饋我們,后續我們一一為大家安排。
那下一期《群體進化之群體遺傳結構分析》再見嘍!


