2018-07-25

群體進化:利用全基因組重測序或簡化基因組測序(dd-RAD)技術獲得某物種自然群體各亞群的基因組信息,通過與參考基因組比對或聚類分析的方法得到大量變異信息,基于SNP信息討論群體的遺傳結構、基因交流情況、物種形成機制以及群體進化動態(tài)等生物學問題。
今天小編和大家學習總結一下群體進化的分析流程之群體遺傳結構的結果分析的展示形式。
材料的選取
? 同一物種自然群體的不同亞種或品系;
? 不同地理分布的群體;
? 各亞群間劃分明顯,個體有一定代表性;
? 每個群體至少10個樣本,總樣本量不少于30個
結果分析(群體遺傳結構分析-個體基因組序列差異的的程度)
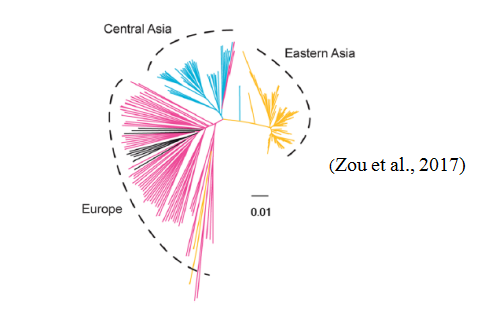
1、系統(tǒng)進化樹
描述群體間分化順序的分支圖或樹,用來表示群體間的進化關系。根據(jù)群體的物理或遺傳學特征等方面的共同點或差異可以推斷出它們的親緣關系遠近。
圖2:擬南芥的系統(tǒng)進化樹分析。系統(tǒng)發(fā)育分析表明,可將擬南芥分為東亞,中亞,歐洲三大類。歐洲材料相比于長江流域和中亞地區(qū)具有更多的群體內SNP,有更高的群體多樣性。黑線表明來自美國、日本和新西蘭的擬南芥。
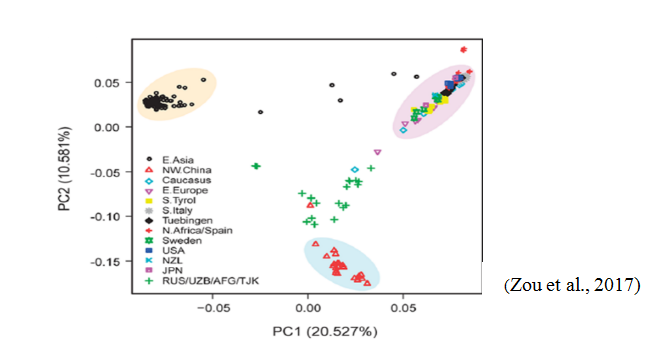
2、主成分分析(PCA)
用較少不相關的變量替代原始大量相關的變量,來研究群體分層,亞群之間的遺傳差異。
圖3:擬南芥樣品的PCA分析。陰影區(qū)域表示來自不同區(qū)域的群體,PCA結果表明可將擬南芥分為東亞,中亞,歐洲三大類。
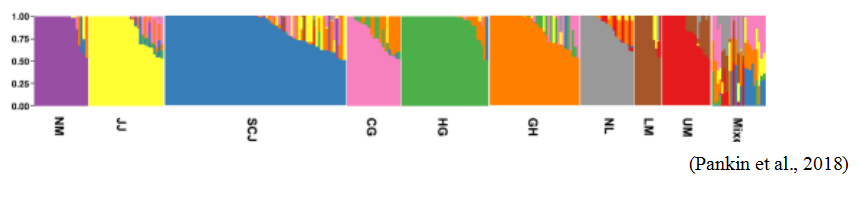
3、群體遺傳結構分析
研究大群體中存在基因頻率不同的亞群,一般可以用來推斷祖先群,個體血緣組成,還有雜交事件。
圖4:大麥的群體結構分析。K=9時,野生大麥基因型的主要成分分析確定了9個群集,群體分為豎條對應于個體的基因型,顏色表示它們在9個子群體中的成員。
主要應用方向
1、人工馴化機制研究
通過對野生型和馴化型群體進行研究,推測群體的親緣關系,發(fā)現(xiàn)人工馴化下受選擇的基因,對動植物育種起到重要的指導作用。
2、自然選擇機制研究
對不同地理環(huán)境下的群體進行研究,挖掘適應性進化過程中受選擇的基因,為育種提供優(yōu)質的基因資源。
3、種群歷史研究?
分析物種的可能起源地及各個分布區(qū)域中群體的基因變異信息,探索物種的進化過程。
群體進化基于群體間的遺傳變異信息
研究群體遺傳結構、遺傳多樣性、物種的形成機制等
從分子層面深入研究該物種的進化歷程
以上常規(guī)分析內容派森諾生物已全部覆蓋!
同時可定制個性化分析項目,為您的文章增添亮點,基于重測序或簡化測序的策略任您選擇~
? 測序平臺:Illumina Hiseq,PE400,2*150bp
? 常規(guī)重測序:>5x/個體
? 低深度重測序結合genotype imputation:≥2×/個體
? 簡化測序dd-RAD:30萬+酶切標簽充分代表遺傳信息
? 平均20×/標簽提升SNP準確度
? 2G/樣品大幅降低成本
? 雙酶切建庫提升數(shù)據(jù)利用率?
還在等什么~
小伙伴們約起來吧~ 更多群體進化小知識,敬請期待~
參考文獻
[1] Pankin A, Altmüller J, Becker C, et al. Targeted resequencing reveals genomic signatures of barley domestication [J]. New Phytologist, 2018, 218(3).
[2] Wu J, Wang Y, Xu J, et al. Diversification and independent domestication of Asian and European pears[J]. Genome Biology, 2018, 19(1):77.
[3] Zou YP, Hou XH, Wu Q, et al. Adaptation of Arabidopsis thaliana to the Yangtze River basin [J]. Genome biology, 2017, 18(1): 239.