2018-07-03
一般做完轉錄組的基本分析,下一步肯定是對分析數據進行挖掘,后期會經常出現各種各樣的聚類圖需求,今天我們就來詳解一種自己DIY聚類圖的方法,快把技能get起來!
首先來看準備清單:
1、兩款分析軟件的安裝
Cluster 3.0 http://bonsai.hgc.jp/~mdehoon/software/cluster/software.htm
TreeView http://jtreeview.sourceforge.net
樣品的差異基因表達量通過 Cluster 3.0 進行基因間和樣品間的聚類分析,得到的聚類結果通過 TreeView 繪制出來。
2、 輸入文件整理
首先對expression.xlsx表格進行整理,原表示例如下:
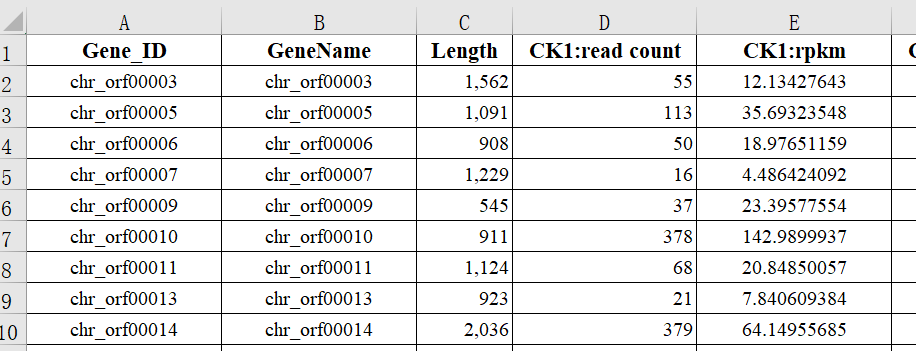
將表格只保留gene-id 和樣本的rpkm列,并將CK1:rpkm改為只有樣品名CK1,示例如下:
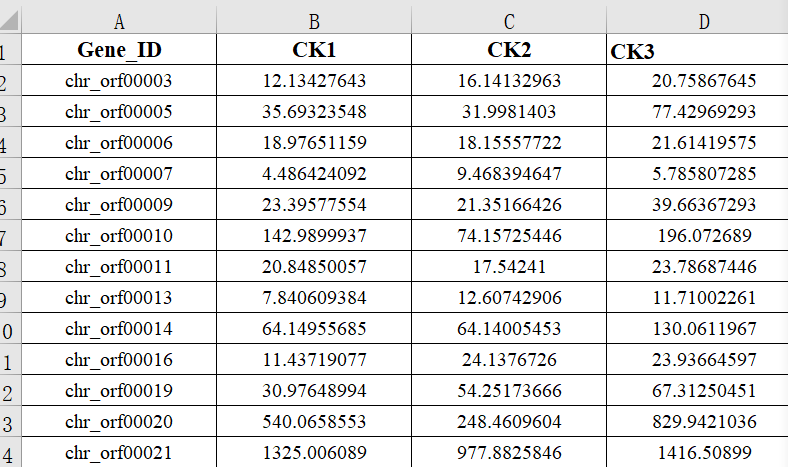
從修改好的表格中篩選需要進行聚類的目的基因,比如挑選9個基因,得到最終表格如下:
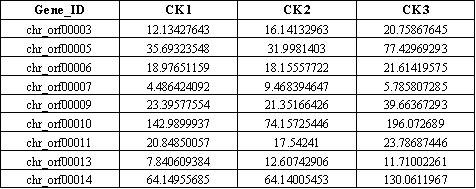
新建一個txt文本文件,將目的表格中的數據粘貼到txt中,比如命名為1.txt,示例如下:
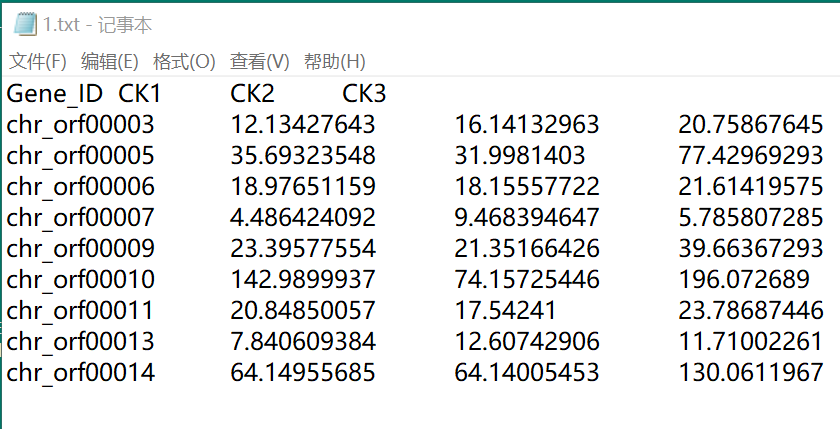
這時輸入文件就整理好啦,下面就進入正經的分析步驟,這時候小本本可以拿出來了:
分析步驟:
Cluster分析
將1.txt文件保存后,導入下載的 Cluster 3.0 軟件中,可以看到中間有個橫條:

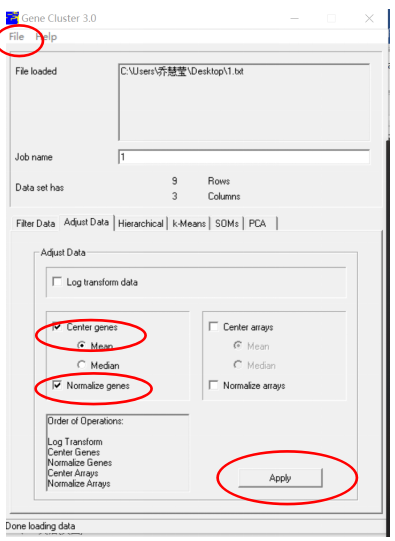
第一個Filter Data點擊后的內容,不做任何選擇,第二個adjust data更改如下,選完后,點擊apply鍵。
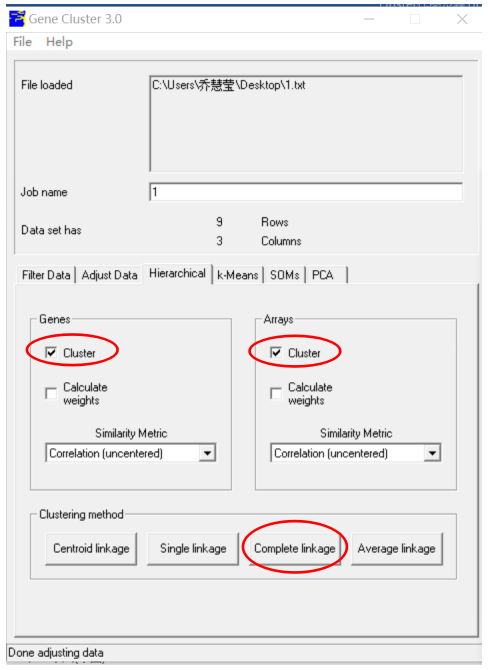
第三個是聚類的方法hierarchical,操作如下,選擇后點擊complete鍵:
這些操作完成后會形成3個文件:
┃ ├───1.cdt 聚類分析文件
┃ ├───1.gtr 聚類分析基因樹文件
┃ └───1.atr 聚類分析樣品樹文件
cdt是用來用TreeView顯示聚類樹圖的,其他文件都可以用文本或者寫字板,或者表格等形式打開,一般比較大的打不開的文件,可以下載editplus軟件來進行打開。
TreeView編輯圖像
打開TreeView,點開file,open打開上一步生成的1.cdt
結果顯示如下:
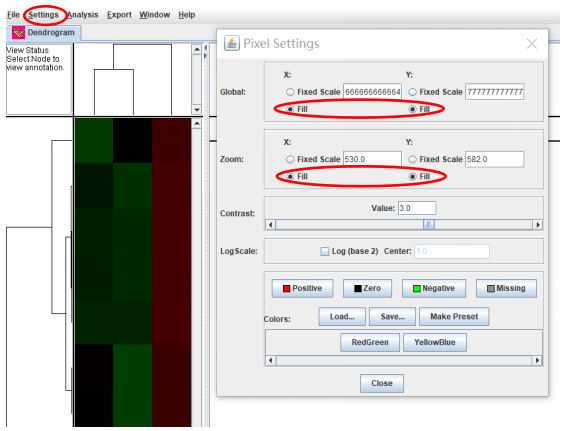
打開最上面工具條里的settings,選擇Pixel settings,設置如下:
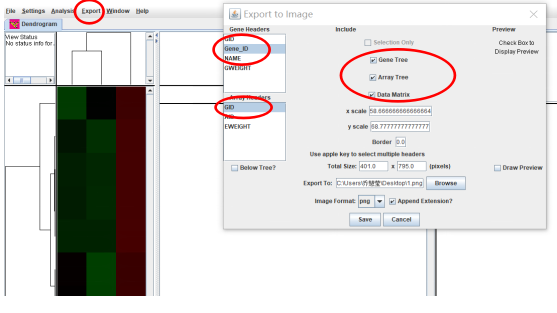
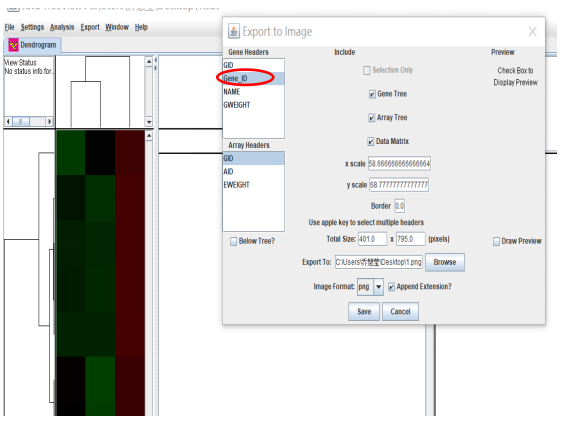
設置好后,點close,然后點開工具條中的Export, 選擇save tree image,設置如下:
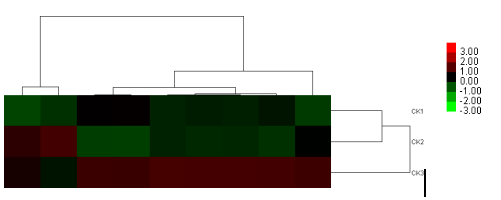
點擊save之后會保存圖片,結果展示如下,右側是樣本名,下方是基因名:
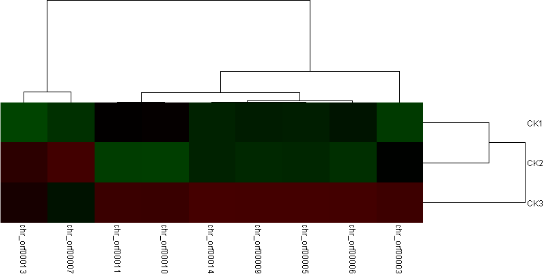
如果只展示樣本名,再上一步export的設置中,如圖所示,gene headers 中的gene -id不選擇:
然后save結果就會只顯示樣品名:

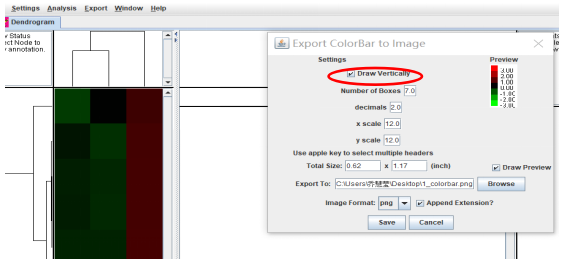
最后點開Export,選擇export Colorbar to image,設置如下:
點擊save之后會出現Colorbar圖片,整合圖片后,即為聚類結果
心動不如行動,需要聚類圖的你趕緊DIY起來吧!
文案:轉錄調控事業部