2018-06-08
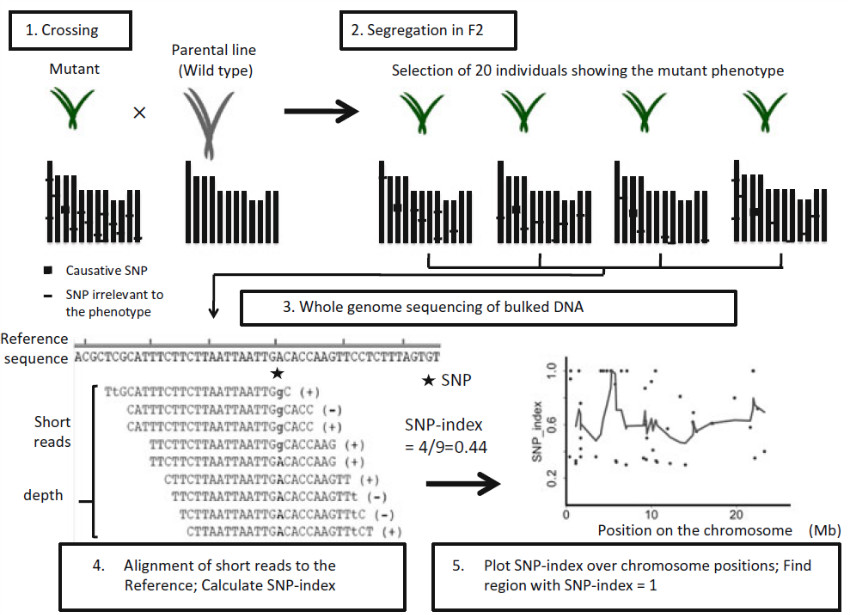
MutMap方法作為一種特殊的BSA方法,其主要思路是:用化學誘變劑EMS處理得到突變體,然后用突變體植株與野生型親本雜交得到F2代,從F2代中挑選突變表型的個體進行混池測序。定位的方法是將測序得到的reads比對到親本基因組上,計算SNP-index。突變體子代是所研究性狀突變位點的純合個體,因此SNP-index = 1。而其他與性狀無關的SNP位點,SNP-index ≈ 0.5。而與突變位點鄰近的SNP位點,由于連鎖效應,SNP-index也會增加,即>0.5 ,從SNP-index圖上看則會呈現一個峰,性狀相關候選基因就位于該區域內。
對于上述mutmap方法基因定位過程,常見路線是對野生型親本和突變F2子代混池進行測序。實際項目中,某些情況下,我們會推薦測野生型子代混池。那么對于野生型F2子代混池是否有必要測序,加測野生型子代對定位結果影響如何呢?下面我們分別從理論上和實際的項目結果來說明這個問題:
首先,對于EMS誘變的植株,SNP包含兩類,普通SNP和誘變產生的SNP,以玉米(基因組2G)為例,兩類突變的頻率和數量分別為:
* 普通SNP: 突變頻率 1x10-3 數量2M SNP
 EMS誘變: 突變頻率 5x10-6 數量 10k SNP
EMS誘變: 突變頻率 5x10-6 數量 10k SNP
可以看出,普通SNP比EMS誘變產生的SNP數目要多得多,即使普通SNP有1%的假陽性或假陰性,也會有20k的SNP,數目也是EMS誘變SNP數的兩倍。因此,對于mutmap類項目,這些普通的SNP作為與性狀無關的背景噪音,對定位結果的影響會更大,我們期望的信號位點可能會被背景噪音所淹沒。
那么,我們如何降低背景噪音的影響呢?除了通過我們常規的過濾手段,保證獲得的SNP的準確性和可靠性之外,可以加測與突變體遺傳背景相同的野生型子代混池的手段。

下面我們通過實例對于是否測野生子代池的結果做一個比較。
研究物種:某植物
測序平臺:Illumina HiSeq
測序策略:野生親本20x ; 突變子代混池30x ; 野生型子代混池 50x
混池樣本數:100株
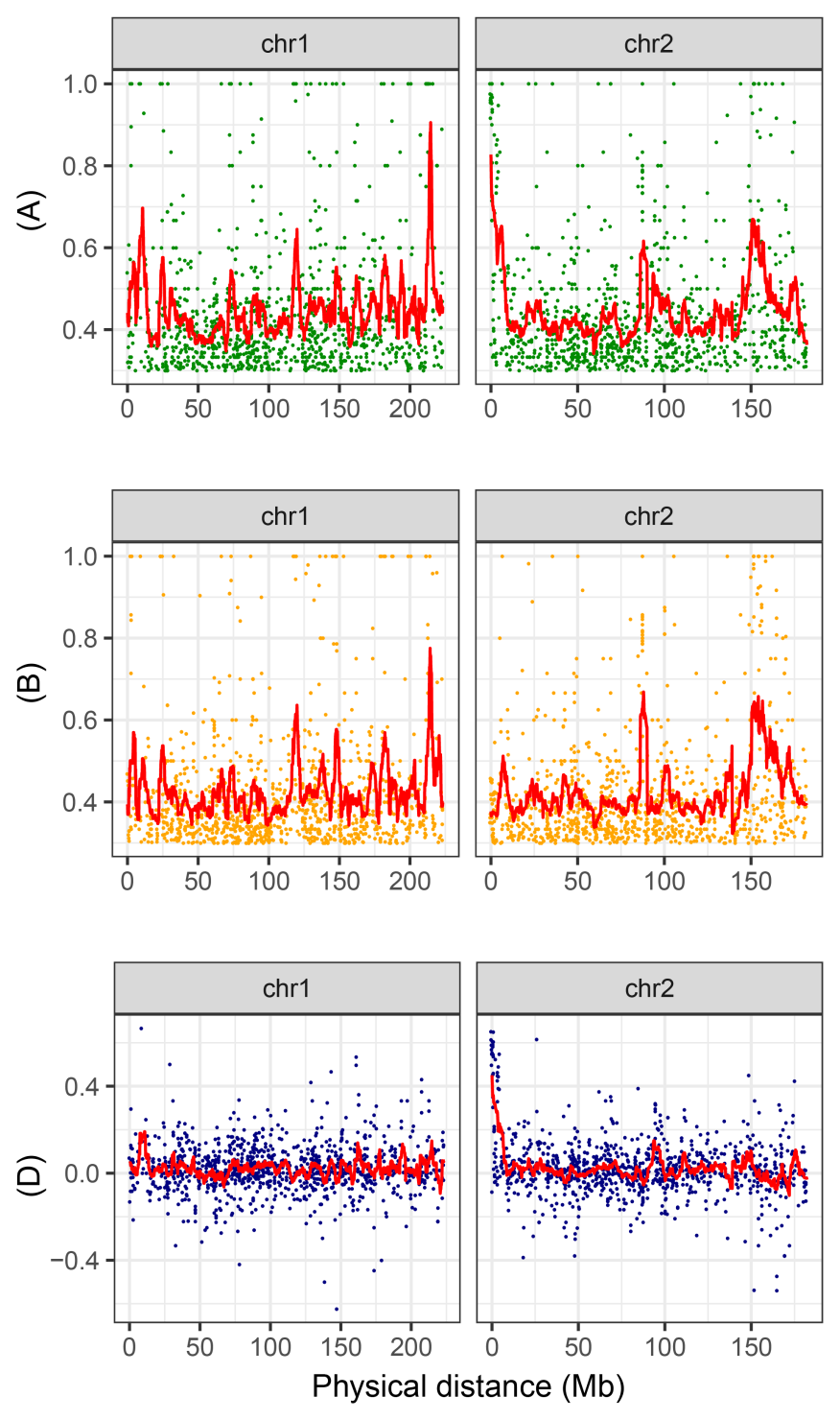
(A圖為突變子代混池SNPindex,B圖為野生型子代混池SNPindex,D圖為Delta-SNPindex)
我們以1號和2號染色體為例,若只測野生型親本和突變子代混池,則我們只得到突變子代池的SNPindex圖,即圖A,從圖中我們可以看出有多個峰,均會作為候選區域做后續分析。
若加測野生型子代池,則會得到野生型子代池的SNPindex,即B圖,兩者相減,可以得到Delta-SNPindex,即D圖。從結果可以看到,多個噪音峰值被消除,最終位于2號染色體頭部的候選區域會被篩選出來。
所以,對于mutmap項目,從分析角度講,只測野生親本和子代突變混池是可以的,但會包含假陽性結果,具體影響程度與不同物種及測序情況有關。若加測野生型子代混池會改善定位結果。對于基因組比較大、比較復雜的物種,推薦大家加測野生型突變混池。


