
2016-11-01
很多時候,我們更希望知道菌群行使的具體功能,也就是解釋“它們在干什么?”。對菌群進行宏基因組測序自然是解決方案,但如此高大上的研究方法,投入成本相對較多,分析方法也相對復雜。如果僅僅知道菌群組成,該怎么把物種的“身份”和它的“功能”對應起來呢?
由此,一款名為PICRUSt的菌群代謝功能預測利器應運而生,相關論文發表在權威期刊《Nature Biotechnology》上[1]。PICRUSt全稱為“Phylogenetic Investigation of Communities by Reconstruction of Unobserved States”,可以通過16S rRNA基因序列,預測對應的細菌和古菌的代謝功能譜。
有了PICRUSt,相當于在菌群的“組成”和“功能”之間搭起了一座橋,即使只有16S rRNA基因的測序結果,我們一樣可以獲得宏基因組級別的研究結果!

1 PICRUSt的基本原理
PICRUSt的總體思路說來很簡單,主要分為3步:
1. 先根據已測微生物基因組的16S rRNA基因全長序列,推斷它們的共同祖先的基因功能譜;
2. 對Greengenes數據庫中其它未測物種的基因功能譜進行推斷,構建古菌和細菌域全譜系的基因功能預測譜;
3. 最后,將測序得到的菌群組成“映射”到數據庫中,對菌群代謝功能進行預測。該步驟具體通過以下方式實現:
i. 對測序獲得的16S rRNA基因序列,進行“封閉式”參考OTU劃分(Closed-reference OTU picking),通過與Greengenes數據庫比對,尋找每一條測序序列的“參考序列最近鄰居”,并歸為參考OTU;
ii. 根據“參考序列最近鄰居”的rRNA基因拷貝數,對獲得的OTU豐度矩陣進行校正;
iii. 根據“參考序列最近鄰居”對應的KEGG/EggNOG等基因功能譜數據,換算預測菌群的整體代謝功能。
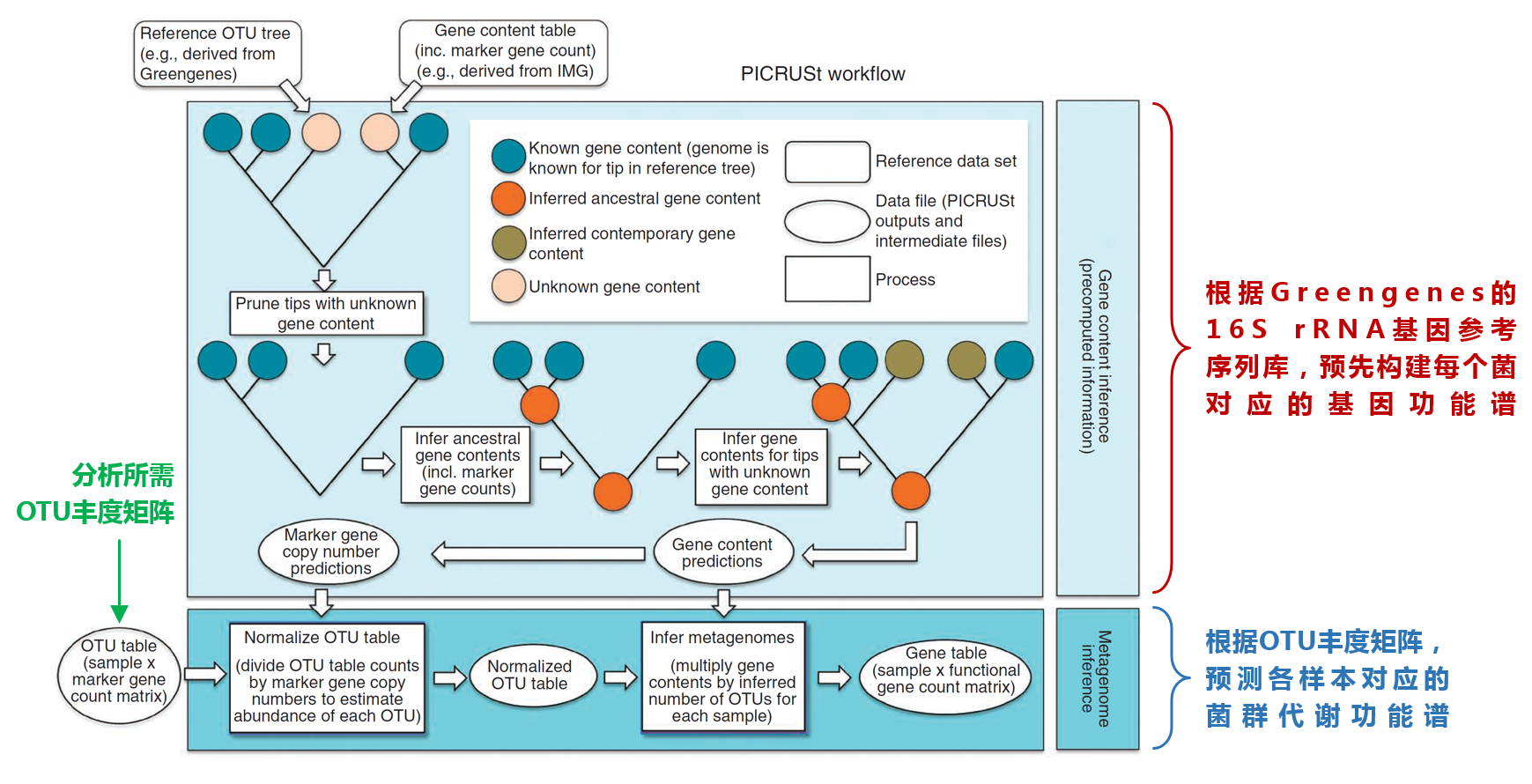
PICRUSt分析的基本流程圖示【1】
2 PICRUSt的特點
PICRUSt算法的一大特點,就是基于Greengenes的16S rRNA基因全長序列數據庫,對菌群測序結果進行“封閉式”參考OTU劃分。PICRUSt開發者對古菌和細菌域的大多數模式微生物的功能進行預測,讓人欣喜的是,絕大多數的微生物預測結果與真實的基因功能譜非常接近(古菌預測精確度為0.94 ± 0.04,n = 103;細菌為0.95 ± 0.05,n = 2487)。也就是說,絕大多數情況下,PICRUSt的預測效果還是相當靠譜滴!
PICRUSt對細菌/古菌基因組預測的精確度
當然,若測序序列與Greengenes數據庫中沒有同源物種的參考序列,則對應的物種將無法被預測。也就是說,原始數據在分析過程中會有一定損失。此外,PICRUSt的特點也表明,它只能對已知微生物的已知功能進行功能預測,所以目前并不能完全代替宏基因組研究,但可以看作宏基因組研究的“近似”結果,在經費、資源有限的情況下,不失為一種理想的選擇。
3 PICRUSt的應用
在小編看來,PICRUSt對于菌群研究的貢獻大大滴!首先,PICRUSt能從菌群組成數據解讀潛在的功能,可謂充分發揮了16S rRNA基因測序簡單、快速、物美價廉的優勢;其次,PICRUSt對菌群功能的預測,可以幫助指導后續宏基因組De novo鳥槍法測序的實驗設計,更合理地篩選用于后續研究的樣本。
同時,PICRUSt分析的操作相當便捷,只需對測序數據進行“封閉式”參考OTU劃分,并將得到的OTU豐度矩陣上傳至Galaxy在線分析平臺(http://huttenhower.sph.harvard.edu/galaxy/root?tool_id=PICRUSt_normalize),按提示操作就能得到預測結果啦!
PICRUSt在線分析平臺
4 16S+PICRUSt,研究新趨勢
PICRUSt這一方法橫空出世后,已有越來越多的研究開始采用這一方法預測菌群組成數據,相關論文發表數量逐年增長,表明該方法已得到大家廣泛的認可。16S+PICRUSt,已成為發表高水平論文的技能;同時與宏基因組分析相比,更方便快捷,成本也更低,何樂而不為!
采用PICRUSt方法的菌群研究文獻逐年增長
比如,今年發表在微生態學旗艦期刊《The ISME Journal》(影響因子9.328)的一篇論文《Cigarette smoking and the oral microbiome in a large study of American adults》[2]中,作者就通過PICRUSt預測了吸煙人群和未吸煙人群的口腔菌群,發現共有83個基因功能代謝通路存在顯著差異,吸煙大大降低了碳水化合物和能量代謝、異型生物質降解等代謝通路的含量。

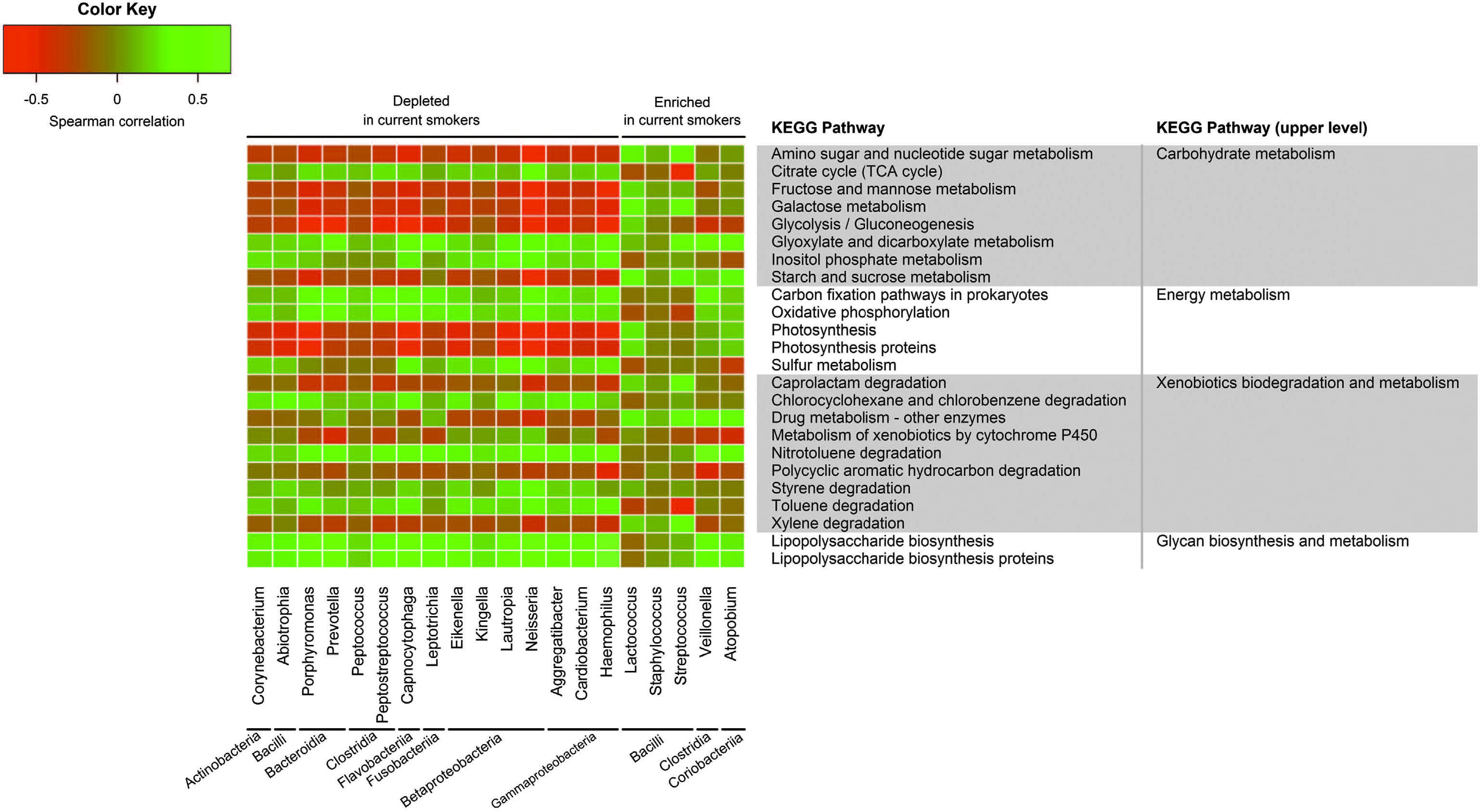
PICRUSt對吸煙人群口腔菌群的功能預測[2]
結語
經過上面的講解,大家是不是感受到了PICRUSt預測菌群功能的強大威力了呢?小編在這里想說,PICRUSt雖然厲害,但并不是萬能,一方面由于它是基于16S rRNA基因的參考序列庫,因此尚無法對真菌群落進行功能預測,而預測過程也會造成原始數據的部分損失,對于不同來源的菌群的預測效果也有所差異(一般對于人源微生物組的預測效果最好);同時,它并不能完全代替宏基因組研究(目前只能對已知微生物的已知功能進行功能預測),但可以對后續實驗設計作出指導。
參考文獻
1. Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, et al. (2013) Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature Biotechnology 31: 814-+.
2. Wu J, Peters BA, Dominianni C, Zhang Y, Pei Z, et al. (2016) Cigarette smoking and the oral microbiome in a large study of American adults. ISME J 10.1038/ismej.2016.37.