2017-01-04
引言
派森諾生物于2016年1月底與美國加利福尼亞太平洋生物科學公司(Pacific Biosciences of California,Inc., PacBio)合作,購入第三代單分子基因組測序系統-Sequel。經過將近 5 個半月的等待,2016年6月7日,華中地區首臺 PacBio Sequel 遠渡重洋正式入駐派森諾生物(該平臺也是當時全中國唯一一臺硬件經過最新升級的 Sequel 平臺)。作為一家商業化運作的公司,派森諾生物早在 2015 年初就開始積極布局基于第三代單分子基因組測序的產品開發,先后推出細菌全基因組完成圖、真菌基因組近完成圖及“白金級”動植物基因組等成熟產品。Sequel 平臺入駐公司后,派森諾生物的技術團隊緊鑼密鼓、有條不紊地對該平臺進行調試、優化,在經歷了最初平臺運行不穩定、測序試劑版本的更新迭代后,目前,派森諾生物的 Sequel 平臺已經進入平穩的商業化運作階段。限于篇幅,我們以眾多案例中的一個來展示 Sequel 平臺的運行效果。
樣本背景信息
樣本為地衣芽胞桿菌,革蘭氏陽性細菌。該菌能調節腸道內微生物之間、微生物與宿主之間處于微生態平衡,同時參與腸道內物質代謝,增強免疫力和抗應激能力。基于該菌開發的制劑主要用于細菌原因引起的腸道菌群失調癥以及腸道需要保健的養殖動物。地衣芽胞桿菌的基因組大小在 3.94 Mb~4.76 Mb,GC 含量為 45.00%~46.30%。
樣本建庫和測序信息
將 DNA 進行片段化后,我們構建了標準的 10 kb 的文庫,采用 PacBio Sequel 平臺進行測序,上機采用的試劑為 Sequel 平臺最新的試劑版本 V1.2.1,上機模式為 Standard 模式,上機測序時間為 360 min。數據產出統計結果見表1,subreads 的長度分布見圖1。
表 1 數據產出統計
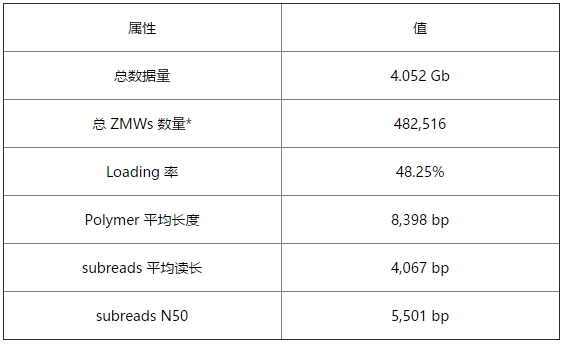
Subreads: 指過濾掉接頭、片段長度 > 200 bp 的序列。ZMWs 數量*: 有數據產出的 ZMWs 孔的數量;
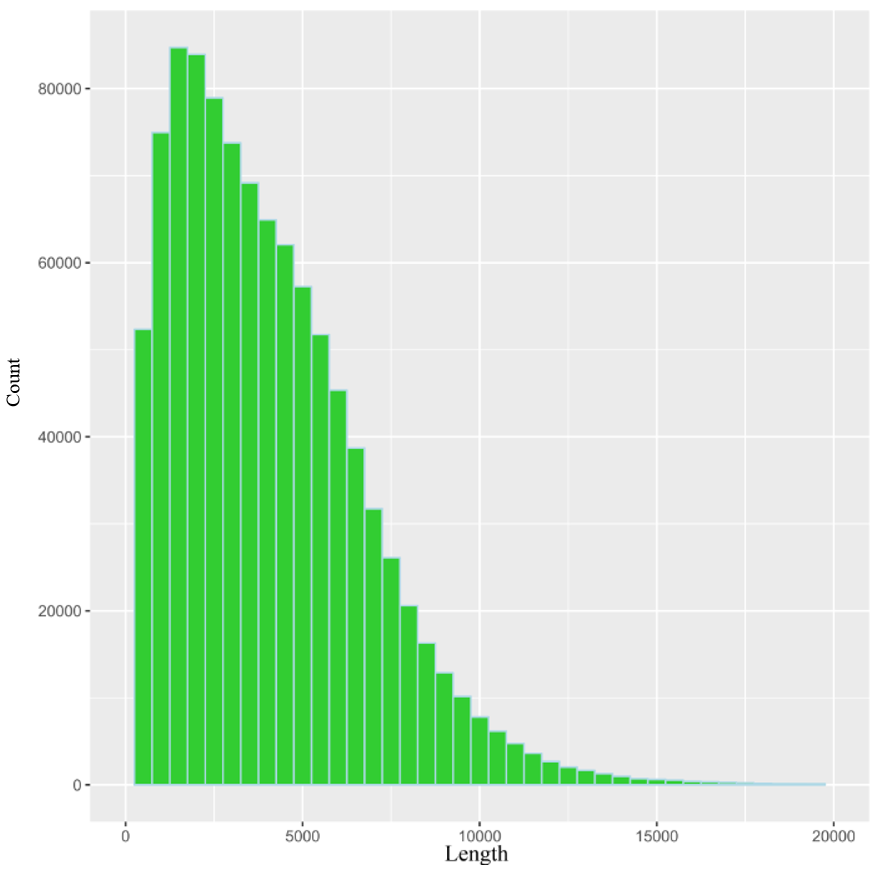
圖 1 subreads 長度分布圖
基因組拼接
采用 falcon 軟件對 Sequel 測序得到的數據進行拼接,總共拼接得到一條完整的序列;接著,利用三代測序 reads 對拼接得到的序列成功環化;隨后,基于二代測序,對拼接的序列進行校正;最后,根據 GC skew 的結果對基因組的起始位點進行調整。最終的基因組大小為 4.352 Mb,基因組的 GC 含量在 45.94%,基因組圈圖見圖 2。
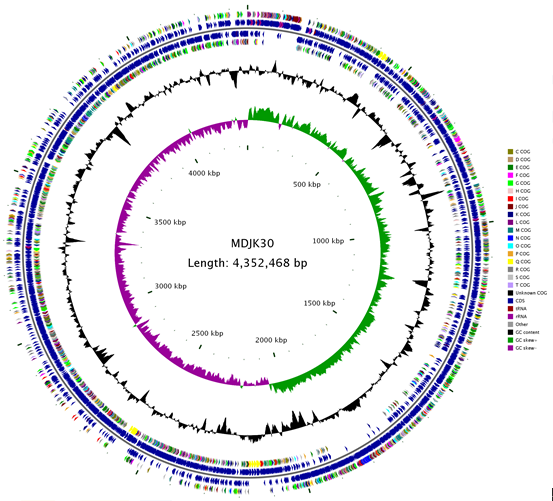
圖 2 基因組圈圖
小結:
(1) 該案例僅僅是派森諾生物 Sequel 平臺商業化運行中的一個成功案例之一;
(2) 除細菌基因組外,目前,公司已有真菌及動、植物的樣品在 Sequel 平臺進行測序,公司也會在基因組拼接結束后,及時地發布這些信息,供廣大科研工作者參考;
(3) 目前,很多公司都拿到了 Sequel 平臺的測試數據,但真正能平穩運行 Sequel 平臺且具有豐富經驗的公司寥寥無幾;
(4) 派森諾生物除了在 Sequel 平臺的測序和拼接方面具有豐富的經驗,在基因組的信息分析及深度的數據挖掘方面也有著豐富的經驗,能協助老師發表高水平的科研論文。


