全長轉(zhuǎn)錄組測序要點(diǎn)匯編IV ——可變剪切分析篇
2017-03-15
以PacBio公司的SMRT單分子實(shí)時(shí)測序技術(shù)(Single molecule real-time sequencing)為代表的三代測序技術(shù),通過其獨(dú)有的環(huán)形一致性測序模式(Circular-consensus sequence,CCS),極大提高單堿基測序的準(zhǔn)確率,遠(yuǎn)超Illumina等二代測序技術(shù)。與傳統(tǒng)轉(zhuǎn)錄組測序項(xiàng)目相比,利用PacBio平臺(tái)的全長轉(zhuǎn)錄組測序技術(shù)可以直接獲得mRNA的全長,保證了mRNA序列的精確性。近期我們將陸續(xù)推出全長轉(zhuǎn)錄組測序技術(shù)相關(guān)文章,供討論和交流。我們在上一期向大家介紹了全長轉(zhuǎn)錄組測序的分析內(nèi)容,本期將為大家對派森諾特色分析內(nèi)容——可變剪切模型可視化進(jìn)行重點(diǎn)講解。
可變剪切模型可視化
可變剪切是造成基因信息多樣化的一個(gè)重要原因,通過對RNA序列測序可以檢測到基因的不同可變剪切模式。今天為大家介紹一種可以將可變剪切模型繪制成圖片的Python軟件SpliceGrapher。
SpliceGrapher可以根據(jù)測序序列預(yù)測可變剪切模型,也可以用已知的基因注釋文件生成可變剪切模型。它需要兩種輸入文件,注釋gtf/gff3文件和測序reads與參考基因組比對的sam文件,如果有EST序列,也可以用上。
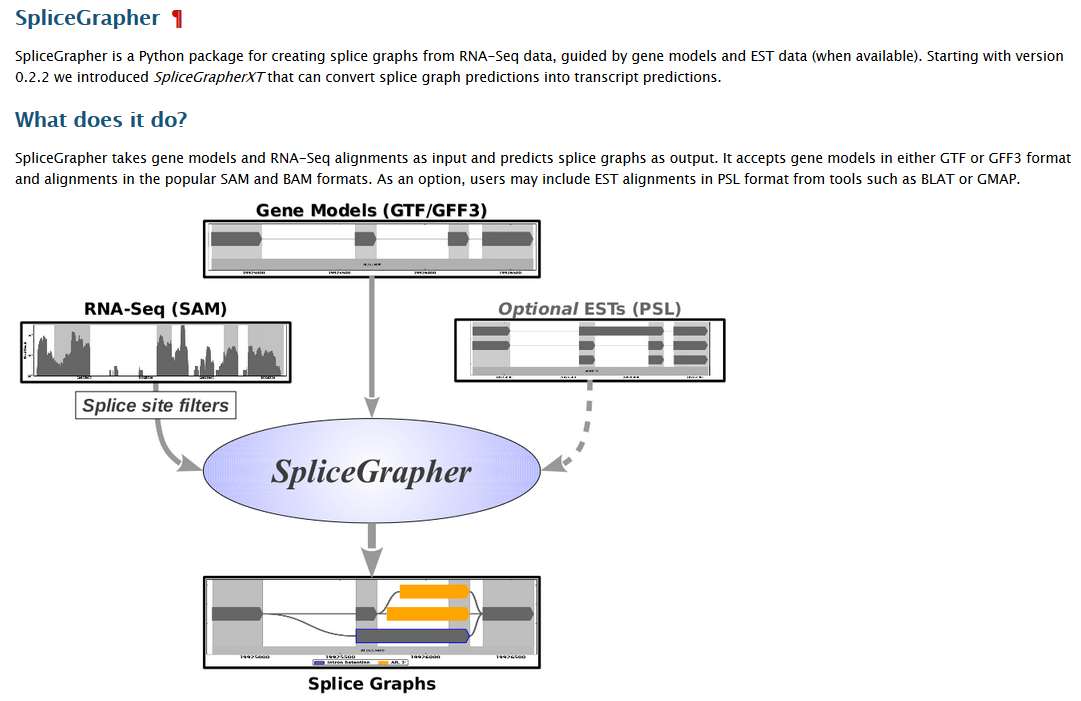
繪制可變剪切模型
SpliceGrapher的使用過程非常簡單。首先需要去掉sam文件中的假陽性可變剪切。這里需要用上SpliceGrapher專門準(zhǔn)備的物種特性篩選文件,在SpliceGrapher文件夾下有上百種物種的特性篩選文件。然后用篩選后的sam文件預(yù)測可變剪切模型,用注釋gtf/gff3文件產(chǎn)生已知的可變剪切模型。最后用SpliceGrapher的繪圖腳本將可變剪切模型畫成圖片。
可變剪切圖解讀
結(jié)果圖蘊(yùn)含了許多信息,下面就為大家介紹一下。
一個(gè)圖片中僅有一個(gè)基因的模型,左下角和右下角的數(shù)字分別代表該基因的起始位點(diǎn)和終止位點(diǎn)。圖片一共分4行,第一行為以該基因的注釋文件作出的基因模型,第二行為根據(jù)測序結(jié)果與注釋文件共同作出的基因模型,第三行也是根據(jù)測序結(jié)果與注釋文件共同作出的基因模型,與第二行不同的是只是畫出具有代表性的isoform,第四行則是測序文件中支持各外顯子的reads數(shù)目。
灰色的五邊形代表外顯子,它們之間的連線表示不同的剪接方式。紫色背景表示有外顯子出現(xiàn)的區(qū)域(這其中包括保留的內(nèi)含子),白色背景表示沒有外顯子出現(xiàn)的區(qū)域(即內(nèi)含子區(qū)域)。圖中白色背景的寬度并不代表真實(shí)的內(nèi)含子長度,由于有的基因內(nèi)含子區(qū)域遠(yuǎn)比外顯子區(qū)域長,為了更清楚地展示可變剪切模型,內(nèi)含子區(qū)域會(huì)被縮短很多。
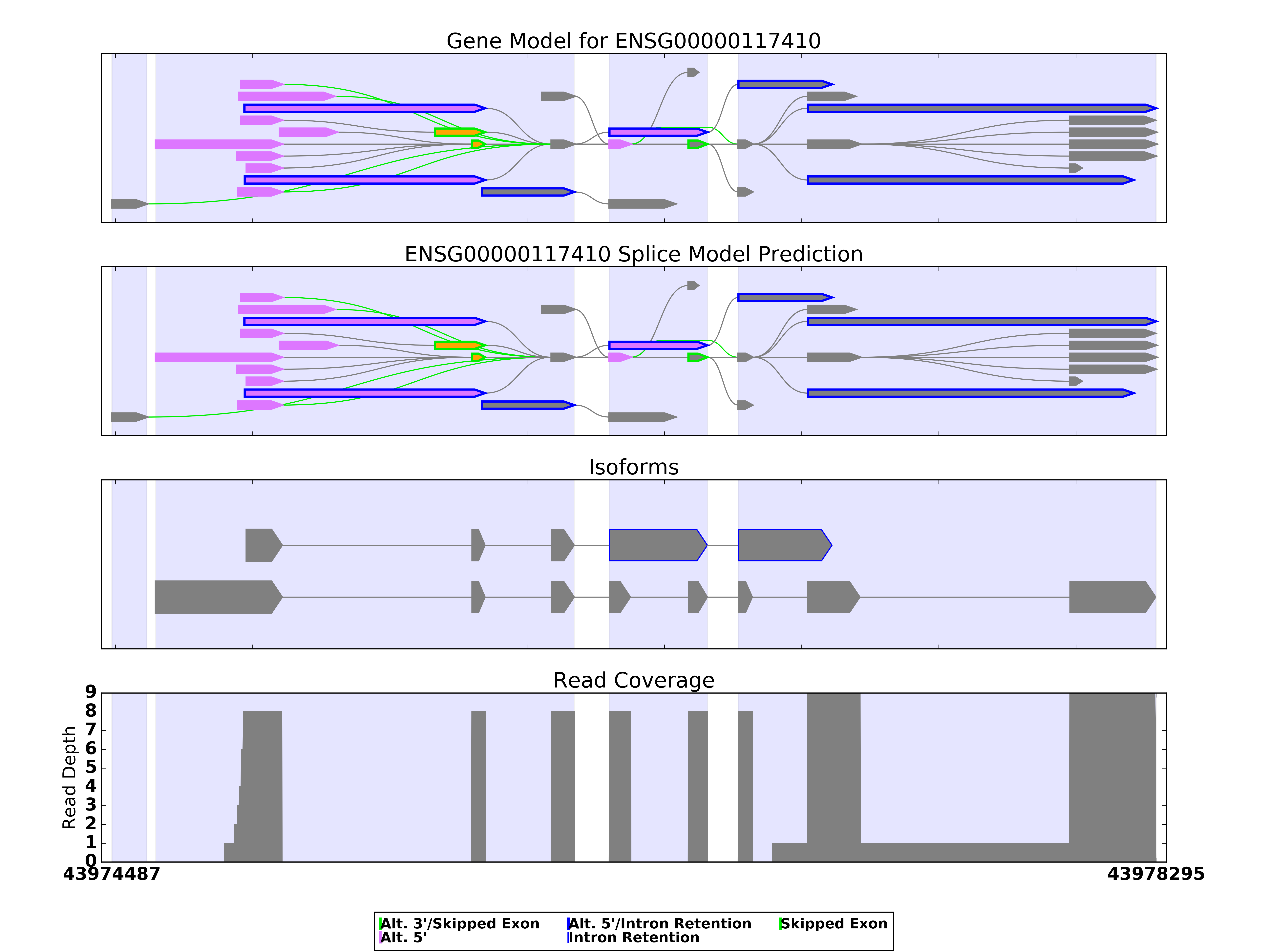
SpliceGrapher可以預(yù)測出多種多樣的可變剪切事件,已知的基因模型中存在Alt 3’(可變3’端)、Skipped Exon(跳過外顯子)、Intron Retention(內(nèi)含子保留)、Alt 5’(可變5’端)、既是Alt 3’又是Skipped Exon、既是Alt 5’又是Intron Retention等多種可變剪切事件。不同的事件用不同的顏色標(biāo)注出來,并在圖片下方的圖例中給予解釋。
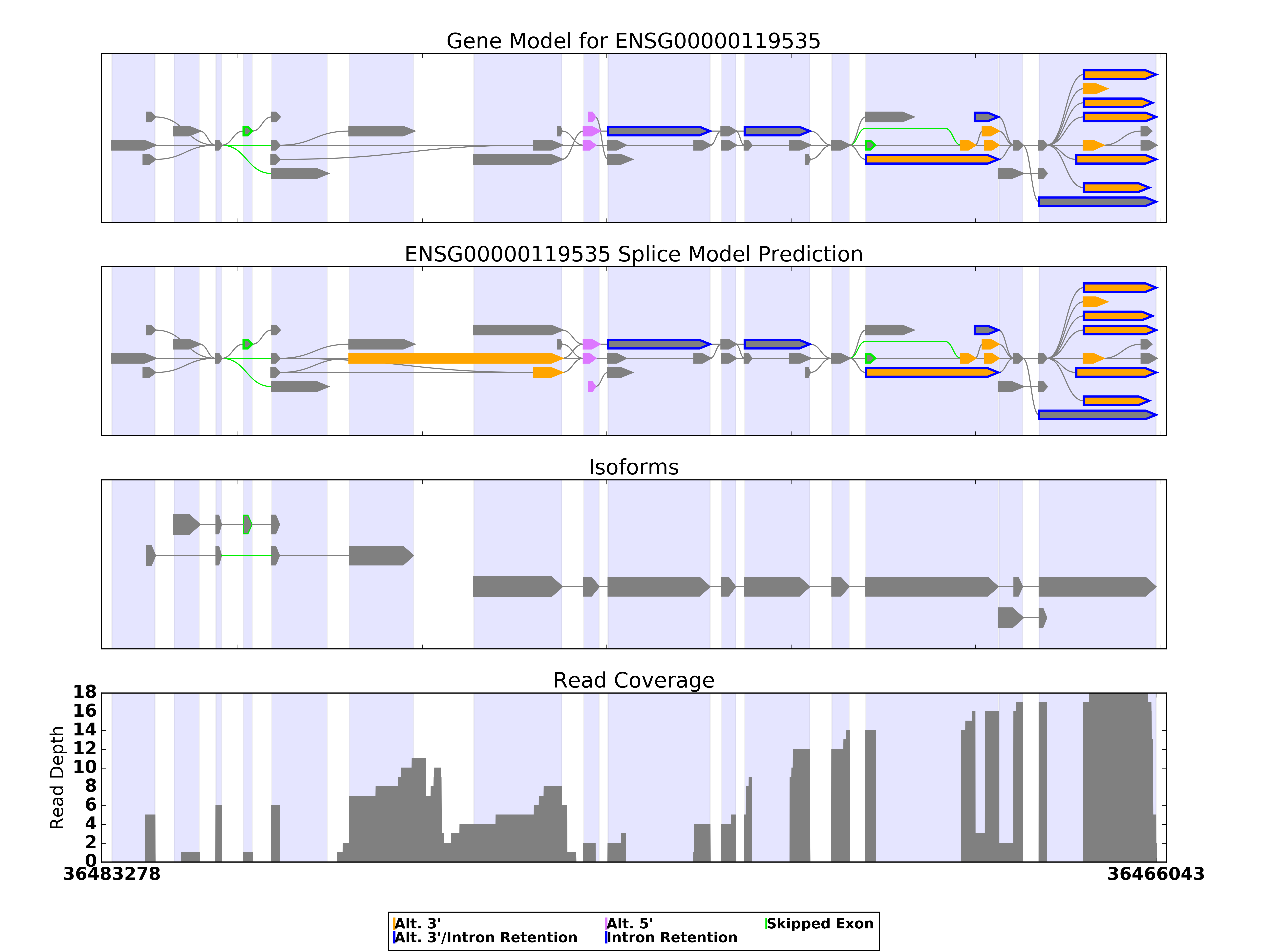
SpliceGrapher預(yù)測出的可變剪切模型可能會(huì)與已知的基因模型不同,如下圖,根據(jù)測序結(jié)果預(yù)測出的基因模型比已知的基因模型多了一個(gè)外顯子,橫跨第6、7個(gè)外顯子區(qū)域和第6個(gè)內(nèi)含子區(qū)域。這一點(diǎn)從Read Coverage上也能看出來,在第6個(gè)內(nèi)含子區(qū)域有不少reads覆蓋度。這也許是一個(gè)從未被發(fā)現(xiàn)的exon。
好了,關(guān)于SpliceGrapher的介紹就到這里了。歡迎大家訂購本公司的全長有參轉(zhuǎn)錄組分析產(chǎn)品,我們的可變剪切預(yù)測項(xiàng)目中會(huì)為大家畫出物種所有的基因可變剪切模型,并進(jìn)行分類梳理。























