2017-03-22
以PacBio公司的SMRT單分子實時測序技術(Single molecule real-time sequencing)為代表的三代測序技術,通過其獨有的環形一致性測序模式(Circular-consensus sequence,CCS),極大提高單堿基測序的準確率,遠超Illumina等二代測序技術。與傳統轉錄組測序項目相比,利用PacBio平臺的全長轉錄組測序技術可以直接獲得mRNA的全長,保證了mRNA序列的精確性。近期我們將陸續推出全長轉錄組測序技術相關文章,供討論和交流。我們在上一期向大家介紹了全長轉錄組測序的可變剪切模型可視化,本期將為大家對派森諾特色分析內容——融合基因染色體分布進行重點講解。
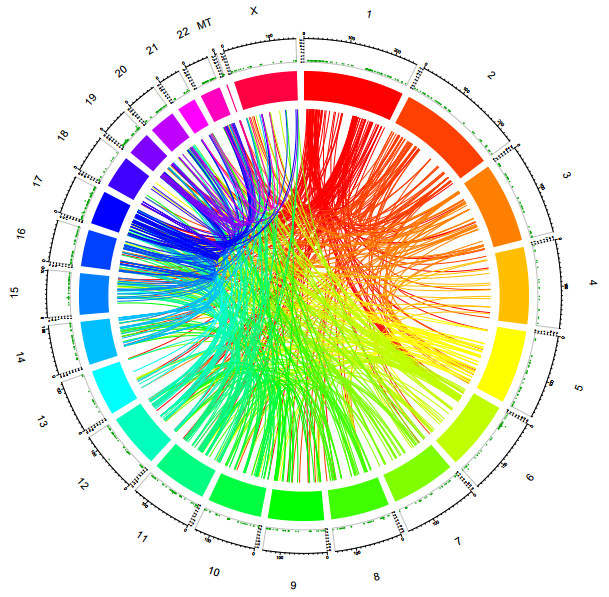
下面這張彩虹圖內圈以連線的形式展示了融合基因(融合基因是指將兩個或多個基因的編碼區首尾相連。置于同一套調控序列控制之下,構成的嵌合基因)的兩個組成基因在染色體上的分布位置,外圈則以散點圖的形式展示了各位點上的基因成為多少種融合基因的組成成分的情況,可以說以可視化的方式在一張圖上展示了滿滿的信息。那么這張圖是如何繪制的呢?
我們所用的工具就是R語言的circlize包。
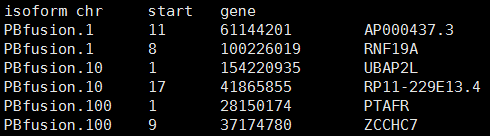
首先導入準備好的文件。這個文件中包含融合基因的兩個組成基因所屬染色體,起始位置,基因名字等信息。另外還需要導入各個染色體的長度,以便按比例畫出每個扇區所占角度。
扇區的范圍需要在開始時設置好,這里按染色體長度設置每個扇區所占角度的大小,我們會設置一個最小角度,以免染色體太小在圖上無法顯示出來,比如MT染色體。
然后從外至內畫每一個軌道(即圈圈)。首先畫第一個軌道,把染色體的名字依次放置在每個扇區的最外層軌道上。
接著畫第二個軌道,即以基因位點為橫坐標,該位點的基因被融合基因用上的頻率為縱坐標的散點圖,這里基因位點就是之前導入的基因起始位置,由于基因區域在染色體這么大的范圍內會被濃縮成一個點,所以我們這里直接采用基因的起始位置代表基因區域。絕大多數的基因僅被1種融合基因融合,但也有例外,從放大圖中可以看到1個基因竟成為了13種融合基因的組成基因之一,是不是難以置信呢?
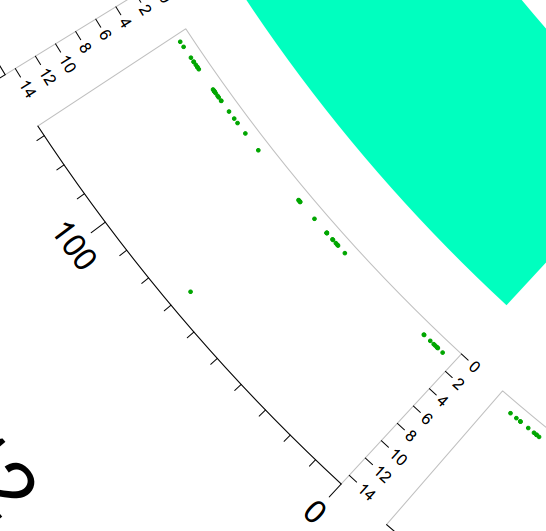
在這個軌道上還加上了染色體的長度范圍坐標,主刻度以100M bp為單位。
再接著畫第三個軌道,即代表染色體的色塊,填充顏色用的是五彩繽紛的彩虹色,更容易區分各染色體。
最后,用弧線連接融合基因的兩個組成基因即可,弧線的兩端分別連接至兩個組成基因的位點上,這里的基因位點與第二個軌道上的基因位點是對應的。連線的顏色是可以設置的,這里我們用第一個基因所在染色體的顏色。可以看到有的染色體上的基因多與同染色體上的基因融合,而有的染色體上的基因多與其他染色體上基因融合,這是否蘊含著某些染色體結構信息呢?


好了,到這里圖就畫完了,如果大家有感興趣的基因,我們還可以將基因的名字在圖中標識出來。


