2017-03-30
SMRT(Single Molecule, Real-Time)測序,即單分子實時測序,該方法基于納米小孔的單分子讀取技術,無需擴增即可快速完成序列讀取。Pacific Biosciences自2013年成功推出商業化的三代測序儀PacBio RSII后,三代測序開始被廣泛應用于基因組研究中。經過不斷的改良和升級,PacBio公司在2015年10月推出全新升級的三代測序儀PacBio Sequel測序系統,其具有的長讀長、高通量、高準確率等特點定將為研究領域帶來全新的三代測序體驗。
第三代測序平臺無疑是基因組項目的不二之選。作為測序行業的佼佼者,派森諾早在2016年便已經購入華東地區首臺Pacbio Sequel測序儀。目前平臺已經平穩運行較長時間并完成大量項目的測序工作。光聽小編說三代平臺怎么好,大家可能還不太相信。那就讓我們來看看三代測序技術為基因組的拼接帶來的巨大改變吧!
2017年3月6日發表于Nature genetics上的一篇文章報道了通過單分子測序、光學圖片和Hi-C技術完成的山羊基因組拼接。
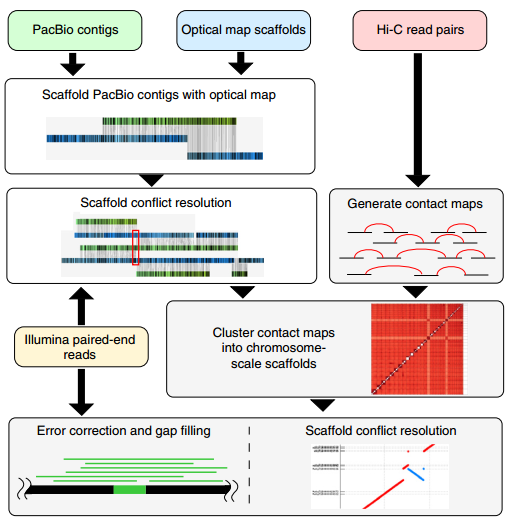
極大提升Contig N50
通過與2015年完成的山羊基因組進行對比,可以明顯的看出拼接效果有了顯著提升。詳見表1。CHIR_2.0為2015年上傳的基因組版本,測序平臺為Illumina,測序深度175.0×,組裝方法SOAPdenovo v.2;ARS1為該研究團隊發表的基因組版本,測序平臺Pabio,測序深度50.0×,組裝方法Celera Assembler v.8.2、BioNano Genomics Irys v.APRIL-2015、Lachesis Hi-C v.JUNE-2015。
表1 不同版本山羊基因組比較
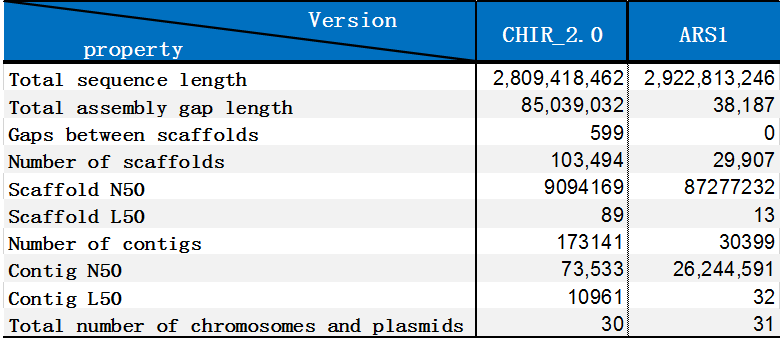
從表中可以看出,Contig N50由原來的73,533bp 提升至26,244,591bp。而這種Contig水平的提升則得益于使用了新一代的單分子測序技術,Pacbio的超長讀長使得組裝得到的Contig有了質的飛躍。在光學圖譜和Hi-C技術的輔助下,Scaffold水平也有很大提升,Gap數大幅下降。
顯著降低組裝錯誤
作者同時比較了幾個不同版本的山羊基因組的組裝錯誤情況,發現使用Pacbio平臺的組裝結果錯誤率要明顯低于單純通過二代測序平臺進行測序與組裝的基因組。
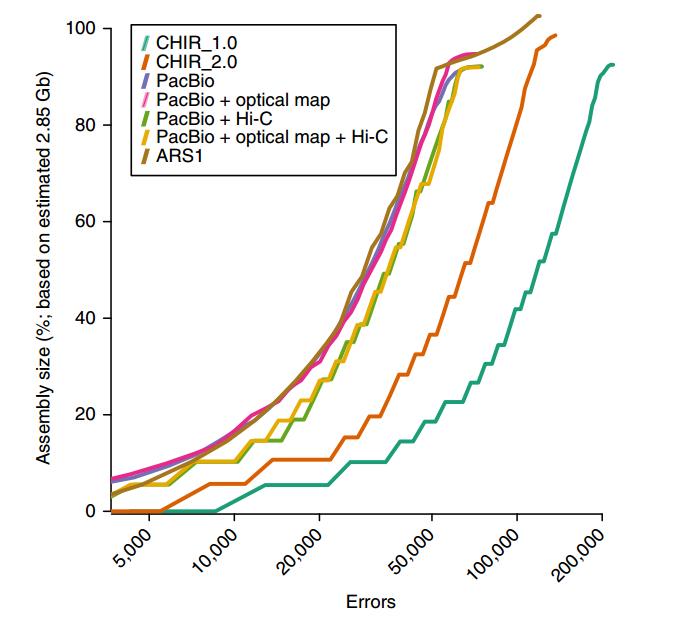
圖中CHIR_1.0和CHIR_2.0均為單獨使用二代測序平臺進行組裝的山羊基因組。
通過對比不同平臺的組裝效果,大家應該不難看出三代測序平臺在基因組組裝上的巨大優勢,如果加上光學圖譜、Hi-C等技術手段,則可以使基因組的組裝水平達到非常理想的狀態。接下來幾期小編將為大家帶來更多三代測序的優質文章分享,敬請期待!


