
2024-11-06
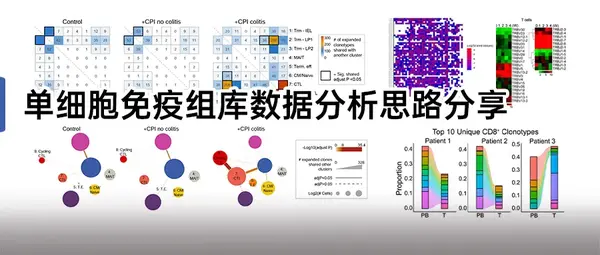
引言
免疫系統的復雜性和多樣性使得在單細胞水平研究免疫細胞的功能和特性至關重要。單細胞免疫組庫測序(5’scRNA + TCR/BCR)技術是一種能夠在單細胞層面同時解析細胞轉錄組信息和 T 細胞受體(TCR)、B 細胞受體(BCR)基因重排情況的前沿技術,這項技術為深入理解免疫細胞的發育、分化、抗原識別機制等提供了可能。在疾病研究中,無論是感染性疾病、自身免疫病還是腫瘤,它都像是一把精準的鑰匙,幫助我們開啟探索免疫反應奧秘的大門,為診斷、治療和疫苗研發等應用領域帶來全新視角。
一、數據來源背景介紹
在機體免疫系統中 T 細胞受體(TCR)和 B 細胞受體(BCR)的序列多樣性的總和被定義為免疫組庫。TCR\BCR具有抗原識別能力的高度可變區域CDR3區分布在mRNA的5端,TCR的CDR3區包括α鏈V、J基因片段和和β鏈V、D、J基因片段,BCR的CDR3區包括輕鏈V、J基因片段和重鏈V、D、J基因片段,V(D)J 基因基因重排或重組構成了免疫組庫序列的多樣性。
單細胞免疫組庫測序(5’scRNA + TCR/BCR)是以 10x Genomics 平臺的液滴微流控技術為基礎在單個細胞層面同時對轉錄組基因表達情況和 TCR/BCR 序列多樣性進行檢測的測序技術,最終,可以獲得兩個數據文庫,分別是單細胞 5 端基因表達文庫和免疫組庫(V (D) J 文庫)。
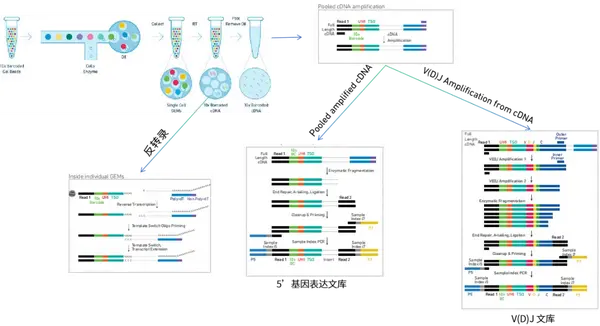
單細胞免疫組庫測序
二、數據分析流程
數據下機后后我們會對數據進行進一步質控過濾,進行細胞類型鑒定和克隆型分析,解析免疫細胞異質性和克隆型多樣性。
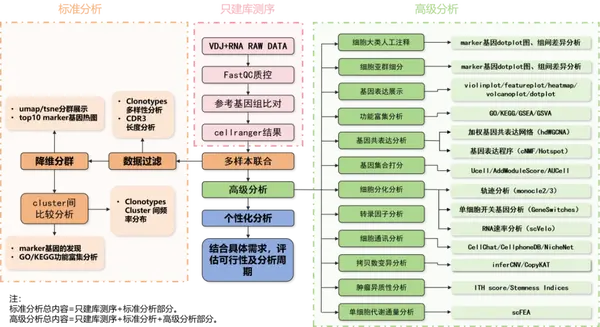
單細胞免疫組庫測序數據分析流程
三、數據分析思路
單細胞免疫組庫數據分析的核心思路就是篩選目標細胞亞型及克隆型,解析免疫細胞的異質性、細胞亞型的分化轉移狀態、克隆型多樣性等。
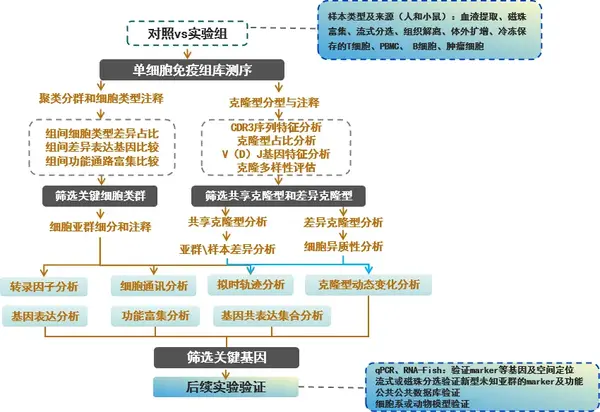
單細胞免疫組庫測序數據分析思路
1)鑒定細胞類型和克隆型
對細胞進行聚類分群和細胞注釋,構建組織細胞圖譜,鑒定免疫細胞類型及不同細胞亞群,解析細胞的異質性及免疫特征。對多種克隆型進行分型和注釋鑒定,解析CDR3序列特征和V(D)J基因特征,將具有相同或相似抗原識別能力(具有相同 TCR/BCR 序列)的免疫細胞歸為同一克隆型(Clonotype),從而了解免疫細胞群體的克隆結構。
分析方法:細胞大類注釋、CDR3序列特征分析、V(D)J基因特征分析、TopN 克隆展示(篩選關注頻率高的克隆型)。
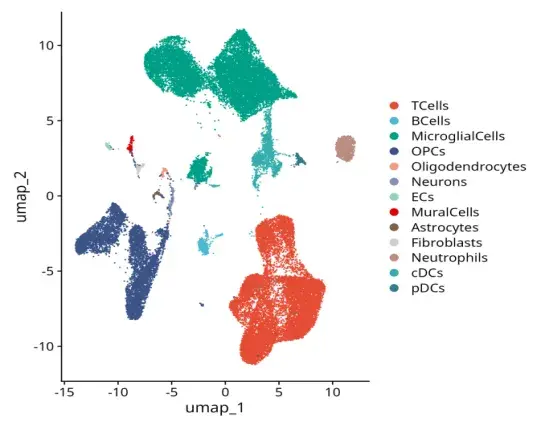
細胞圖譜
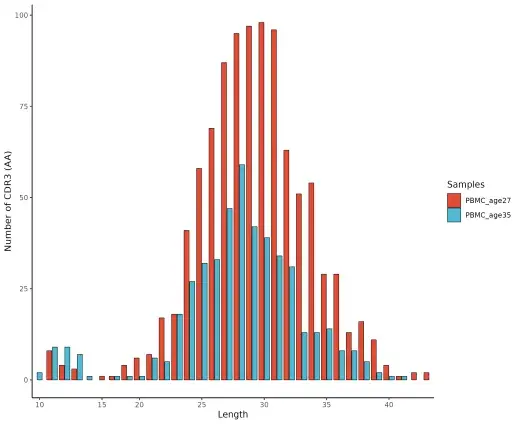
CDR3長度分布圖
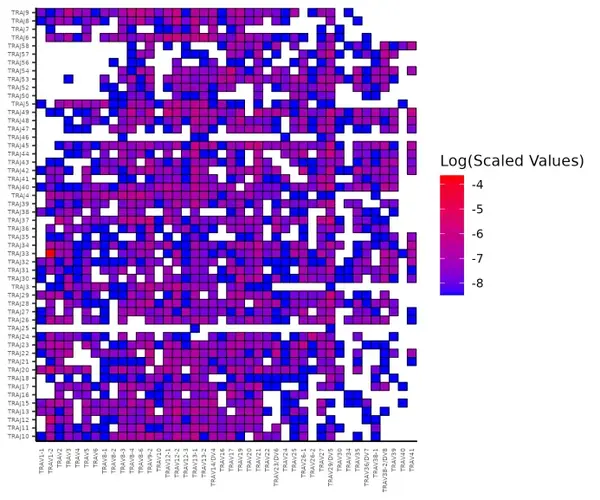
VJ基因使用分布
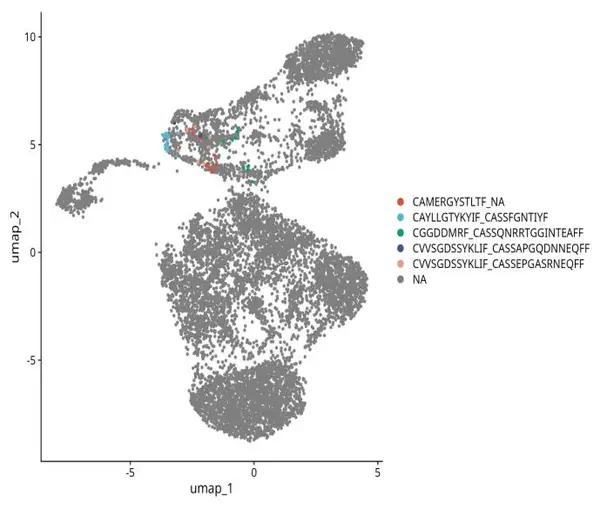
TopN 克隆型在 UMAP 中的映射圖
2)免疫細胞亞群克隆擴增差異分析
為了揭示細胞亞型的TCR\BCR克隆多樣性,先對T\B細胞進行再分群分析,鑒定不同的亞型細胞異質性和功能,通過對基因表達數據的分析和組間差異分析篩選出目標免疫細胞亞型,對目標細胞群統計該細胞中的抗原受體克隆多樣性及克隆型擴增頻率,同時篩選潛在的V(D)J基因。例如,發現某些細胞亞型具有獨特的克隆型分布,這可能與它們在免疫反應中的特殊功能相關。
分析方法:細胞亞群細分、組間差異分析、免疫細胞各亞群 clonetype頻數分布(解析不同細胞亞型的克隆擴增情況)、免疫細胞亞型V(D)J基因 Usage 熱圖(分析不同免疫細胞亞型中V(D)J基因的使用情況)。
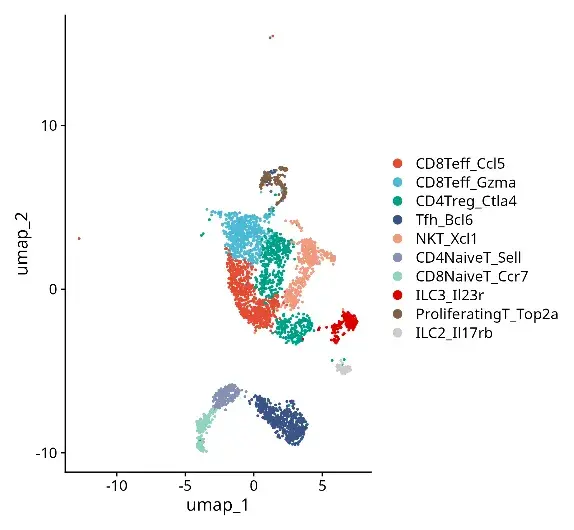
細胞亞群細分
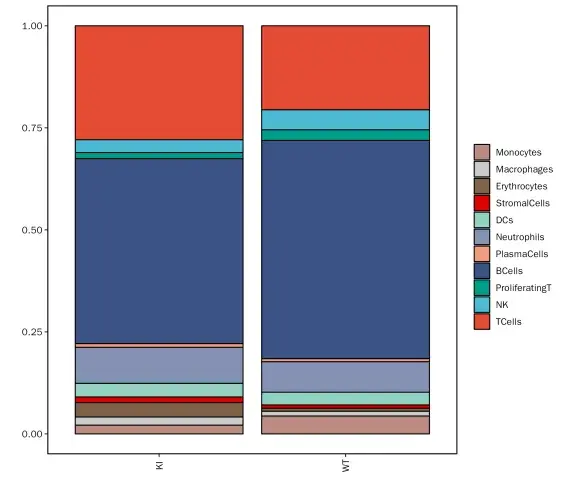
細胞亞群組間占比分析
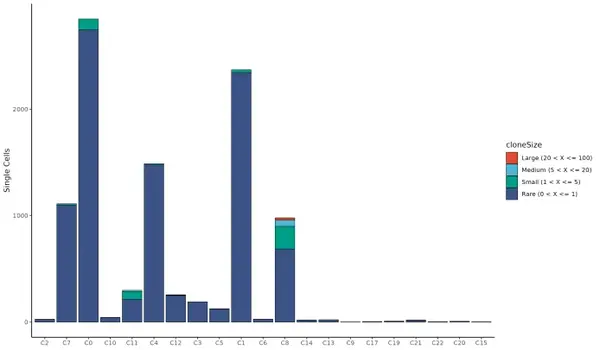
克隆型頻數
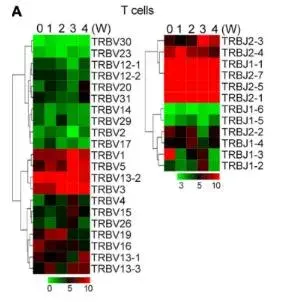
V(D)J基因 Usage 熱圖[1]
3)克隆型差異基因分析
對已鑒定的免疫細胞進行再分群分析,基于克隆型分型,通過分析不同克隆型亞型的特征基因,揭示每個克隆型獨特的基因表達模式,進行功能富集分析,解析具有不同克隆型的免疫細胞激活分化功能機制。對細胞亞群或樣本進行共享克隆型和差異克隆型分析,查看相同亞群或不同樣本中的共享或差異克隆型分布,找出免疫細胞亞型在不同樣本中對應的克隆型,探究clonetype 在不同樣本-細胞類型-分組之間的動態變化關系,對關心的克隆型進行亞群間的差異基因分析,篩選挖掘與疾病或其他免疫性狀相關的候選基因及其對應的受體基因。
分析方法:免疫細胞亞型共享克隆型分析、樣本間共享克隆型分析、克隆型動態變化分析、功能富集分析。
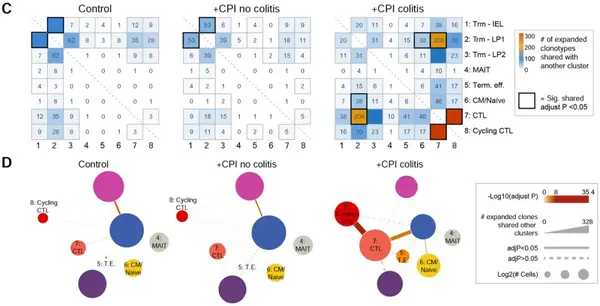
細胞亞群間共享克隆型分析[2]
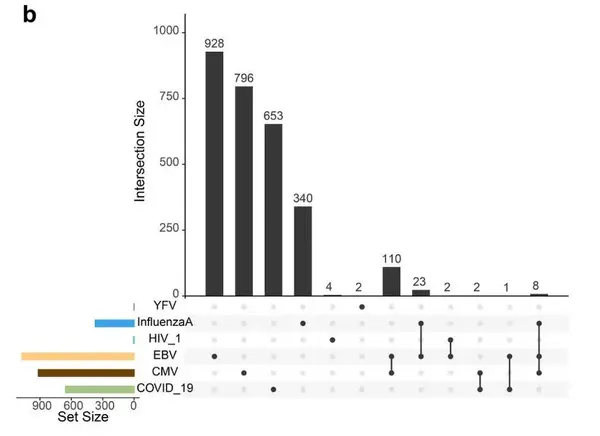
多樣本間獨有及共享克隆型分布[3]
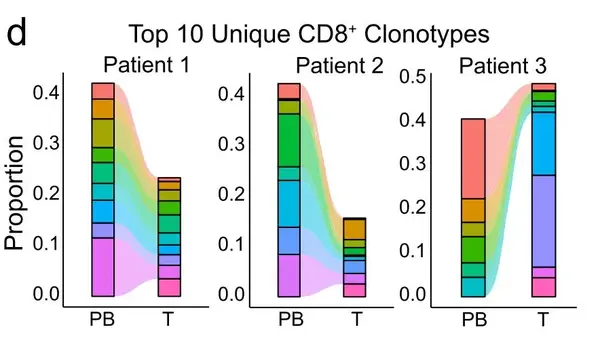
克隆型(可選擇某一組 topN 克隆型)樣本間變化桑基圖[4]
4)免疫細胞亞群分化轉移分析
根據研究目的篩選出目標免疫細胞亞型,通過功能富集分析研究亞型的細胞功能,最終篩選出具有克隆型擴張程度較強的亞型。利用擬時分析對目標亞型構建細胞軌跡,解析亞型細胞的轉移狀態,在每個軌跡中提取具有相同克隆型的細胞,使用 RNA 速率和擬時分析研究不同軌跡的方向性,從而最終確定它們的轉移方向和過程中的細胞狀態,了解免疫細胞在免疫反應過程中的動態變化,以及這些變化與 TCR/BCR 克隆型變化之間的關系。
分析方法:功能富集分析、擬時分析、 RNA 速率分析
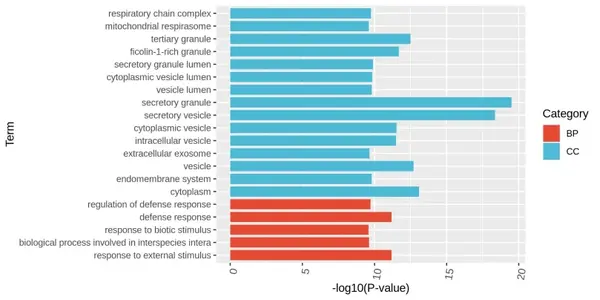
GO\KEGG功能富集分析
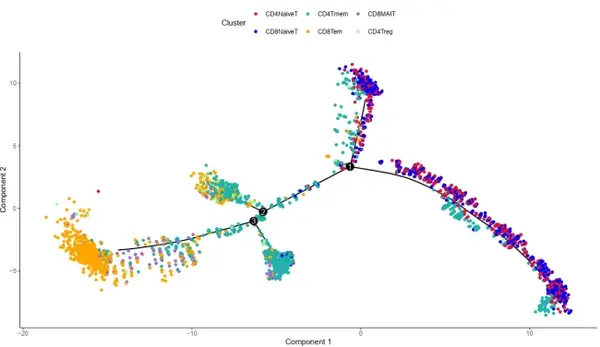
擬時序軌跡分析

RNA速率分析[2]
四、應用案例思路
案例1:構建免疫細胞圖譜
題目:A blueprint for tumor-infiltrating B cells across human cancers
中文題目:人類癌癥中腫瘤浸潤B細胞的藍圖
期刊:Science
影響因子:44.5
研究背景:
在腫瘤免疫過程中,腫瘤浸潤性B細胞在腫瘤免疫過程中扮演著重要的角色。這些B細胞表現出多種功能,主要是通過它們分化為漿細胞產生抗體的能力,但在不同的癌癥類型中,它們在時空上存在差異。解剖不同癌癥類型中B細胞的豐度和分化狀態有望改善免疫治療反應。
研究思路:
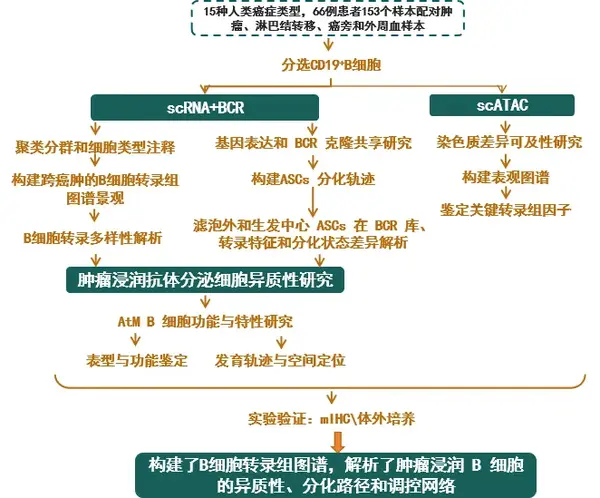
研究結論:
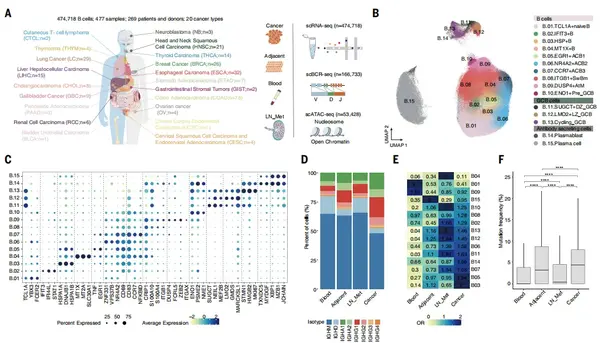
1. 該研究繪制了跨20種人類癌癥的腫瘤浸潤B細胞藍圖,揭示其異質性及兩條動態分化途徑,為后續研究提供基礎參考。
2. 發現腫瘤中存在生發中心(GC)和非生發中心(EF)兩條ASC分化途徑,且不同癌癥類型對二者有偏好,EF途徑主導的癌癥與不良臨床結果相關。
3. 明確了調控B細胞分化的轉錄因子及表觀遺傳網絡,不同途徑相關的B細胞亞群有特定的調控模式和表觀遺傳特征。
4. 表明EF途徑中的AtM B細胞具有耗竭和旁觀者表型,獨立于GC途徑發育,在空間上與T細胞相互作用,對T細胞功能有影響。
5. 證實谷氨酰胺代謝通過影響表觀遺傳修飾建立AtM B細胞的身份,且EF途徑相關的B細胞與免疫抑制微環境和不良預后相關,為B細胞靶向免疫療法提供思路。
案例2:腫瘤免疫機制研究
題目:Molecular Pathways of Colon Inflammation Induced by Cancer Immunotherapy
中文題目:癌癥免疫療法誘導結腸炎癥的分子途徑
期刊:Cell.
影響因子:45.5
研究背景:
靶向CTLA-4或PD-1/PD-L1通路的單克隆抗體—免疫檢查點抑制劑(CPI)治療可在多種人類惡性腫瘤中誘導持久反應,但也會誘導炎癥毒性,統稱為免疫相關不良事件(irAEs)。然而,導致這些嚴重炎癥副作用的免疫機制仍然知之甚少。
研究思路:
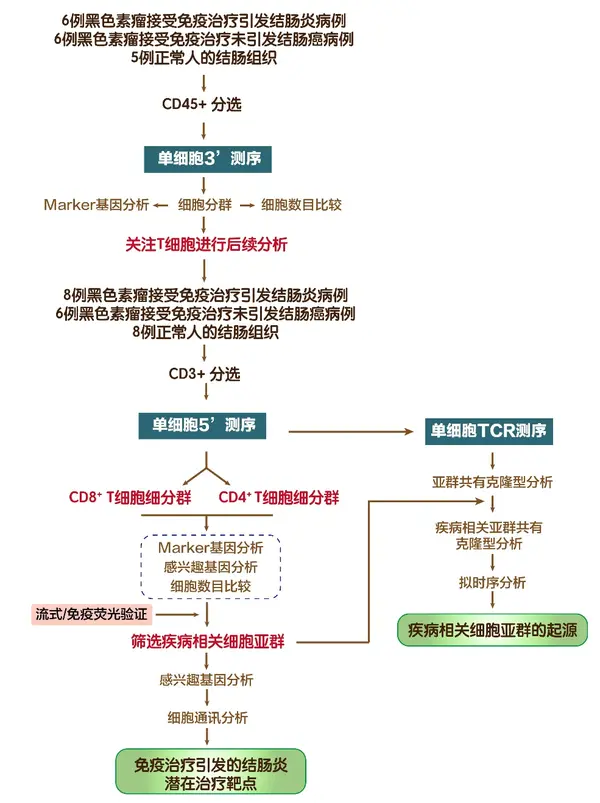
研究結論:
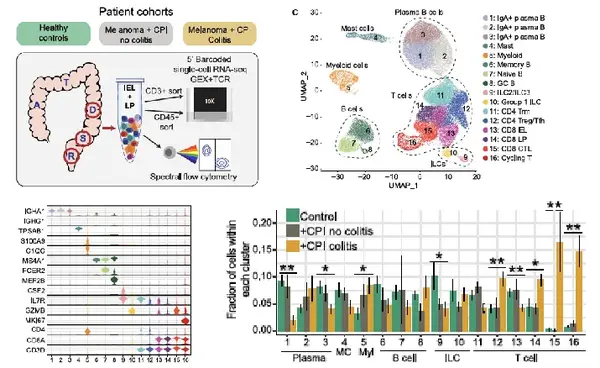
1. CPI結腸炎患者免疫細胞組成改變,CD8 T細胞毒性與增殖增強,CD4 T細胞亞群變化且Tregs未耗竭,髓系細胞頻率增加且基因表達改變。
2. 結腸炎相關CD8 T細胞簇與組織駐留記憶T細胞簇在TCR克隆型上存在動態聯系,提示分化軌跡。
3. 相關T細胞簇中抑制性受體及細胞因子相關基因表達變化,體現免疫細胞功能狀態及IFNγ的重要作用。
4. 發現多個潛在治療靶點,如趨化因子相關分子和細胞因子等,為CPI結腸炎治療提供方向。
案例3:抗體篩選
題目:Potent Neutralizing Antibodies against SARS-CoV-2 Identified by High-Throughput Single-Cell Sequencing of Convalescent Patients' B Cells
中文題目:通過對恢復期患者 B 細胞進行高通量單細胞測序鑒定出針對 SARS-CoV-2 的強效中和抗體
期刊:Cell
影響因子:45.5
研究背景:
COVID - 19 大流行急需有效的治療和預防干預措施。SARS - CoV - 2 的刺突(S)糖蛋白介導病毒進入宿主細胞,其受體結合域(RBD)與宿主細胞的 ACE2 受體結合。康復患者血漿中的中和抗體對治療 COVID - 19 有一定效果,但血漿大規模生產受限。從康復患者記憶 B 細胞中分離中和性單克隆抗體(mAbs)具有治療潛力,但傳統篩選方法耗時費力。本研究旨在應用高通量單細胞 RNA 和 VDJ 測序技術,快速高效地從康復患者中鑒定 SARS - CoV - 2 中和抗體。
研究思路:
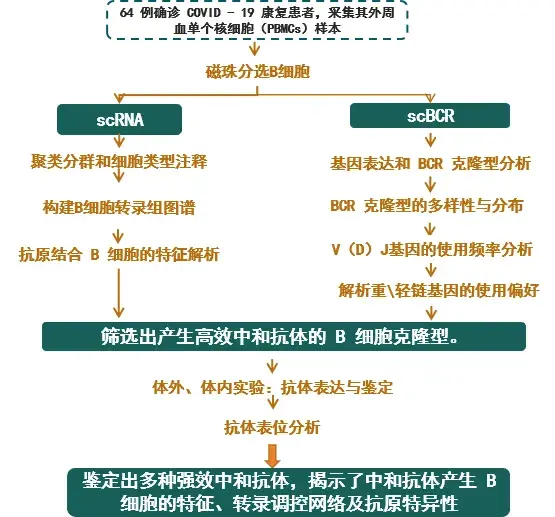
研究結論:
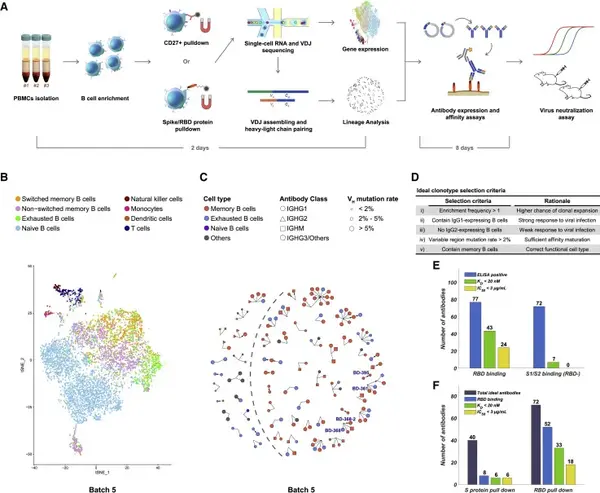
1. 運用高通量單細胞測序技術,從COVID - 19康復患者中鑒定出14種強效SARS - CoV - 2中和抗體,其中BD - 368 - 2效力最強。
2. BD - 368 - 2在hACE2轉基因小鼠模型中展現出高治療和預防功效,能改善感染小鼠生理狀況并降低病毒載量。
3. 解析中和抗體BD - 23 - Fab與S三聚體刺突蛋白的結構,發現其通過競爭抑制ACE2 - RBD結合來中和病毒,為篩選提供參考。
4. 基于CDR3H結構相似性篩選出部分中和抗體,提示可能存在針對SARS - CoV - 2特定表位的刻板型B細胞受體,為抗體療法提供思路。
參考文獻:
[1] Yang H Q , Wang Y S , Zhai K ,et al.Single-Cell TCR Sequencing Reveals the Dynamics of T Cell Repertoire Profiling During Pneumocystis Infection[J].Frontiers in Microbiology, 2021, 12:637500-.DOI:10.3389/fmicb.2021.637500.
[2]Luoma AM, Suo S, Williams HL, et al. Molecular Pathways of Colon Inflammation Induced by Cancer Immunotherapy. Cell. 2020;182(3):655-671.e22. doi:10.1016/j.cell.2020.06.001.
[3]Wang P,Jin X,Zhou W,et al.Comprehensive analysis of TCR repertoire in COVID-19 using single cell sequencing[J].Genomics, 2020, 113(2).DOI:10.1016/j.ygeno.2020.12.036.
[4]Borcherding N , Vishwakarma A , Voigt A P ,et al.Mapping the immune environment in clear cell renal carcinoma by single-cell genomics[J].Communications Biology, 2021, 4(122).DOI:10.1038/s42003-020-01625-6