2024-06-11
克隆細菌群體依靠單個細胞的轉錄變異來產生增加適應性的特殊狀態,了解所有細胞狀態需要在單細胞水平上研究同基因細菌種群,因此,利用單細胞測序對微生物進行細胞間異質性研究迫在眉睫。與此同時,因為微生物本身mRNA豐度低、半衰期短導致難以從單個細胞中捕獲足夠量的轉錄本,沒有poly-A尾不能通過3'端富集mRNA,細胞壁結構復雜裂解條件特異性強等問題,減緩微生物單細胞測序發展腳步。
2023年4月3日來自普林斯頓大學的Ryan McNulty研究團隊在Nature Microbiology期刊上發表一篇Probe-based bacterial single-cell RNA sequencing predicts toxin regulation的研究文章,文中介紹了一種 probe-based bacterial sequencing (ProBac-seq)的單細胞測序方法,通過利用10X Genomics微流控裝置將單個細菌細胞的微流體液滴封裝與 DNA 探針的原位雜交相結合,以實現高靈敏度和高通量微生物單細胞測序。作者將ProBac-seq應用于枯草芽孢桿菌和大腸桿菌,結果發現ProBac-seq可正確識別已知的細胞狀態,并揭示以前未報道的轉錄異質性;同時研究發現ProBac-seq應用于細菌發病機制下的產氣莢膜梭菌可以通過乙酸鹽調控亞群對毒素的異質性表達。總而言之,ProBac-seq是一種可用于揭示同基因微生物種群的異質性,并識別影響致病性的調控的微生物單細胞測序方法。
接下來,帶大家解析這篇文章:
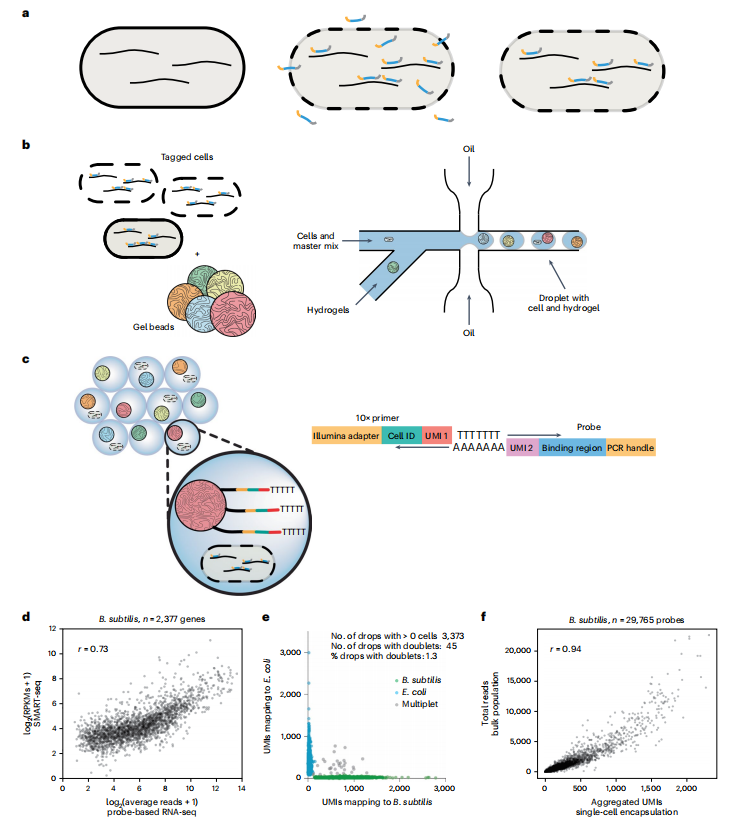
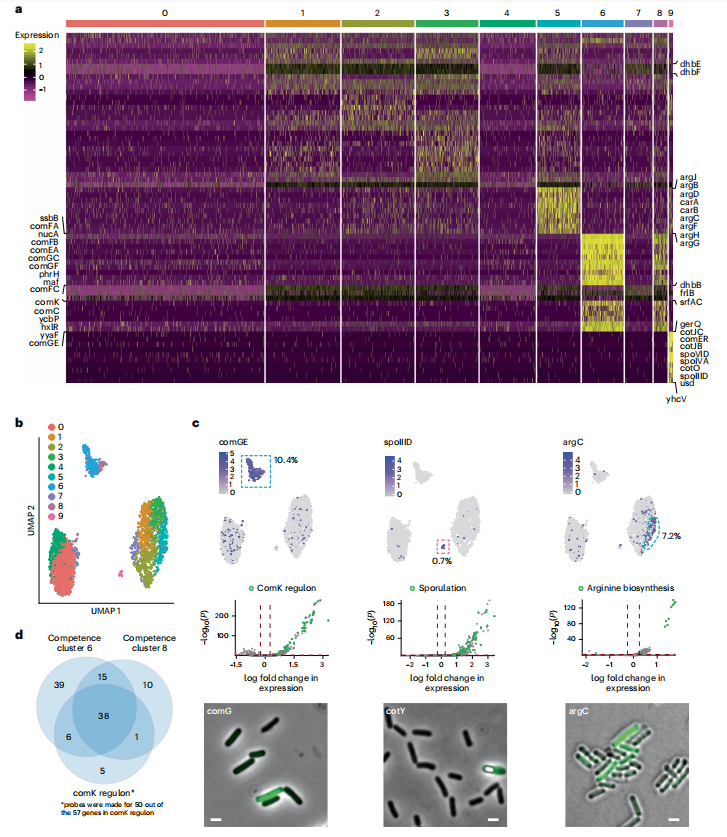
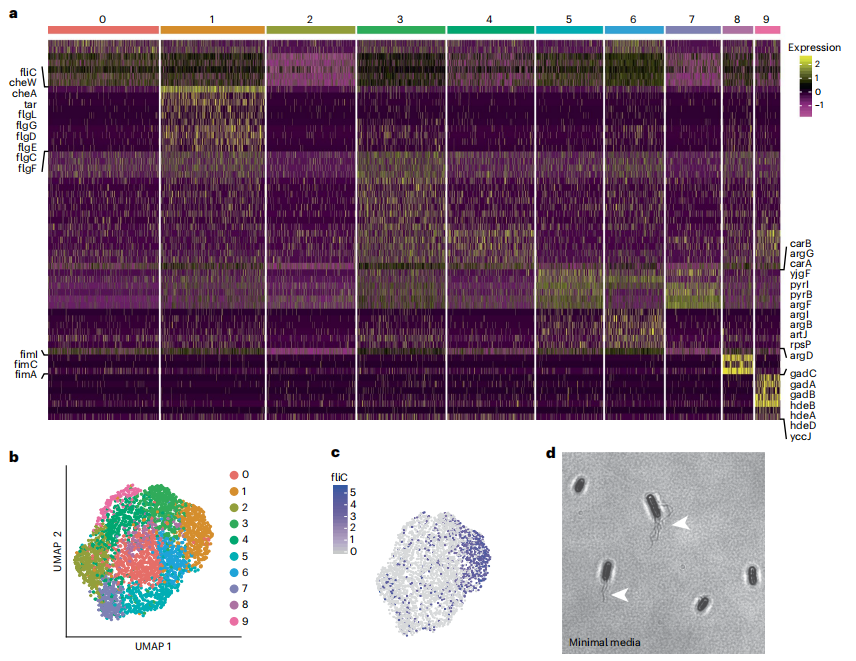
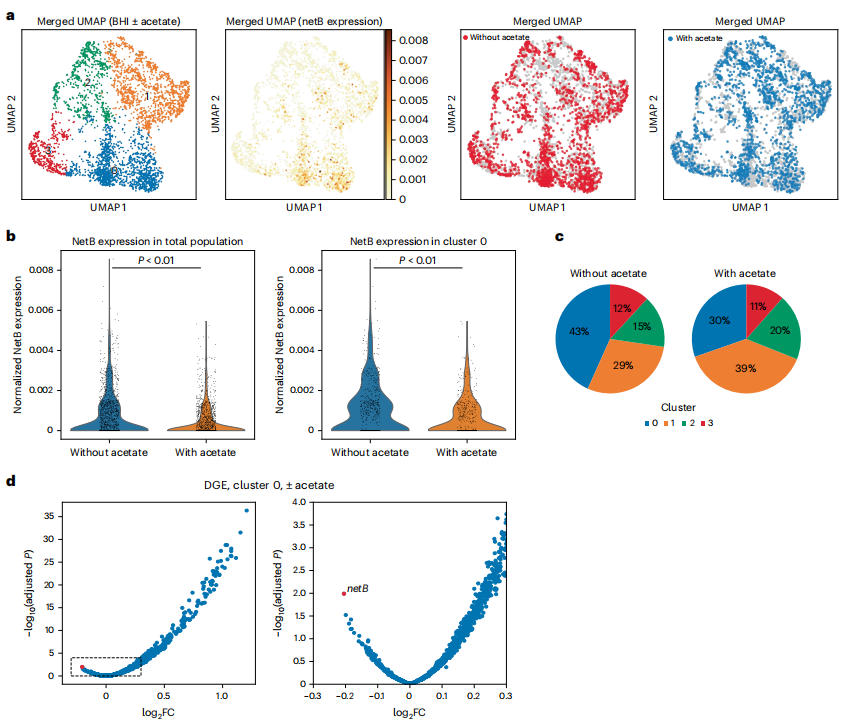
技術原理 為了利用現有的微流控單細胞測序平臺,作者設計了一種用DNA探針標記單個轉錄本的方法。首先,通過生信軟件或已發表的寡核苷酸陣列中確定獨特的50bp mRNA 結合序列,作為單鏈DNA探針的靶區,探針組成包括用于后續5' PCR擴增的PCR Handle、通過序列互補與相應PCR雜交的結合區、唯一分子標識符(UMI)、3' poly-A尾。為每個基因設計了多個探針(互補于不同的區域),以提高轉錄捕獲效率并減少因雜交不良或任何給定探針擴增不足而引起的背景噪音。選擇phi29 DNA 聚合酶進行滾環擴增(RCA),該方法在產生足量單鏈DNA產物進行scRNA-seq的同時,不易像傳統PCR那樣產生復合錯誤。在探針制備的最后一步時,通過引物擴展將UMI和poly-A尾添加到原探針的3 '端。滾環擴增方法允許重新擴增探針組,用于無限制的后續實驗,而無需重新訂購探針。 探針制備完成后,將細菌固定在 1% 多聚甲醛中,并通過溫和的溶菌酶處理進行透化以允許探針穿透,然后將相應的DNA探針與透化細菌孵育以雜交到固定化的mRNA,并通過洗滌去除未雜交或錯誤雜交的探針。選擇一定量處理后細胞與PCR試劑和引物混合,利用10×Genomics微流控裝置生成液滴,建庫生成3' 單細胞轉錄組文庫。 圖1 ProBac-seq方法與驗證 細菌scRNA-seq驗證 為驗證ProBac-seq能否表征細胞的轉錄狀態,作者將晚期枯草芽孢桿菌等分為兩份,分別使用SMART-seq和ProBac-seq進行處理,結果表明,50℃條件下利用雜交探針得到文庫結果與SMART-seq文庫結果相似(圖1d)。為驗證通過10×Genomics是否可以通過封裝單個細菌進行單細胞轉錄組測序,將大腸桿菌和枯草芽孢桿菌細胞進行獨立固定,并用對應于其各自基因組的探針進行預處理,將制備好的樣本與帶有DNA引物的PCRMIX混合,上10×Genomics生成油包水后進行建庫,結果表明,微流控平臺可以成功地通過Barcode區分不同細菌的單個細胞(圖1e),同時發現兩個樣本捕獲探針的豐度高度相關,說明來自單細胞的信號提供了轉錄狀態的良好表征(圖1f)。 為了證明ProBac-seq對異質性研究的廣泛應用,作者利用ProBac-seq對不同類型微生物種群進行單細胞測序分析: 1、ProBac-seq鑒定枯草芽孢桿菌的異質性 作者在添加蘋果酸的M9基礎培養基中選擇指數后期的枯草芽孢桿菌進行ProBac-seq,捕獲2784個細胞,分析得到由10個細胞簇組成的四種不同轉錄組特征(圖2a、b)。與作者預期一致的是,觀察到了具有遺傳能力特征的細胞亞群(簇 6 和 8)。探針靶向的 50 個 comK 調控子基因中有 45 個在簇 6 和 8 中差異過表達(圖2c、d)。 總而言之,利用ProBac-seq對枯草芽孢桿菌進行分析,不僅可以獲得已知的細胞狀態,并且能夠識別以前未報道的細胞狀態,揭示潛在基因組結構的特征。 圖2 ProBac-seq分析揭示了枯草芽孢桿菌的不同轉錄狀態 2、克隆大腸桿菌群體中的表達異質性 作者使用ProBac-seq來表征大腸桿菌MG1655的轉錄異質性,該菌在M9基礎培養基中生長,并化學固定在對數中期。捕獲3315 個細胞,通過聚類將細胞分為9個細胞簇(圖3a、b),作者發現與枯草芽孢桿菌相似的是,某些簇可以通過DGE分配給特定的生物過程,例如簇 1 中的細胞獨特地上調與細胞運動有關的基因,鞭毛關鍵基因(鞭毛蛋白,fliC)優先由簇1中的細胞表達(圖3c);簇 5、6 和 7 中顯示出氨基甲酰磷酸酯的不同用途;簇 8 中 fimI、fimC 和 fimA 的顯著上調,而fim 操縱子的下游基因編碼Ⅰ型菌毛的成分,這些成分與生物膜的形成和致病性有關,并且已知是異質表達的。為了證實細胞亞群的存在,使用Remel Flagella染色劑對在相同條件下生長的細胞進行染色并觀察(圖3d),結果發現大約36 %的細胞具有組裝的鞭毛,這一比例高于在簇1中的14.5%,導致結果差異的原因可能是一些有鞭毛的細胞可能已經退出轉錄狀態,但仍然保留了翻譯的蛋白質產物。 綜上所述,作者對大腸桿菌和枯草芽孢桿菌的觀察表明,廣泛的表型異質性是細菌的一般特征,精氨酸代謝等過程通常局限于特殊細胞。此外,與毒力相關的基因分離到特定的細胞群中,表明特定的細胞類型可能與致病性有關。 圖3 在基礎培養基中生長的大腸桿菌的異質基因表達 3、產氣莢膜梭菌毒素表達異質性 為了驗證ProBac-seq能否在真正的病原體中鑒定不同細胞亞群,作者選擇產氣莢膜梭菌研究其毒素的產生,產氣莢膜梭菌是一種革蘭氏陽性孢子形成細菌,是人類和牲畜的重要病原體,導致壞死性腸炎的A型產氣莢膜梭菌的毒力因子為NetB。通過BHI培養基中生長至指數期晚期的產氣莢膜梭菌進行單細胞分析,捕獲1508個細胞,通過聚類將細胞分為4個細胞簇(圖4a)。雖然NetB在所有簇中都有表達,但NetB差異過表達定義了簇0(占比43%)。 產氣莢膜梭菌中的另一種外毒素 pfoA 的產生已被證明受短鏈脂肪酸(如乙酸鹽和丁酸鹽)的調節,短鏈脂肪酸在胃腸道中含量豐富,并已被證明可以抑制 pfoA 外毒素,控制脂肪酸降解的基因在NetB過表達簇中上調,因此作者假設乙酸鹽可用于降低產氣莢膜梭菌的NetB 表達。為了驗證假設,作者在培養產氣莢膜梭菌的培養基中加入乙酸鈉來擾亂產氣莢膜梭菌的細胞外環境,并選擇指數后期的產氣莢膜梭菌進行ProBac-seq,結果發現,NetB 的表達再次集中在簇 0 中,其中與未處理的細胞相比,用乙酸鹽處理的細胞表達的毒素顯著減少,乙酸鹽的添加顯著降低了處于初級產毒狀態(簇0)的細胞比例從43%降低到30%(圖4c),毒素基因表達的減少與在含有乙酸鹽的培養基中生長的培養物中分泌的細胞外NetB蛋白的減少一致。 綜上所述,NetB由專門的細胞亞群差異表達,并且提供有利于替代細胞狀態的生長條件可以降低克隆細菌群體中毒力細胞的比例。 圖4 產氣莢膜梭菌中的NetB毒素優先由細胞亞群表達,并可通過添加乙酸鹽下調表達 結論 綜上所述,ProBac-seq研究細菌群落的異質性的優勢在于,它為研究數千個單細胞的全基因組表達水平提供了一種無偏倚的方法,從而可以全面分析基因-基因和細胞-細胞相關性,最大限度地捕獲轉錄本,避免核糖體RNA干擾。在ProBac-seq中,由于同一探針的多個拷貝不能在任何給定的轉錄本上占據相同的位置,并且測序文庫直接由雜交的DNA探針制成,而不產生cDNA,因此歸一化UMI的數量與每個細胞中存在的mRNA靶標的數量直接相關。此外,針對每個轉錄本不同區域的多個探針的設計允許檢測任何給定 mRNA 分子的更高機會,并為每個轉錄本提供額外的獨立測定,可用于為低水平表達的基因獲得足夠的統計意義。但同時也存在一定局限性,例如,依賴于探針雜交,需要基因組的先驗知識以及設計和訂購大量探針的前期成本,同時依賴于10×平臺。但盡管存在這些局限性,ProBac-seq仍具有快速、經濟高效的特點,并可生成具有準確轉錄本定量的高分辨率數據集。