
2022-05-16

《Journal of Fungi》
影響因子:5.816
近日,派森諾生物與北京林業大學合作,在真菌基因組領域的《Journal of Fungi》發表新研究成果!在本研究中,對分離得到的3個靈芝屬菌株進行了全基因組測序,并結合該屬的其他物種進行系統發育樹構建和比較基因組學分析。本研究揭示了靈芝基因組一系列特定的遺傳特征,為進一步刻畫這一重要類群的生態特征提供了基礎信息。
研究背景
靈芝屬(靈芝科、多空目、擔子菌科)是在森林生態系統中發現的具有生態學和經濟意義的真菌,其作為木材腐爛真菌,可以有限分解植物細胞壁的木質素、纖維素和半纖維素。此外,作為藥物,靈芝子實體被用于生產提高人體免疫力的藥物,以及用于抗衰老和抗癌治療。靈芝屬是靈芝科最大的屬,根據《Index Fungorum》分類可以分為461個分類單元。然而,迄今為止,只有4個靈芝物種的完整基因組被報道。本研究另外對3個靈芝屬基因組進行和測序和分析,并將其與之前測序的其他類群靈芝進行了比較,重點分析了細胞色素P450s的編碼基因、與次生代謝和木材降解相關的轉運蛋白和調控蛋白。研究了靈芝基因集的多樣性,并確定不同物種特有的遺傳特性。
研究材料與方法
1.實驗材料:櫟樹上分離得到的南方靈芝Ganoderma australe strain Cui 17254、 白肉靈芝G. leucocontextum strain Dai 12418和 靈芝G. lingzhi strain Cui 9166
2.測序平臺: Pacbio RSII、Illumina Miseq
3.分析內容:真菌基因組近完成圖測序、轉座子分析、CAZYme預測、分泌蛋白預測、蛋白酶預測、共線性分析、ITs進化樹構建、基因家族分析、單拷貝基因進化樹構建和次級代謝產物基因簇預測等。
研究結果
靈芝的基因組特性
本研究對G.australe strain Cui 17254,G. leucocontextum strain Dai 12418和 G. lingzhi strain Cui 9166進行了全基因組測序,結果發現G.australe strain Cui 17254基因組大小為84.27M,編碼基因個數為20460個,這是迄今為止已報道的最大靈芝基因組。G. leucocontextum strain Dai 12418和 G. lingzhi strain Cui 9166的基因組的基因大小較低,分別為60.34M和60.56M,預測得到的編碼基因個數分別為15,007和16,592個。BUSCO完整性為73.1%-99.8%,表明此次測序組裝得到了基因組上的大部分編碼基因。
表1 三種新測序靈芝基因組的基因組特征
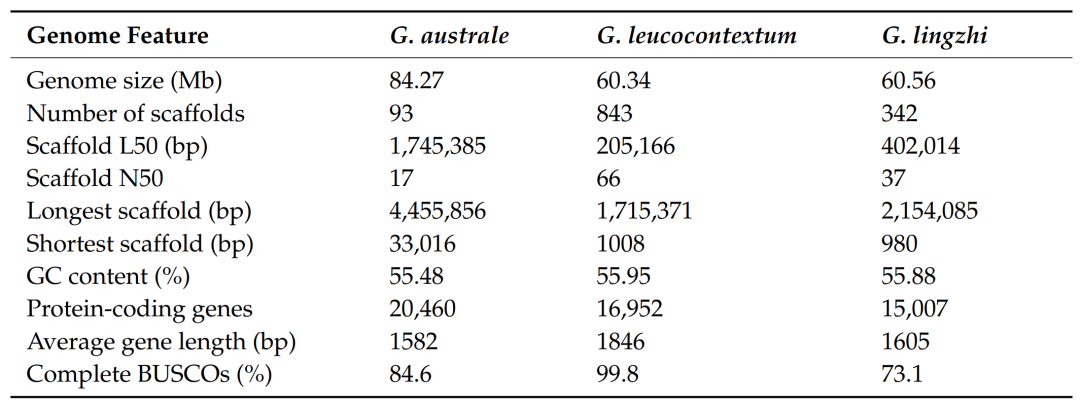
本研究將3個新測的靈芝基因組與4個已發表的靈芝屬基因組和其他51個擔子菌門基因組進行了比較,結果發現這58個基因組大小為19.66-109.88M,預測得到的基因個數在6785-26226之間。其中南方靈芝的基因組最大, G. australe和G. boninense比其他木材腐生物基因組大2-3倍;白腐菌、褐腐菌和外生菌根菌的平均基因組大小差異不顯著。
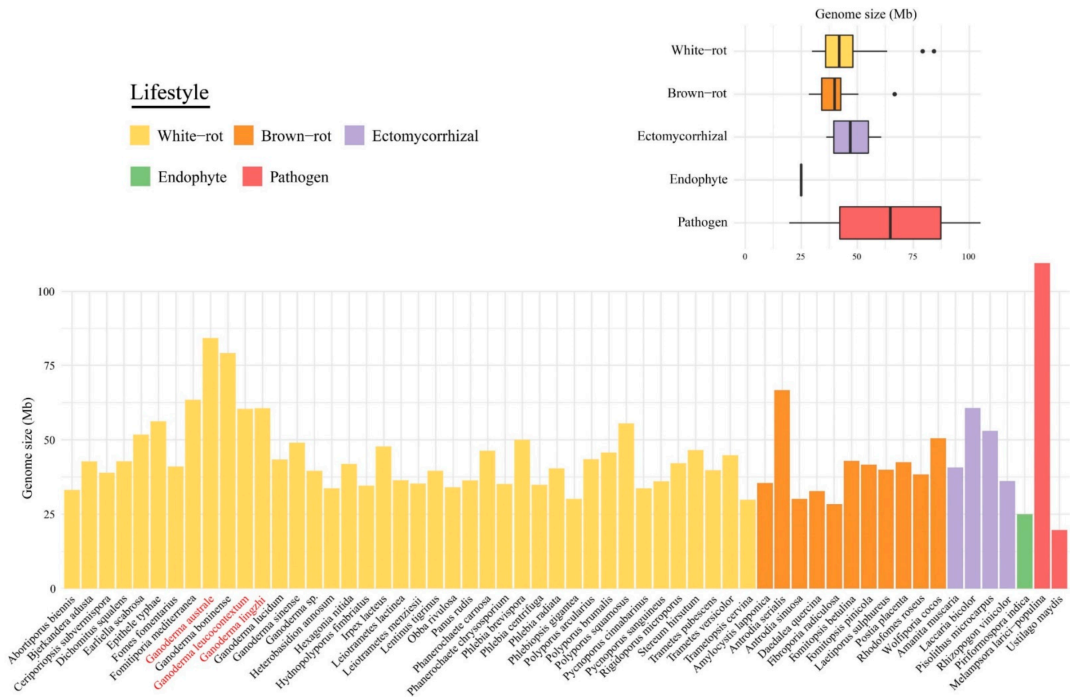
圖1 58個基因組大小比較圖
靈芝基因組共線性分析
選擇靈芝基因組中占總組合13%-75%的前10條contig進行共線性分析,結果發現與其他物種相比,G.lingzhi和G.lucidum的共線性比例最高(72%),而G.boninense和 G. leucocontextum的共線性片段比例較低,反映了較高的基因組差異。
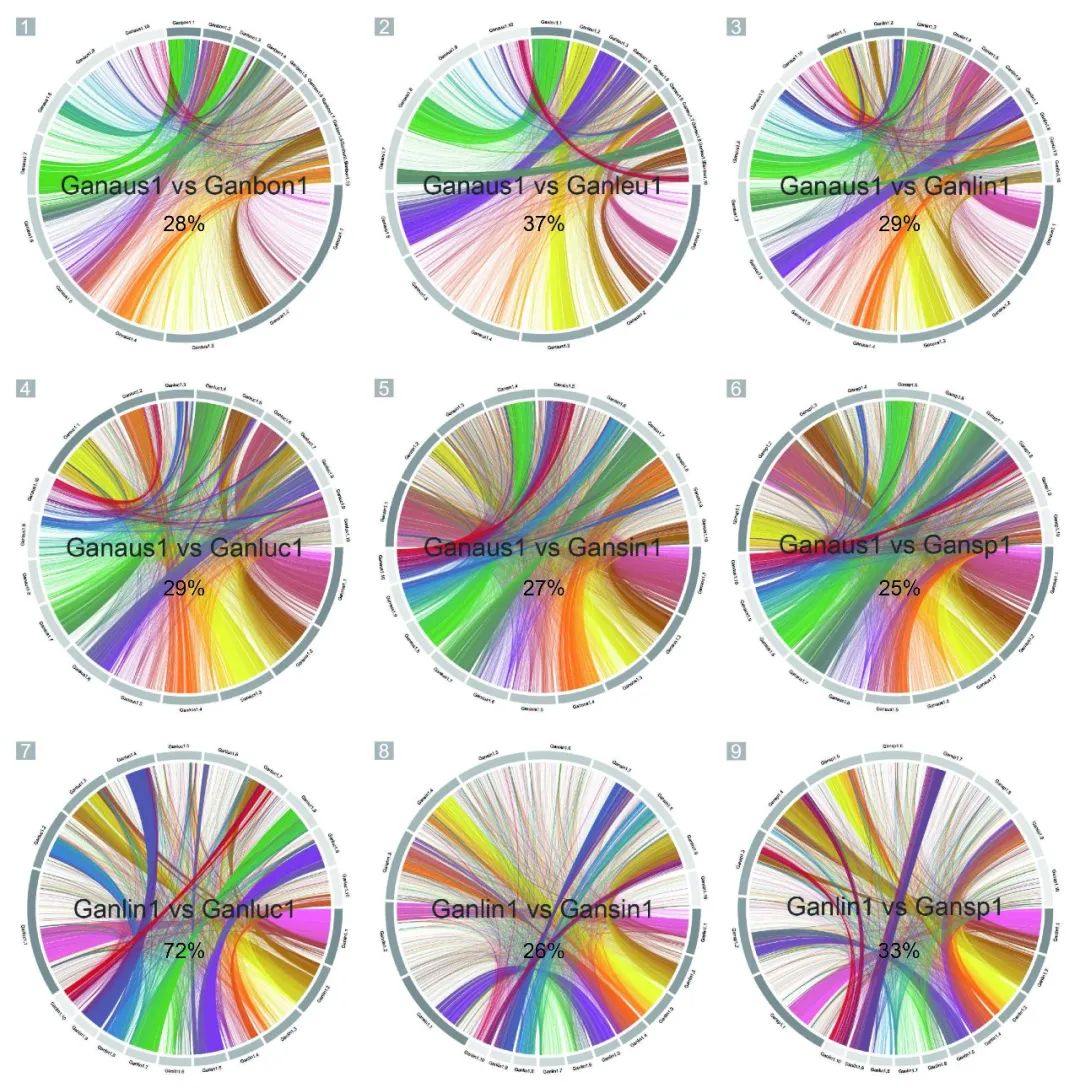
圖2 基因組共線性分析圖
靈芝菌株的系統發育分析
以Sanguinoderma sp.為外群,對12個種靈芝的23條ITs序列進行系統發育分析。結果發現有2個靈芝沒有聚在一起,說明G.260125-1實際上應該被鑒定為靈芝。Ganoderma sp. 10597 SS1 和G. sessile聚在一起,表明這一菌株可能這一門的固有種。
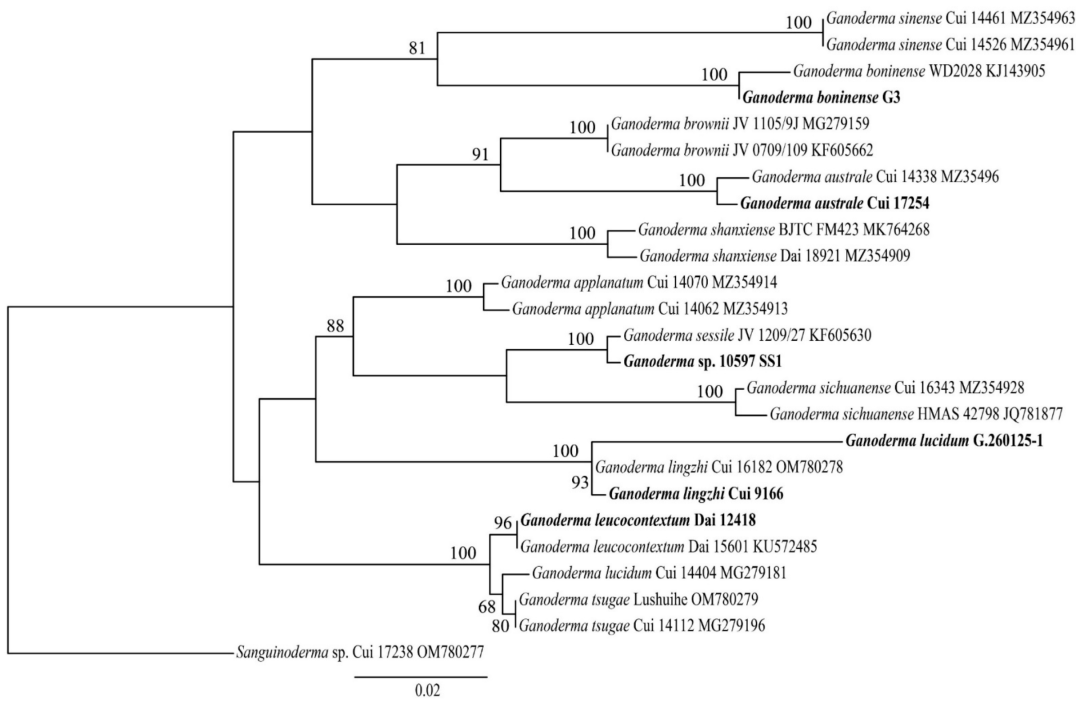
圖3 ITs系統發育樹
靈芝屬植物及其近緣多孔目的系統發育分析
基于143個單拷貝基因的蛋白序列進行進化樹構建,結果確定了多孔孔目的5個主要分支:核心多孔空類分支、前孔類分支、凝膠多孔類分支、靜脈類分支和殘留的多孔空類分支。其中靈芝物種聚集在核心多孔孔類分支中,這與共線性分析的結果一致。褐腐菌和外生菌根菌形成單系群,目前的分析也證實了白腐病的生活方式是進化的多品種。
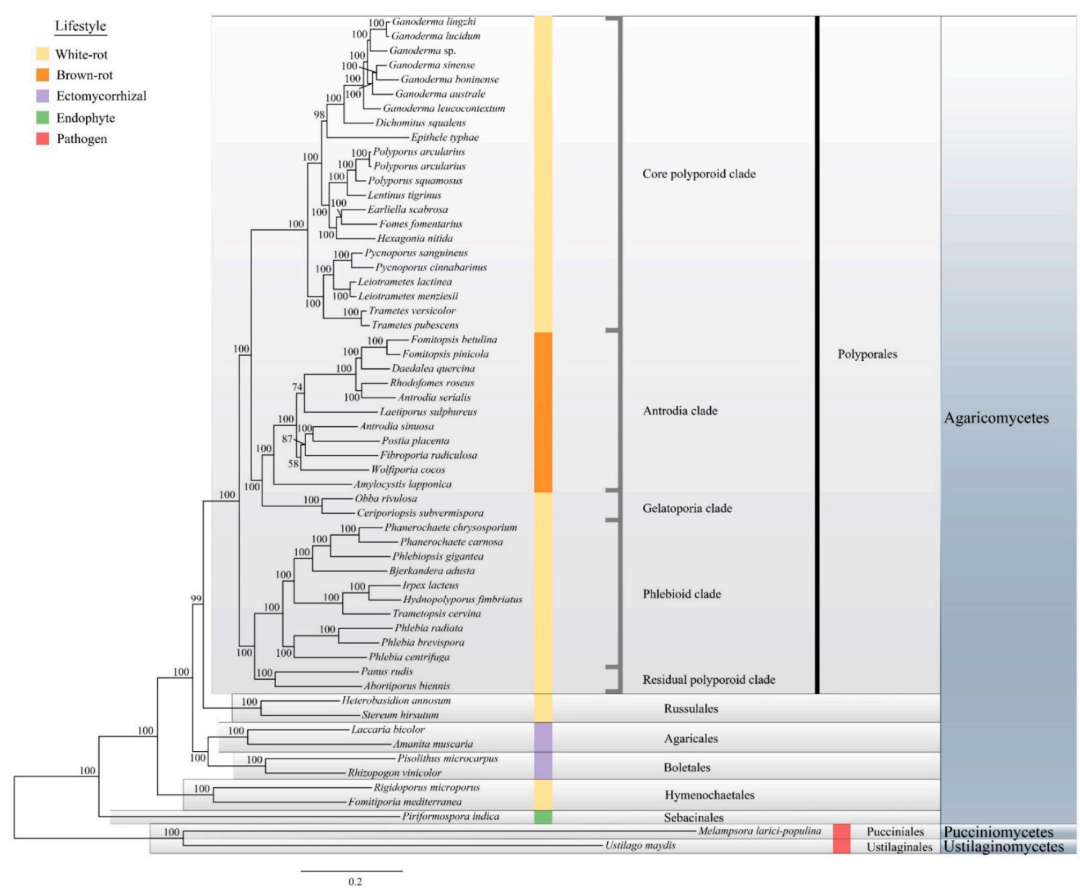
圖4 單拷貝基因系統發育樹
轉座元件識別
靈芝屬中優勢TEs屬長末端重復(LTR)反轉錄轉座子,未知TEs在靈芝屬中所占比例較大,尤其是G. australe(占總組裝量的10.5%)和G. lingzhi(占總組裝量的11.1%),說明它們可能在基因組重排中發揮了關鍵作用。G. leucocontextum的TE覆蓋率最大(占總組裝量的18.32%),TE分布最多樣化,包括DNA轉座子的簡單重復、IS3EU和Dada、非LTR反轉錄轉座子的RTEX和L1,這是靈芝品種中唯一的重復元件。與其他物種相比,G. leucocontextum中IS3EU和Dada序列數量顯著增多。與其他靈芝品種相比,南方靈芝含有更多的T ad1非LTR逆轉錄轉座子和Helitron DNA轉座子。58個被分析基因組的TE覆蓋率從0.37%到41.69%。
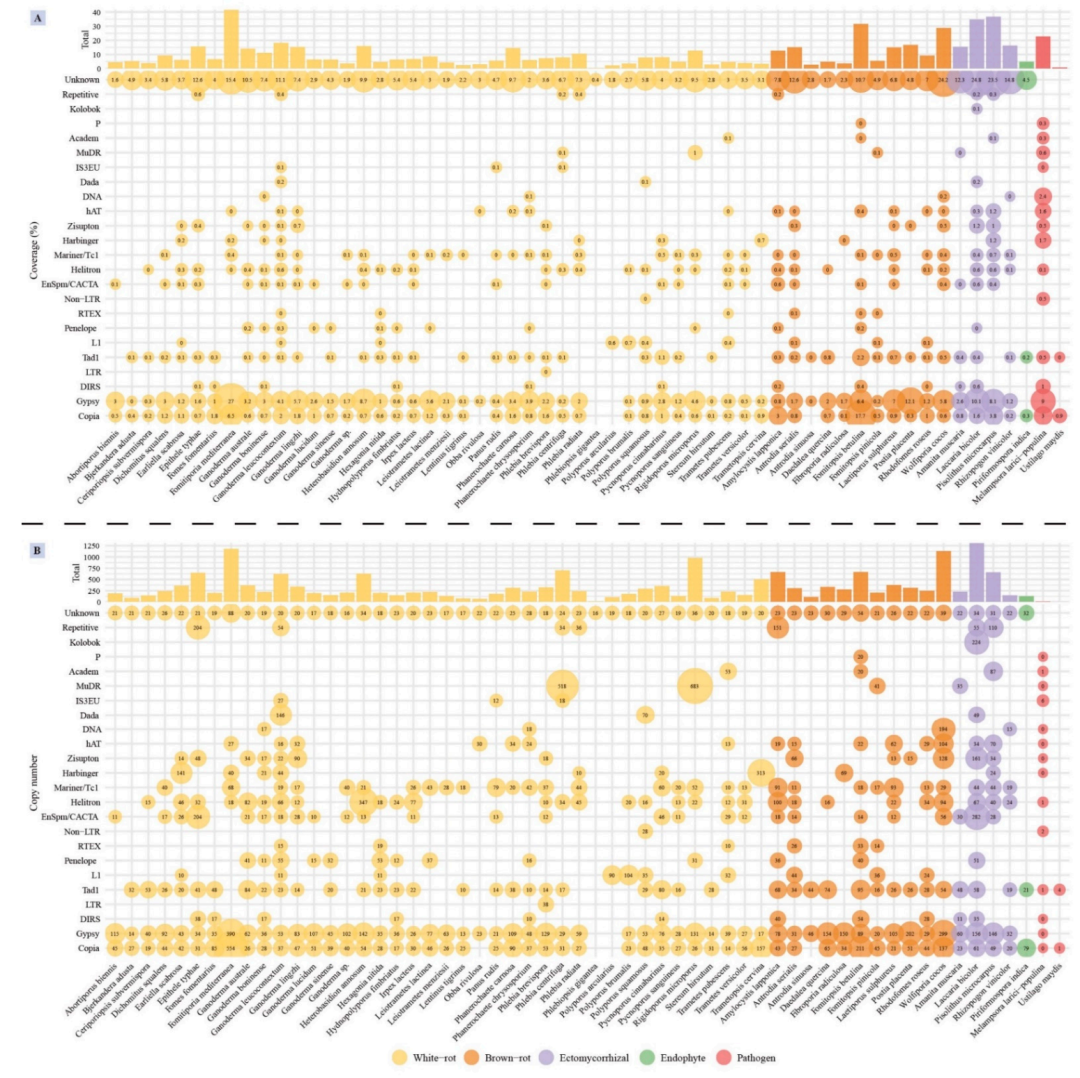
圖5 轉座元件在58個基因組中的分布、覆蓋率和拷貝數圖
次級代謝產物基因簇分析
本次分析一共鑒定了16種SM生物合成簇,除Rhizopogon vinicolor外,病原菌、外生菌根真菌和內生真菌的SM基因數量普遍低于腐生菌,這一差異主要是由于t1pks、萜類相關基因以及被標記為“假設”和“其他”的基因含量較低所致,有褐腐菌屬組成的前肢亞支通過t1pks、吲哚和脂肪酸相關基因的富集以及NRPS基因簇的缺失而與其它的Polyporales亞支分離。在Polyporales下的白腐真菌中,phlebioid分支的t3pks含量較高,而萜類相關基因含量較低。其核心類群包含了具有多種生物合成基因簇的物種,其中靈芝中含有31個萜類相關基因簇和80個生物合成基因簇。值得注意的是,以靈芝和G. lucidum為主要藥用相關種的萜類相關基因群數量最少,而南灰三萜和致病性博南三萜含量最高。G. lucidum、G. leucocontextum、T. rametes versicolor和T. pubescens基因組編碼了一個與lantipeptide產生相關的簇,前三個基因組被鑒定為與t1pks相關。
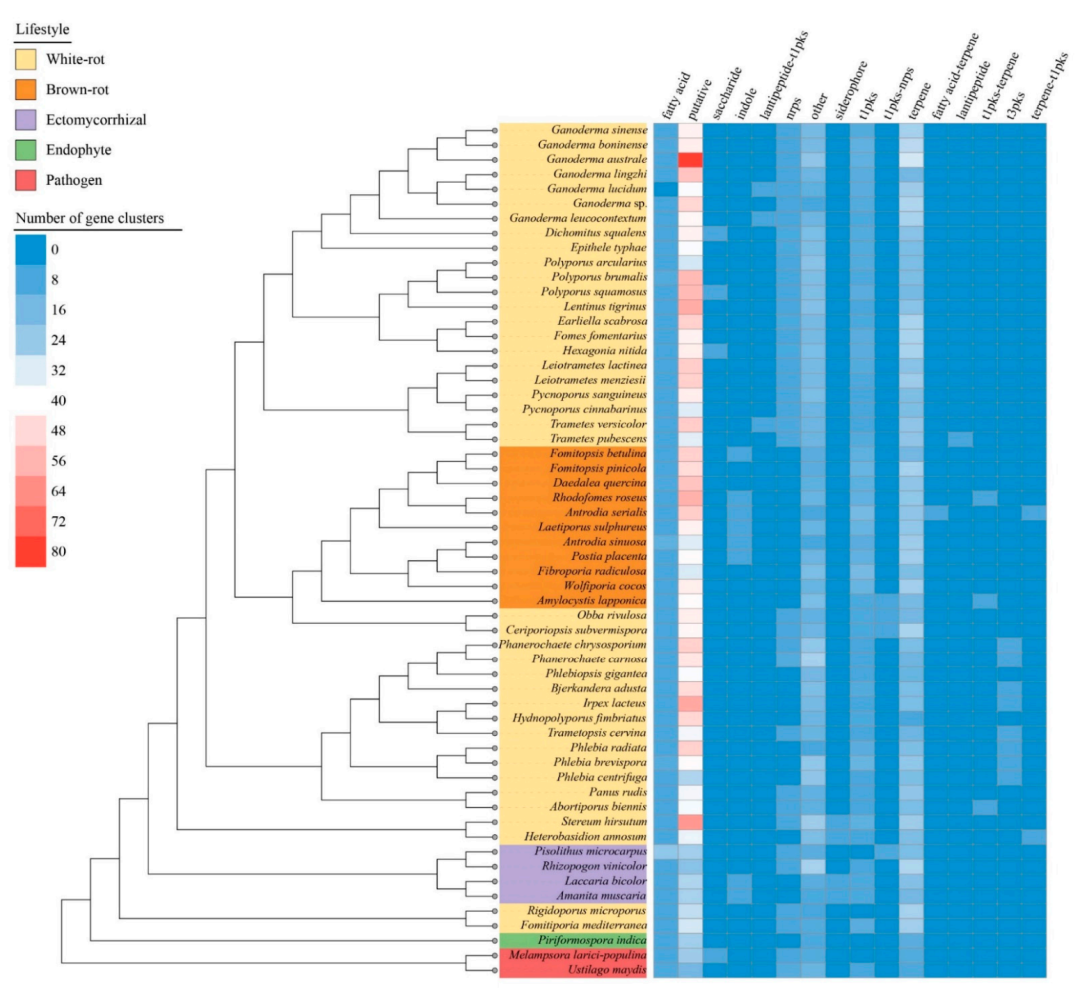
圖6 次生代謝產物生物合成相關基因簇豐度與物種系統發育之間的關系
Pfam蛋白結構域分析
本次分析確定了超過5000個Pfam蛋白結構域,根據基因拷貝數(>100)共對32個Pfam類進行分類。澳洲靈芝的Pfam蛋白結構域數較高,約為墨西哥黑穗病菌(Ustilago maydis)的5倍(864個蛋白結構域)。G.australe中有13個顯著的蛋白結構域,包括參與SM的細胞色素p450、與植物不親和性(VI)有關的異核體不親和性蛋白和主要促進子超家族的膜轉運蛋白。此外,其他推測在表觀遺傳調控中發揮作用的蛋白結構域和蛋白-蛋白相互作用也在G.australe中含量比較近豐富。與其他靈芝品種相比,G.boninense和G.leucocontextum分別編碼了額外的蛋白結構域,這些蛋白結構域僅存在于寄生真菌和共生真菌中。蛋白激酶結構域蛋白-蛋白相互作用結構域在外生菌根真菌中特別富集。
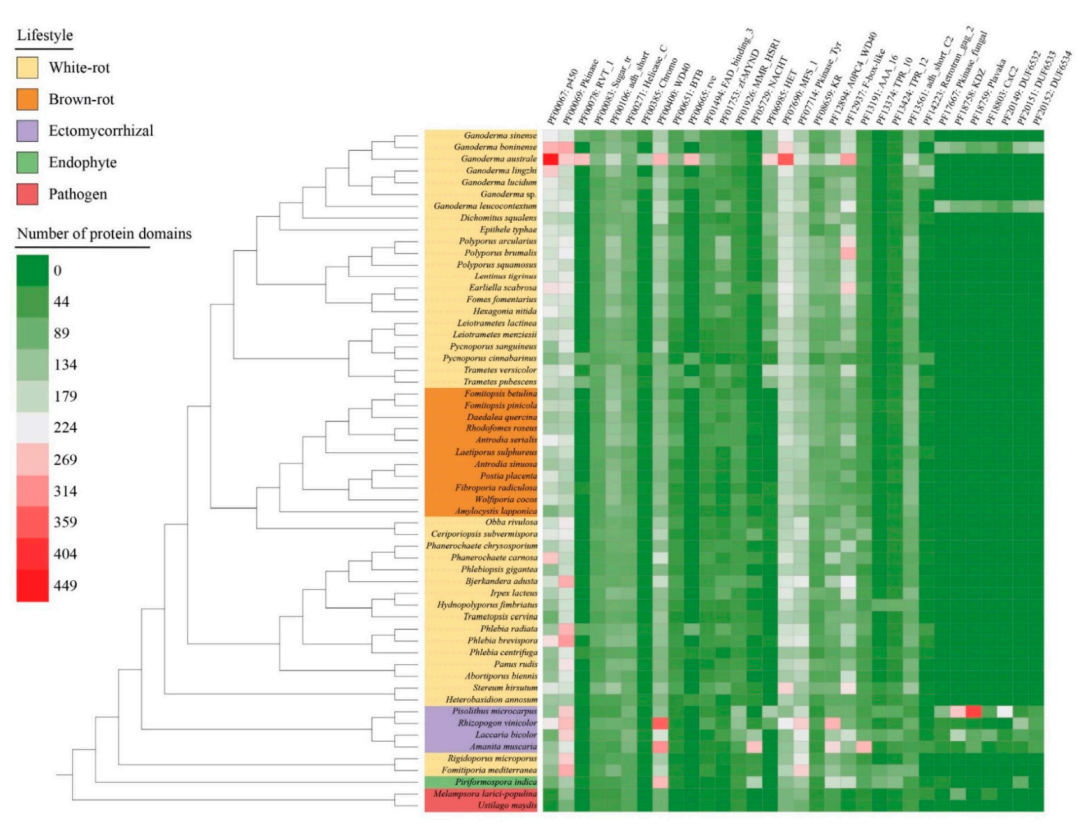
圖7 58種植物中前100個Pfam蛋白結構域的分布和豐度
分泌相關基因分析
在58種入選真菌中,靈芝的分泌量最大。小分泌蛋白(SSPs)占總分泌蛋白的41% ~ 81%,其中4% ~ 28%的SSPs被標記為CAZymes、脂肪酶或蛋白酶。大多數已知的SSP被注釋為CAZymes,特別是在G. australe和G. lingzhi中。
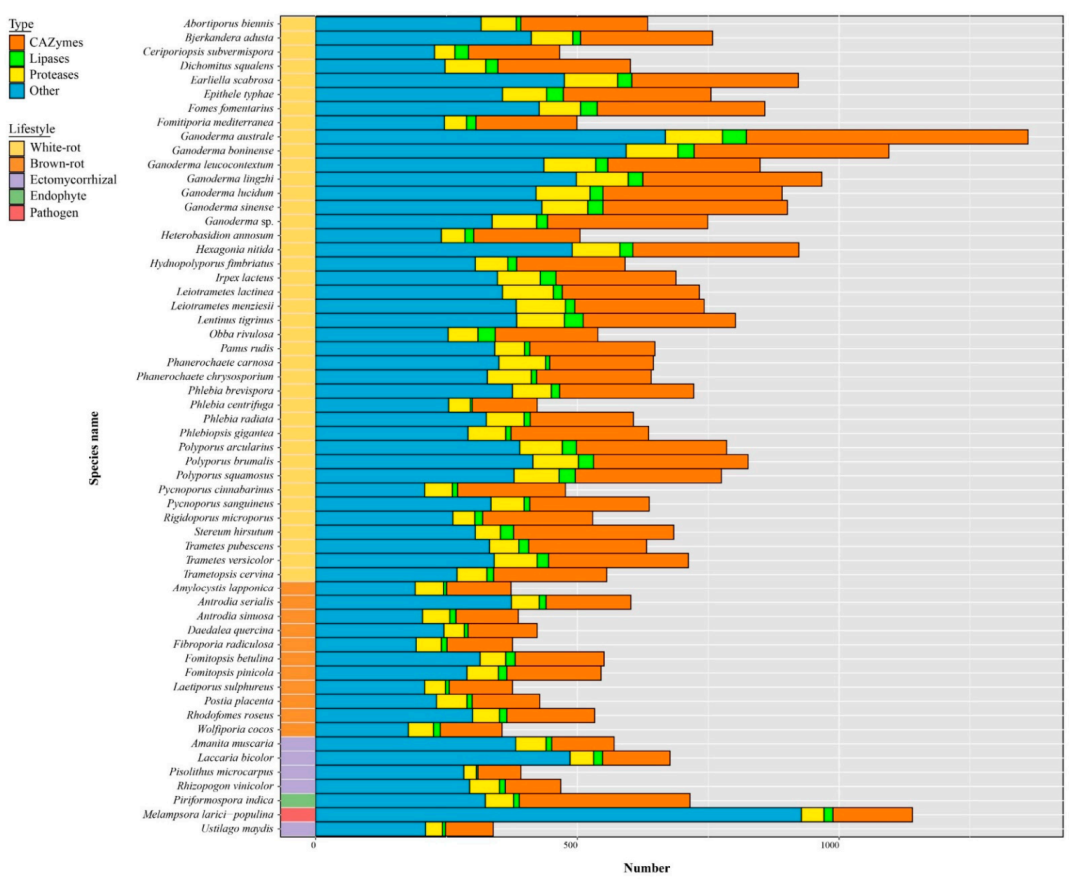
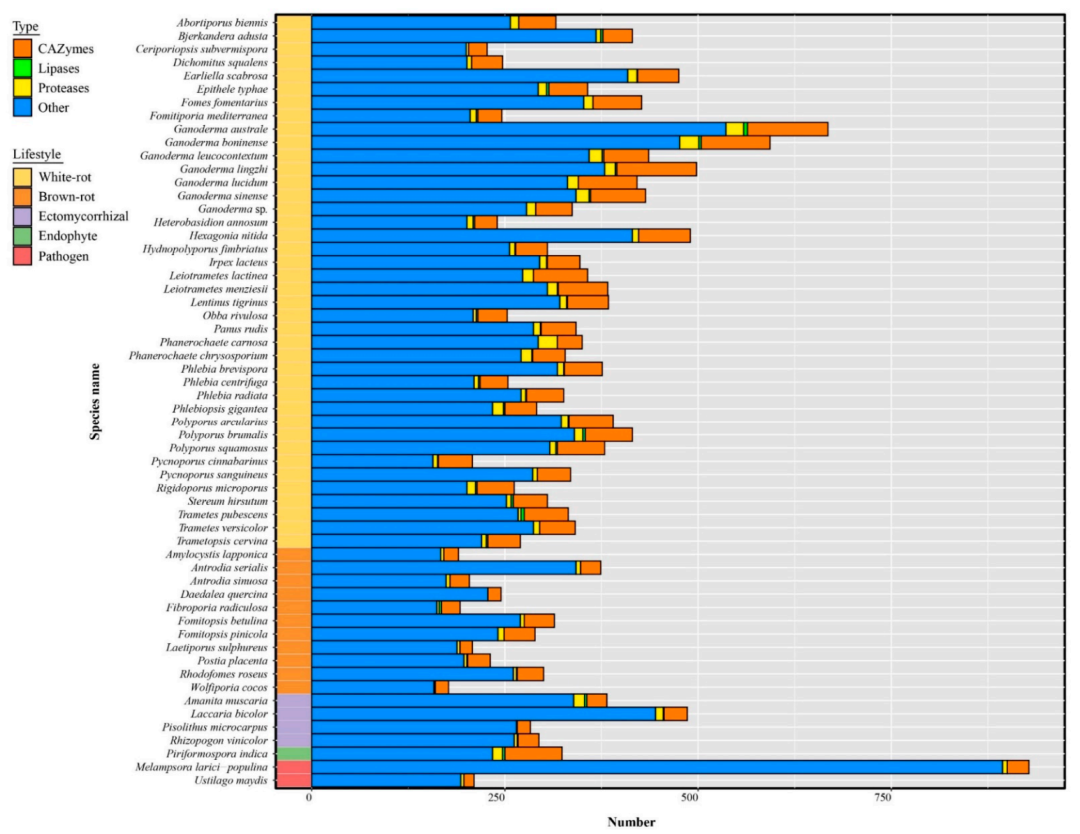
圖8 分泌蛋白和小分泌蛋白在靈芝屬和其它真菌中的分布
在多孔菌目的不同支系中,CAZyme的含量各不相同,靈芝在其核心的多孔菌支系中也有不同的CAZyme含量。由褐腐菌組成的前菌支分泌的CAZymes含量是Polyporales白腐菌的2倍。在具有分泌基因代表的148個CAZymes亞家族中,有48個家族的基因在褐腐菌中比在Polyporales中的白腐菌中數量少。CAZymes整體富集于核心的polyporoid分支,而非靜脈生物類分支。這種富集與AA1、AA14、CE1、EXPN、GH16、GH17、GH18、GH25和GH30家族的擴展有關。在其核心類群中,靈芝物種富集了CAZymes,并擴展了11個家族,包括GH18、GH16、AA1、GH43、CE16、GH3、GH128、GH47、GH115、GH25和GH1家族。而PL4在核心多孔類進化枝的其他物種中富集。與其他物種相比,G. australe分泌CAZymes的數量最多,其中GH18和EXPN家族含量較高,它還含有最多的PCWDEs、MCWDEs和作用于果膠、肽聚糖和幾丁質的酶。
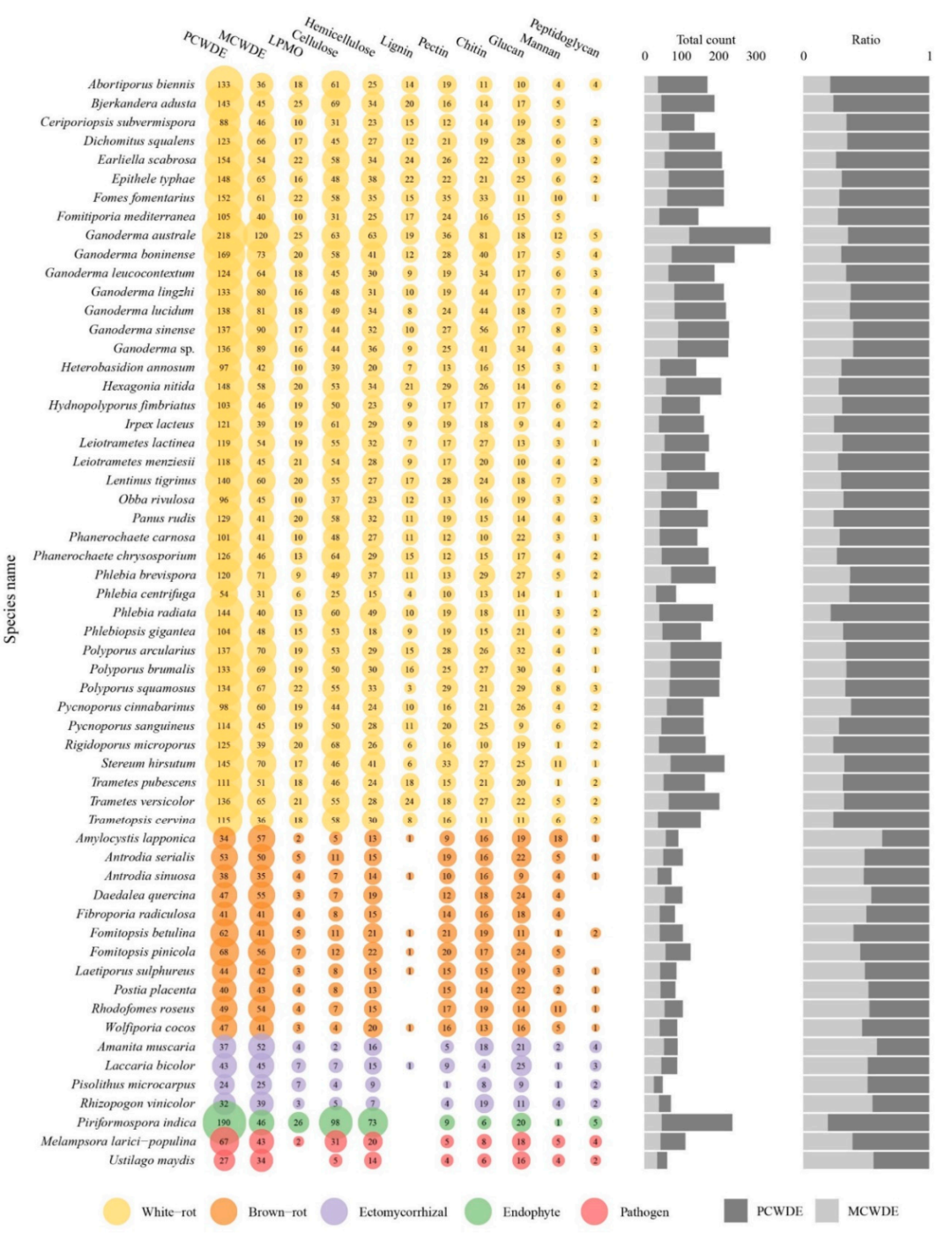
圖9 CAZYmes基因在靈芝屬和其它真菌中的分布
在58個真菌基因組中共鑒定出3880個編碼分泌蛋白酶的基因(在73個MEROPS家族中),8個蛋白酶家族,其中以A01A為代表的蛋白酶亞家族最多,有1332個蛋白。腐生菌與共生菌間無明顯差異,僅在白腐菌中發現5個蛋白酶家族(C40、M24X、M57、M77、S08B),且基因拷貝數較低。其中,在G.australe, G. leucocontextum, G. lingzhi和G. lucidum中發現了包含4個蛋白的M57蛋白酶家族。此外,靈芝屬的G01、M28E和M35蛋白酶含量較高。在分析的58種真菌分泌物中,G.australe的分泌脂肪酶數量最多,其中以GGGX脂肪酶最為豐富。
研究結論
1.對G. australe、G. leucocontextum和G. lingzh的基因組進行了測序,基因組大小為60.34 ~ 84.27 Mb,編碼15,007 ~ 20,460個基因。
2.對擔子菌科58種真菌(包括40株白腐真菌、11株褐腐真菌、4株外生菌根真菌、1株內生真菌和2株病原菌)的143個單拷貝基因進行了系統基因組學分析,證實了靈芝屬心多孔孔類分支。
3.與其他選擇的物種相比,靈芝物種的基因組編碼了更多的萜類代謝和分泌蛋白(CAZymes、脂肪酶、蛋白酶和SSPs)編碼的基因。
4.G. australe具有最大的基因組大小,沒有明顯的全基因組重復,但顯示轉座因子(TEs)擴增和最大的萜類基因簇,表明其生產藥用萜類化合物的能力很高。G. australe還編碼了含有細胞色素p450、異核體不相容和主要促進因子家族結構域的最大一組蛋白。此外,G.australe分泌組的大小最大,可能在宿主定植過程中增強細胞壁碳水化合物、蛋白質和脂肪的分解代謝能力。
5.本研究為進一步表征這一重要分解者群的生態特征提供了基礎資料,目前的基因組資源將用于進一步開發生物技術和醫藥應用,以及靈芝物種的生態學研究。
本研究的denovo測序和部分數據分析由上海派森諾生物科技有限公司完成。如需進一步討論,歡迎發郵件或者致電我們喲(郵箱地址:[email protected],聯系電話:025-58185663-836)!
文章索引:
Sun, Y.-F.; Lebreton, A.; Xing, J.-H.; Fang, Y.-X.; Si, J.; Morin, E.; Miyauchi, S.; Drula, E.; Ahrendt, S.; Cobaugh, K.; Lipzen, A.; Koriabine, M.; Riley, R.; Kohler, A.; Barry, K.; Henrissat, B.; Grigoriev, I.V.; Martin, F.M.; Cui, B.-K. Phylogenomics and Comparative Genomics Highlight Specific Genetic Features in Ganoderma Species. J. Fungi 2022, 8, 311. https://doi.org/10.3390/jof803031