
2021-12-14

期刊:《Frontiers in Microbiology》
影響因子:5.640
近日,派森諾生物與福建醫科大學附屬協和醫院李彬老師課題組合作,在微生物基因組領域的《Frontiers in Microbiology》發表研究成果!本研究針對大腸桿菌的遺傳多樣性和系統發育進行研究,揭示了大腸桿菌 ST1193 在中國福州和非中國地區傳播的不同進化軌跡,進一步支持了在特定引入地理區域之后該譜系的全球傳輸和本地化譜系擴展。
研究背景
大腸桿菌(E.coli)是最常見的革蘭氏陰性細菌性條件致病菌,它主要棲息于人類和其他溫血動物的下腸道,在臨床和流行病學方面都有極大的挑戰。E.coli可導致多種臨床感染,包括腹瀉、無并發癥的尿路感染和敗血癥等,每年導致全球超過200萬人死亡。
在過去的 20 年中,由于E.coli產生超廣譜β-內酰胺酶,對氟喹諾酮類和超廣譜頭孢菌素的耐藥性迅速增加,而這種耐藥性增加與特定克隆--ST131菌株在全球范圍內的傳播有關。有研究表明,E.coli ST131在耐氟喹諾酮類E.coli菌株中占近一半,有趣的是,E.coli種群結構是動態的。E.coli ST1193是一種新的序列類型,被報道為一種新興的耐氟喹諾酮類 (FQ-R) 和有毒的大腸桿菌譜系。該類型菌株通常從動物和人類中分離出來,會導致各種腸外感染,如敗血癥、尿路感染和腦膜炎等。此外,該譜系的占比在 2010-2017 年期間從 4.4% 急劇上升至 22.2%,其增長頻率比 ST131 譜系飆升了五倍,直到現在E.coli ST1193類型菌株成為繼E.coli ST131-H30之后的第二個大克隆群。
近年來關于E.coli ST1193的流行病學的研究非常多,但是對這個譜系的進化過程和差異的了解較少。本研究旨在使用全基因組測序檢測E.coli ST1193 譜系的菌株水平遺傳多樣性和系統發育,并在全球范圍內深入了解該譜系,調查與抗菌素耐藥性 (AMR) 相關的遺傳元件的存在可能會給這種新興細菌病原體帶來選擇性優勢。
研究材料與方法
1、實驗材料
本研究共計涉及76個 ST1193 菌株,其中51株是在中國福建醫科大學協和醫院分離得到的ST1193 類型菌株,25株從Enterobase數據庫中下載。
2、測序平臺
Illumina NovaSeq
3、分析內容
細菌框架圖測序、耐藥&毒力基因分析、核心基因組和輔助基因組分析、核心基因組Indel鑒定、藥敏實驗等。
研究結果
本研究共包括從中國福州和非中國地區的人類中收集的 76 個E.coli ST1193 類型,具有fimH64、filCH5和fumC14 等位基因,其中94.7% (72/76) 的E.coli ST1193 為O型O75。其余四個分離株無法分型。在這項研究中發現了兩種類型的膠囊變體(K1 和 K5),其中7株(9.33%)E.coli ST1193具有K5莢膜基因型,68株(90.67%)具有K1莢膜基因型。一個分離株既沒有 K1 莢膜基因型,也沒有 K5 莢膜基因型。具有K5莢膜的非中國大腸桿菌ST1193為16.67%(4/24),比例高于從中國福州采集的分離株(5.88%,3/51)。對76株菌進行CRISPR預測,并未發現任何一株菌具有潛在的CRISPR結構。
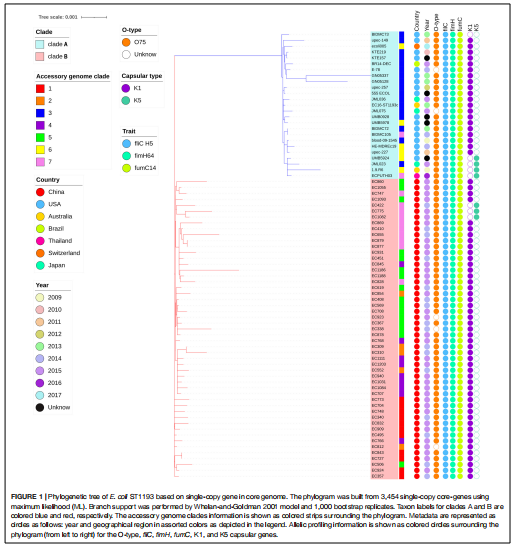
圖1 76株菌的核心基因組系統發育樹
基于檢測到的 3,454 個單拷貝核心基因構建核心基因組系統發育樹,結果顯示所有E.coli ST1193 被分為兩個進化枝(進化枝 A 和 B):進化枝 A 包括 25 個非中國E.coli ST1193,進化枝B含有來自中國福州的所有分離株。同時,B分枝菌株比整個世界范圍內的集合更加多樣化。為了進一步探討核心基因組進化樹中不同分支的差異,比較基因組學結果表明,共在150個分支特異基因中發現了Indel,對這些特異基因進行GO富集分析發現超過70%的基因功能屬于生物過程和分子功能中。除此之外,本研究對1134個輔助基因進行了附屬基因組進化樹構建,結果得到了7個不同的分支,將附屬基因組簇與核心基因組進化樹進行比較,結果發現附屬基因組簇和核心基因組進化高度相似,揭示了從中國福州分離得到的51株E.coli ST1193型菌株具有較高的遺傳多樣性。
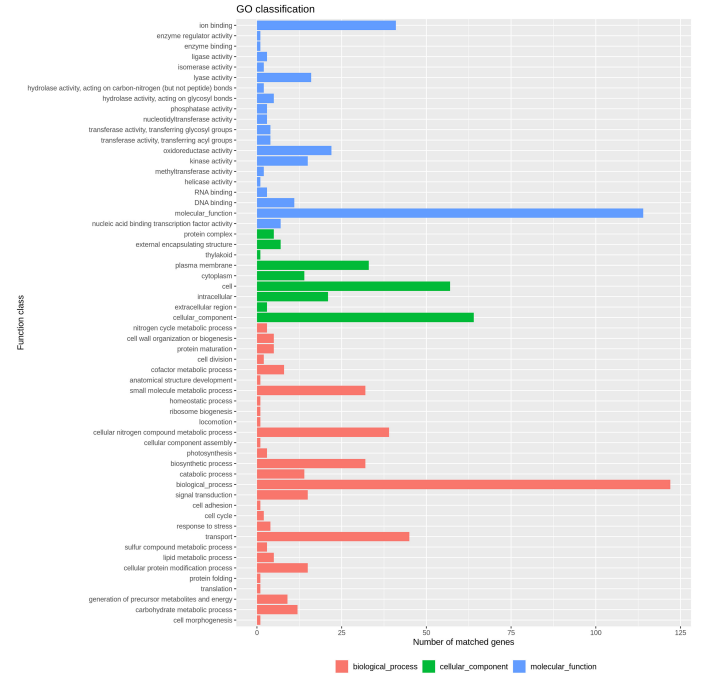
圖2 GO富集分析
根據毒力基因(VF)分析發現,不同分離株間的VFs分布存在差異。70%的E.coli ST1193基因組具有毒力基因,如外膜血紅素受體基因chuA(76/76, 100%)、鐵載體受體基因fyuA(76/76, 100%)、菌毛蛋白基因yfcV(76 /76, 100%), 空泡自轉運基因 vat (76/76, 100%), 分泌自轉運基因 sat (72/76, 94.7%), IrgA 同源粘附素基因 iha (71/76, 93.4%), 谷氨酸酶基因 decar gad (70/76, 92.1%) 和質粒攜帶的腸毒素基因 senB (58/76, 76.3%)等,這些基因均可以在所有地理區域中檢測到。值得注意的是,進化枝 A(非中國E.coli ST1193 分離株)和進化枝 B(來自中國福州的分離株)之間的 VF 存在顯著差異,包括 cia、neuC、gad 和 traT。
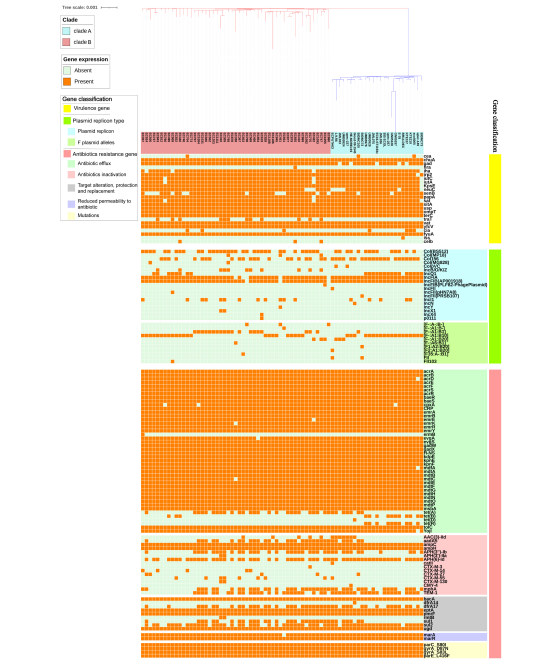
圖3 76株 E.coli ST1193型菌株毒力因子、質粒復制子類型和耐藥基因熱圖
質粒復制子類型
對所有菌株進行質粒復制子類型分析,結果表明幾乎所有的分離株(75/76,98.7%)都至少攜帶一種質粒復制子類型,其中包含19種一致的質粒不親和類型。E.coli ST1193包含兩種F型復制子(IncFIA和IncFIB),而IncFII復制子僅在3株菌株中檢測到。本研究基于質粒F等位基因進行質粒MLST分型, 結果顯示 IncFIA 等位基因 (A1) 在E.coli ST1193 中高度流行;IncFIB 等位基因是多樣的,并且鑒定了 IncFIB 的四種不同等位基因(B-、B1、B10 和 B20);Col (BS512) 和 Col156 在本研究中所有地理區域的分離株中高度流行。
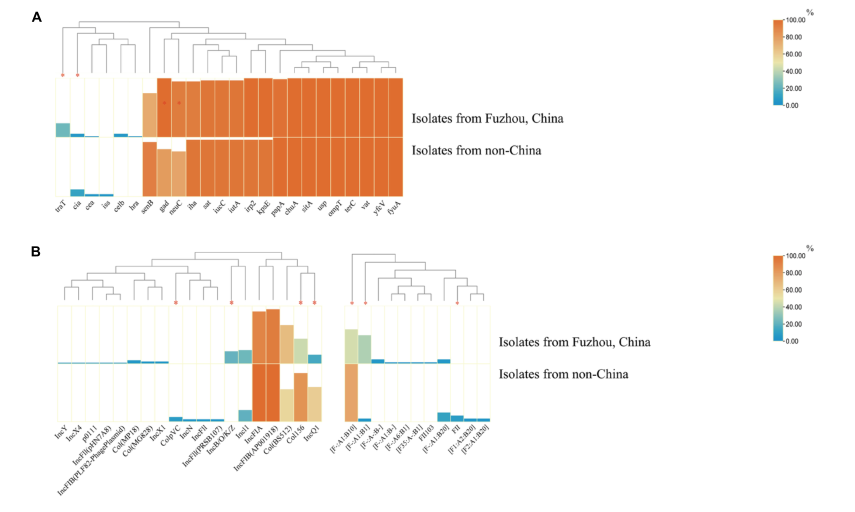
圖4 福州和非中國區分離株的毒力因子和質粒復制子類型比較
此外,進化枝 A(非中國大腸桿菌 ST1193 分離株)和進化枝 B(來自中國福州的分離株)之間攜帶的質粒復制子類型存在顯著差異。IncQ1 在 60.9% 的非中國E.coli ST1193 中被鑒定,但僅在來自中國福州的 39.1% 分離株中被發現(p < 0.05)。Col156 在 84.0% 的非中國E.coli ST1193 中檢測到,但在中國福州的分離株中僅發現 43.2%。IncB/O/K/Z 常見于來自中國福州的E.coli ST1193 (11/51, 21.6%),在非中國分離株中未檢測到 (0/25, 0%)。
抗生素抗性基因
在本研究中,所有E.coli ST1193型菌株的關鍵基因 parC (S80I)、parE (L416F) 和 gyrA (D87N 和 S83L) 中都包含相同的四個非同義突變,而這四個看家基因的突變會導致氟喹諾酮抗性。在76株E.coli ST1193菌株中均存在一些抗生素外排基因和與抗生素滲透性降低相關的基因,超過50%的分離株包含獲得性耐藥相關基因,如blaTEM-B(60.5%)、sul2(77.6%)、dfrA17(55.3%)等。而blaCTX-M-130 和 blaCTX-M-14僅在進化枝 A(非中國E.coli ST1193 分離株)中檢測到,tet(B) 和 tet(R)僅在進化枝 B(來自福州、中國)檢測到。此外,我們觀察到進化枝 A 和進化枝 B 之間攜帶的 ARG 存在顯著差異,包括 blaCTX-M-55、blaTEM-1、sul2、tet(B)、tet(R)、APH(6 )-Id 和 AAC(3)-IId等。
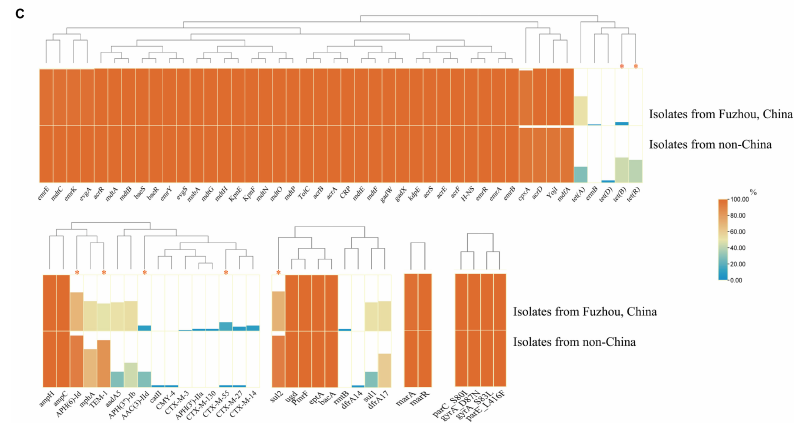
圖5 福州和非中國區分離株的抗生素抗性基因比較
blaCTX-M-55 通常在E.coli ST1193 中發現,可能在 ST1193 譜系的克隆擴增中發揮重要作用,這促使我們進一步研究該基因在ST1193中的遺傳環境。本研究在9個分離株中發現blaCTX-M-55基因,這些分離株均來自亞洲,其中 8 個來自中國福州,1 個來自泰國。從 blaCTX-M-55基因存在的質粒相關序列中,可以確定這些分離株和AMR基因相關。此外,發現 blaCTX-M-55和 IncI1 和/或FIB在 55.6% 分離株的同一contig中被鑒定出來。在所有9個分離株中,blaCTX-M-55位于 ORF477基因的下游。同時在blaCTX-M-55 的下游或上游檢測到不同的移動元件,包括 ISEc9、Tn3 和整合酶基因;而blaTEM-1位于中國福州的兩個分離株中 blaCTX-M-55的上游。
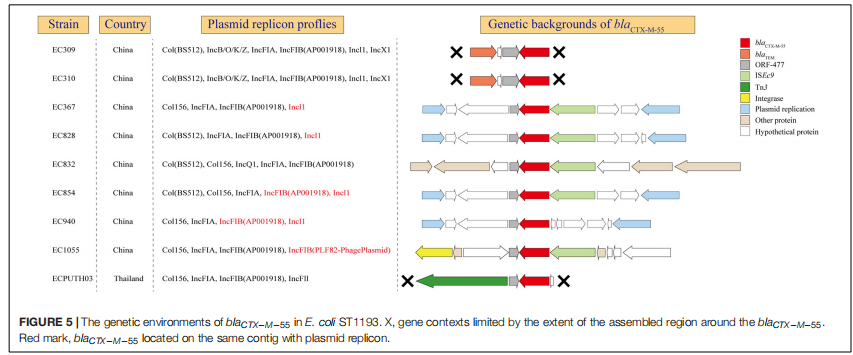
圖6 E.coli ST1193 中 blaCTX-M-55 的遺傳環境
研究結論
本研究為耐氟喹諾酮類藥E.coli ST1193菌株系統發育關系提供了重要依據。研究結果表明,E.coli ST1193在中國福州和非中國地區的傳播軌跡不同,其中中國福州分離株似乎比整個全球范圍內的更加多樣化。本結果是一個很好的引航分析,可以為進一步擴展分析提供一個起點。應進行戰略性和持續性監測,以防止E.coli ST1193 引起的感染。