2020-08-04

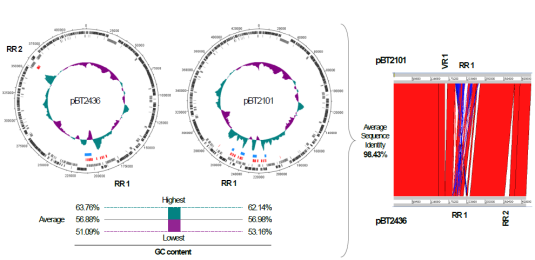
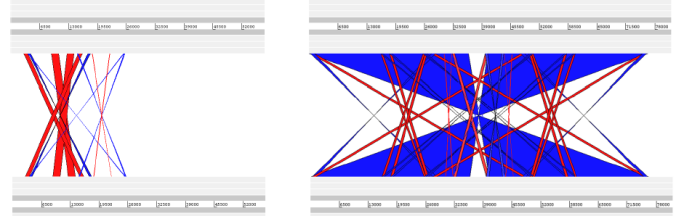
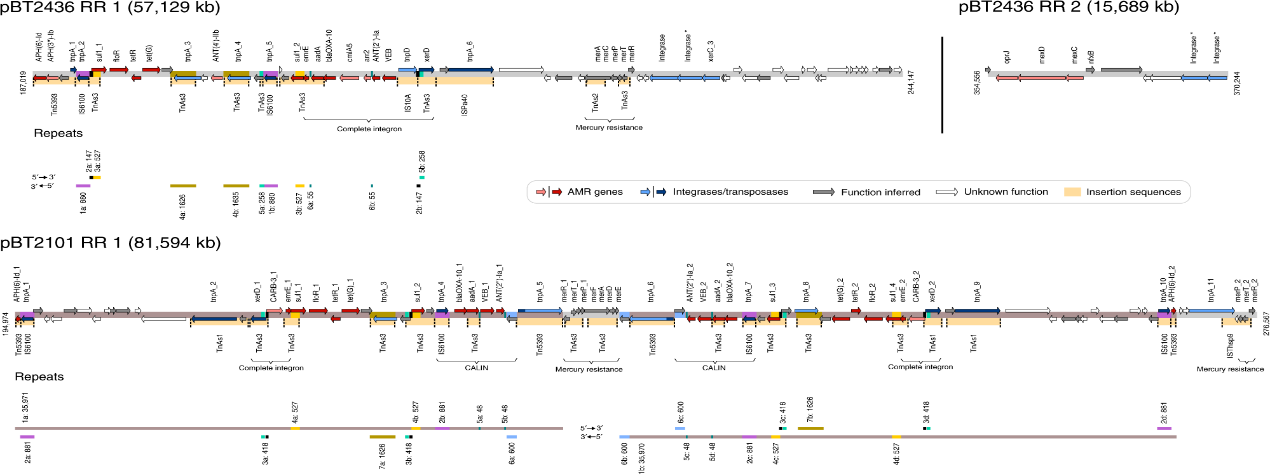
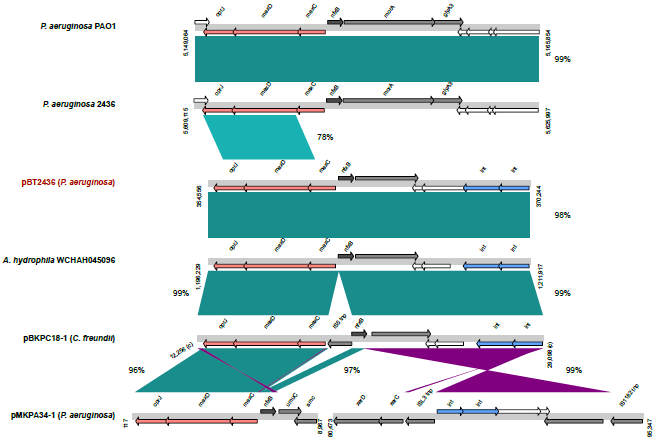
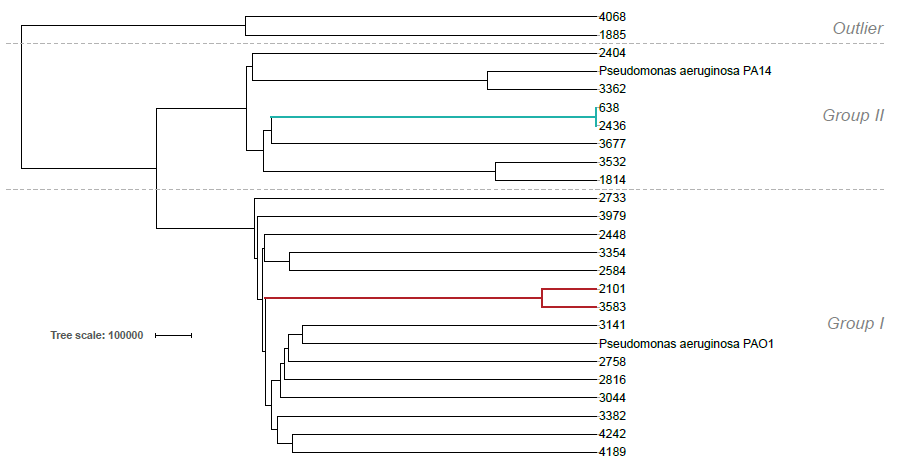
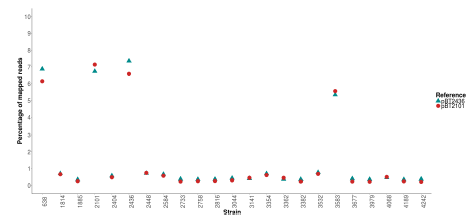
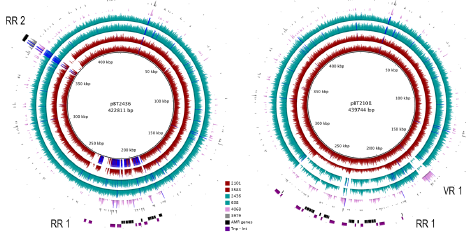
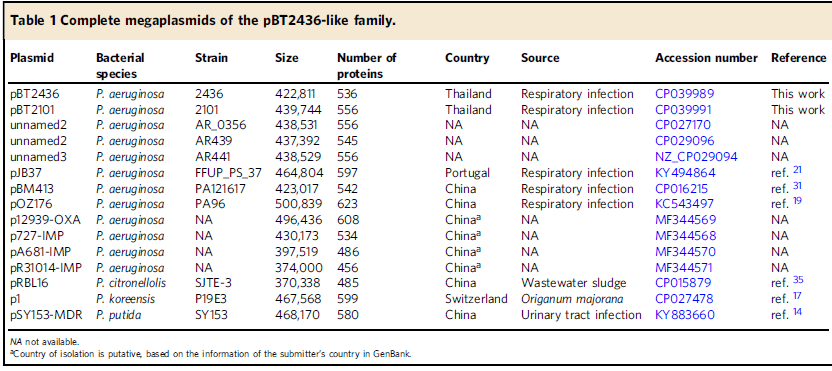
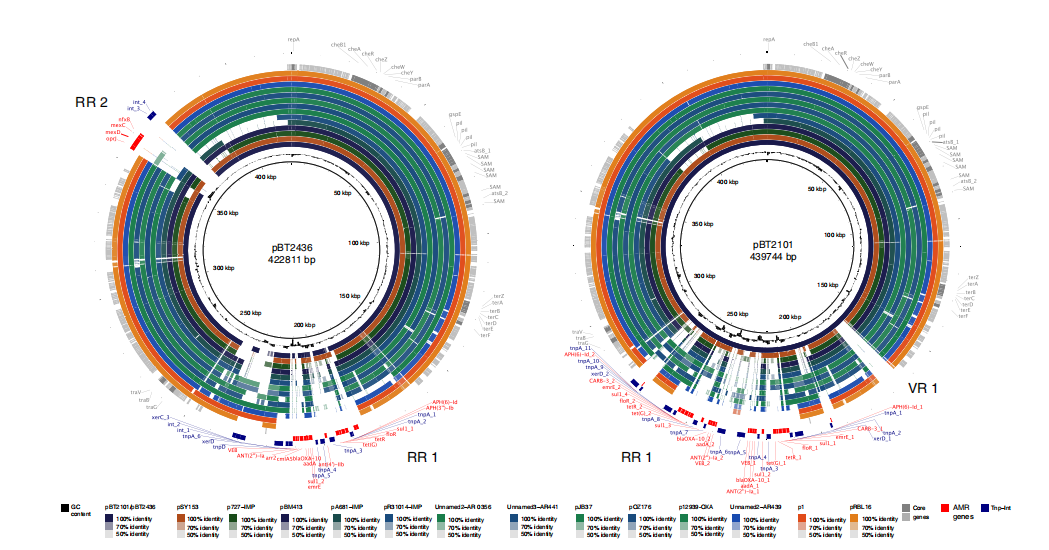
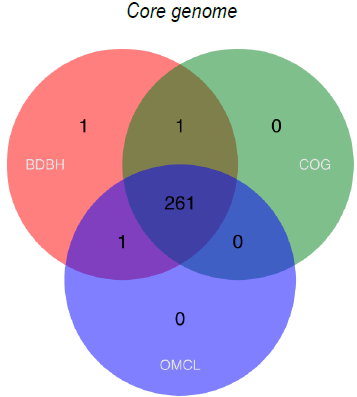
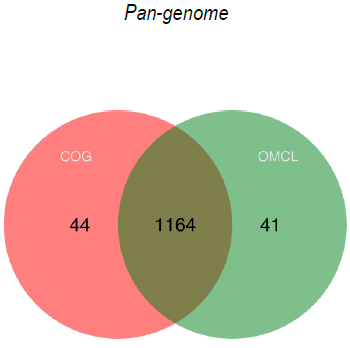
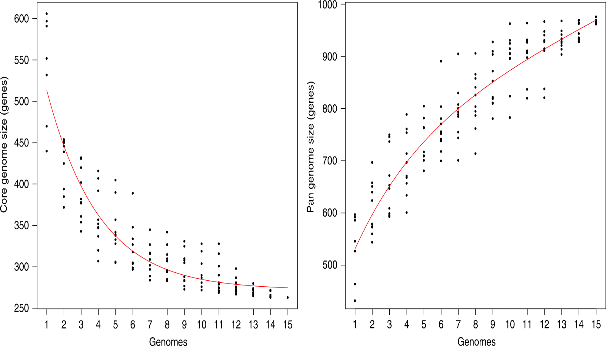
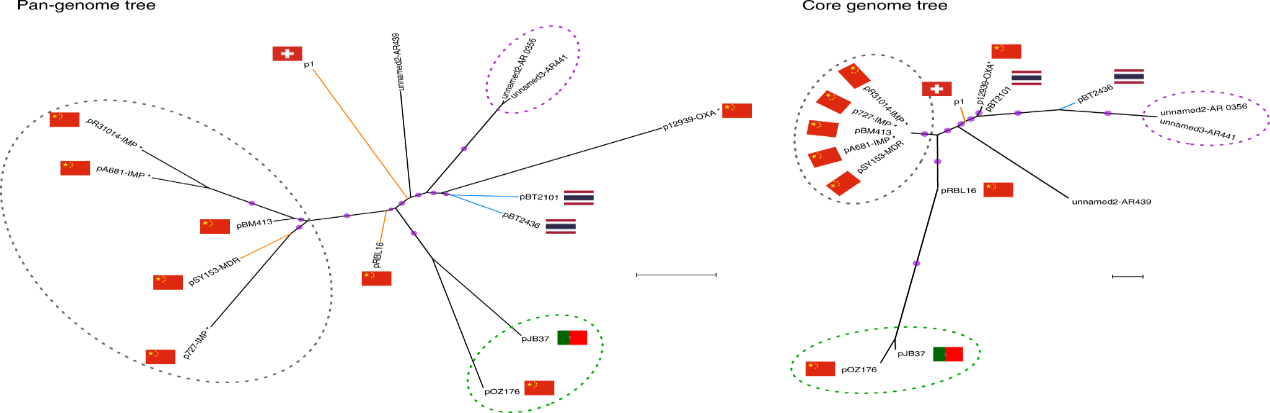
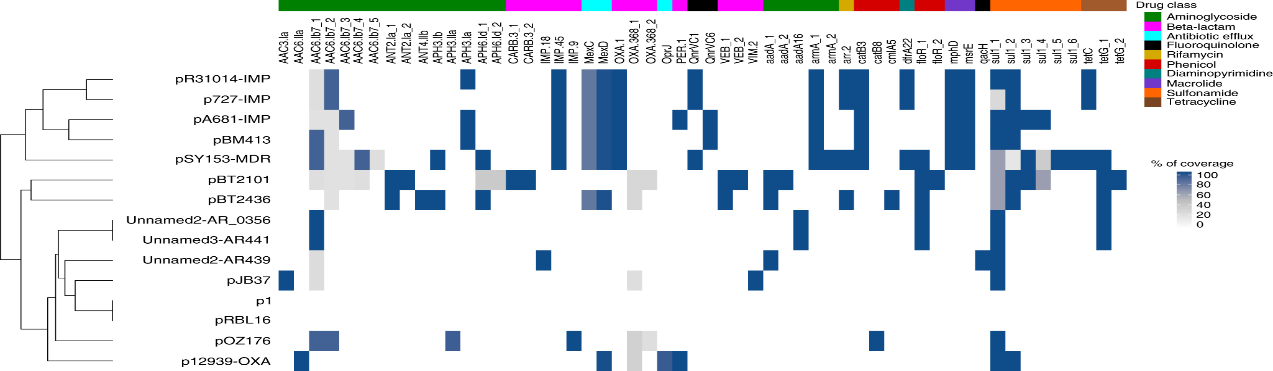
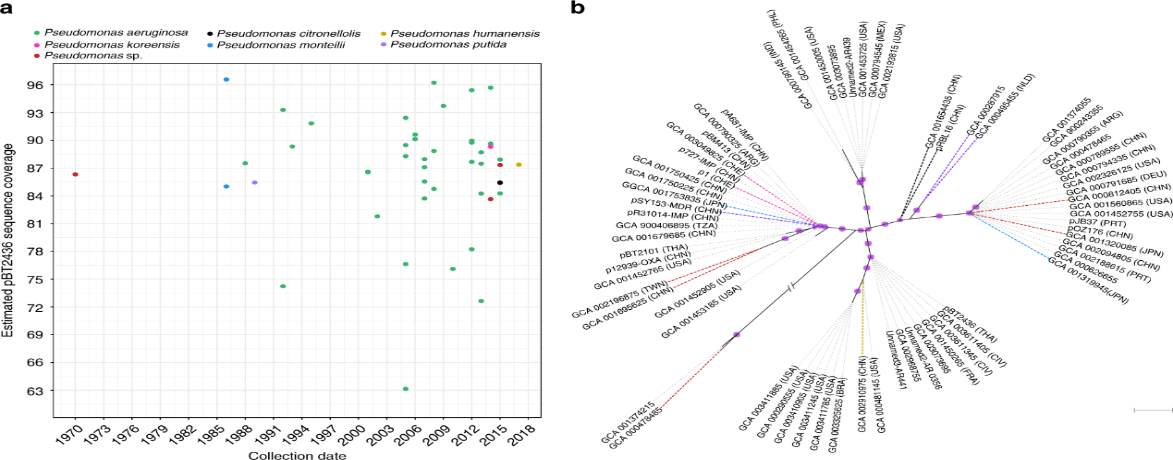
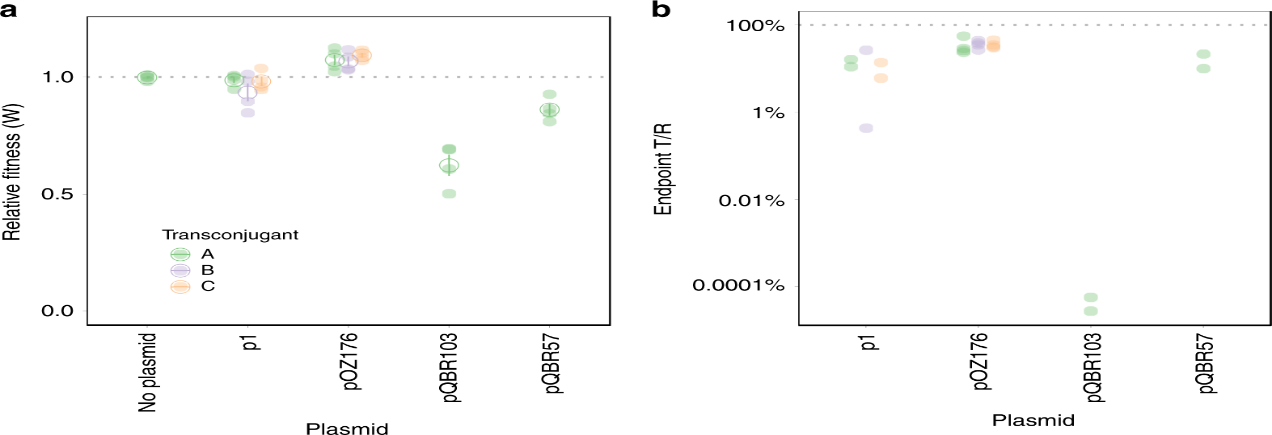
2020年6月《nature communications》期刊發表了一篇題為“A megaplasmid family driving dissemination of multidrug resistance in Pseudomonas”的文章,揭示了假單胞菌中的大質粒家族中的多重耐藥性的特性及相關大質粒的進化關系。 一、研究背景 抗生素的耐藥性(AMR)傳播被認為是對人類健康的一個關鍵的全球挑戰,ESKAPE病原體(糞腸球菌、金黃色葡萄球菌、肺炎克雷伯菌、鮑曼不動桿菌、銅綠假單胞菌和腸桿菌屬)是全世界醫院內感染的主要原因。特別是銅綠假單胞菌,該菌通常與多重耐藥性(MDR)相關,已被世界衛生組織列為與抗生素耐藥性相關的重要病原體。在泰國,銅綠假單胞菌對碳青霉烯的耐藥性是一個日益嚴重的問題,之前的研究試圖闡明在臨床分離的銅綠假單胞菌中碳青霉烯耐藥的遺傳基礎,但對質粒的作用知之甚少。本文采用二代+三代的測序方法對銅綠假單胞菌進行測序,獲得假單胞菌中與多重耐藥相關的質粒,通過比較基因組等分析研究其水平轉移能力以及進化關系等。 二、材料與方法 1、實驗材料 從泰國曼谷瑪希隆大學Ramathibodi醫院獲得48株與不同感染相關的分離菌株,對48株分離株進行藥敏實驗,根據結果將其分為敏感型、中間型以及抗性菌株。 2、測序方法及組裝 (1)二代:從48株中選取23株敏感型和抗性菌株進行Illumina Miseq測序,測序數據采用A5-Miseq進行組裝; (2)三代:選取抗性最強的3株進行Pacbio RSII測序,測序數據采用HAGP v.3進行組裝。 3、基因組注釋、預測及比較基因組分析 對測序獲得的序列進行基因組拼接組裝以及基因預測,對獲得的序列進行比較基因組分析。 三、實驗結果與分析 1、臨床分離株的抗菌藥敏性 對收集到的48株與不同感染類型相關的銅綠假單胞菌分離株進行藥敏實驗,其中有9株對5種抗生素全部具有抗性,有6株對5種抗生素中的4種具有抗性。 2. pBT2436和pBT2101 抗生素耐藥性研究 選取抗性最強的3株分離株2101、2436和4068進行Pacbio測序,2101和2436分別得到了一條完整染色體(6,782,092bp、6,573,638bp)和一個環形質粒(命名為pBT2101(423kb)和pBT2436(440kb))。對這兩個質粒進行比較發現,pBT2101有一個重復區域(RR1)和一個可變區(VR1),pBT2436有兩個重復區(RR1和RR2),但沒有可變區。其中分離株4068組成5條contig,有一個51kb的質粒但是不含有抗性基因,故本文未做進一步研究。 對pBT2101和pBT2436進行注釋,發現RR1區含有大量的AMR基因,包括一些共有的和特有的mer操縱子、轉座酶、整合酶、重復序列,但在核苷酸水平上基因含量、序列同一性及排列位置上存在差異;同時轉座子插入導致一部分基因長度發生變化從而導致共有區域存在一定的差異。對兩個質粒進行IS序列預測,得到8種IS序列類型,其中Tn5393,IS6100和TnAs3為兩者共有的,預測得到的序列中包含一部分抗性基因,這表明IS序列在獲得不同來源的抗性中起關鍵作用。 將pBT2436染色體進行比對發現,與菌株PAO1具有相同的染色體序列,菌株2436、PAO1與pBT2436的RR2區比對僅有78%的相似性,排除RR2區來源于菌株染色體;將RR2區序列在數據庫中進行blast,發現同源性最高的為A. hydrophila WCHAH045096,與非相關質粒pBKPC18-1(C.freundii) 和 pMKPA34-1(P. aeruginosa)相似性也比較高,主要區別是存在與各種IS相關的基因,暗示了這些基因在傳播中的作用。這些抗性區的動態性質與其余質粒主鏈的一般保守性相反,表明抗性基因已在不同質粒背景中獨立組裝。 3. 臨床分離株中相關大質粒的分布 為了確定相關質粒是否存在于同一家醫院的其他P. aeruginosa中,選擇了23種耐藥性較高和耐藥性較低的臨床分離株(包括2101和2436)進行二代測序。從基因組中提取的多基因組序列類型(MLST)和基于kmer序列比較表明,分離株在遺傳上高度多樣化。 以pBT2101和pBT2436為參考基因組對測序分離株進行mapping,分離株3583和638分別與pBT2101和pBT2436保持高度相似性,表明這兩個分離株中存在相關質粒。其中分離株2436和638來源于同一患者但是不同來源的樣品中分離得到的,它們具有相同的序列(ST);來源于不同患者的分離株2101和3583在7個MLST基因座中的6個具有相同的序列,它們的質粒具有很高的一致性,但分離株3583中的質粒僅攜帶一個抗性基因拷貝而不是兩個。 4. pBT2436-like大質粒家族 為了更好的研究與多藥耐藥性質粒相關的大質粒家族,以pBT2436和pBT2101序列在NCBI數據庫中進行blast,結果得到13種其他大質粒,大小在370至501 kb之間,編碼基因在456至623之間。其中10個大質粒屬于P. aeruginosa,另外三個屬于P. putida(人的尿路感染有關), P. citronellolis 和 P. koreensis。 對15個完整大質粒序列的比較分析顯示,它們具有較高的同義度和廣泛的序列相似性,檢測到的變異分布在整個基因組的離散位點上(主要集中在富含AMR基因、轉座酶和整合酶基因的大區域),但大質粒組的任何其他成員都不存在pBT2101 的7.7kb可變區(對VR1進行注釋發現該去并無AMR基因)。 5、pBT2436-like大質粒的核心和輔助基因組分析 基于15個質粒基因組進行比較得到261個直系同源蛋白質組組成的核心基因組,平均每個質粒約有48%的核心基因組(范圍為42–57%),泛基因組分析得到1164個共有基因家族。核心基因組和泛基因組的稀釋和積累曲線表明,隨著基因組數量的增加其核心基因組的數量不會受到太大影響。 6、 pBT2436-like大質粒家族的系統發育分析 基于(a)核心基因的核苷酸組成(n = 105)和(b)泛基因組(存在或不存在蛋白質簇)構建系統發育樹,發現: (1)在中國分離得到5個巨質粒(包括P.putida); (2)兩個大質粒pOZ176(來自中國)和pJB37(來自葡萄牙); (3)兩個了解很少的大質粒(菌株AR_0356和AR441)分別聚集成簇。 pBT2436和pBT2101通過基因組分析比通過核心基因組分析更緊密地聚集在一起,這表明由于加入了輔助基因(與AMR相關)而導致了一些融合。 根據AMR基因含量繪制熱圖,發現: (1)在非銅綠假單胞菌(p1和pRBL16)中發現的兩個巨質粒來自于非人類來源,不具有AMR基因; (2)質粒pST153從中國一例泌尿系感染菌株P. putida 中分離得到的,與其他來源于中國的質粒在AMR基因上具有一定的相似性。 7、pBT2436-like巨質粒家族的廣泛分布 為了確定大質粒家族的廣泛分布,將pBT2436序列放到NCBI上檢索假單胞菌基因組中相關的大質粒,從比對上的基因組中獲取其原始數據。以pBT2436序列為參考基因組進行mapping,發現mapping率最高為96.6%,是1986年從日本分離得到的P.montelii中的質粒;比對上最早的是1970年從日本發現的P. aeruginosa,mapping率為86.3%。從核心基因中選擇4個核心基因進行系統發育樹構建,揭示了新的多樣性模式。值得注意的是,只要4個系統發育標記基因來推斷樹,表明這些基因可以形成一個分型系統的基礎。 8、大質粒的穩定性和適應性成本 通過使宿主細菌在非選擇性培養基中循環生長三輪(約60代)并篩選維持妥布霉素抗性的方法來檢測pBT2436的穩定性,結果并沒有抗性喪失。為了解大質粒獲取的適應性進行競爭性適應性檢測,在檢測中帶有大質粒的P.fluorescens直接與等基因的、標記不同的無質粒競爭者競爭。令人驚訝的是,盡管pOZ176和p1的規模很大,但它們都沒有對競爭增長造成顯著的成本;但檢測到兩種質粒均大量轉移至競爭者中,并且未檢測到pOZ176或p1的丟失。這些實驗數據表明,由于適應性成本低,傳播速率高和基因含量高,pBT2436-like大質粒可能是環境中假單胞菌之間傳播抗藥性的有力載體,這與基因組分析結果一致。 四、結論 1.獲得了兩個緊密相關的大質粒(> 420 kb)的完整序列,攜帶著位于離散、復雜和動態耐藥區域的大量抗生素耐藥基因,并揭示了廣泛的復制和重組事件。 2.全面的基因組學和系統基因組學分析表明: 1)大質粒中包含假單胞菌不同成員中新出現的家族屬,并與多種來源(地理,臨床或環境)相關; 2)大質粒編碼各種適應生態位的輔助性狀,包括多藥耐藥性; 3)大質粒家族的輔助基因組具有高度的靈活性和多樣性 3.對可用數據庫的分析中可以推斷出大質粒家族的歷史,表明攜帶多個抗性基因的成員至少可以追溯到1970年代。 點擊閱讀全文:https://www.nature.com/articles/s41467-020-15081-7