
2020-07-21
群體進化前面兩期帶大家實戰了基于群體SNP的PCA和進化樹分析(點擊查看),我們今天帶大家進行實戰群體遺傳結構分析,依然含分析和繪圖代碼哦。
所謂群體遺傳結構(Structure)是指遺傳變異在物種或群體中的一種非隨機分布。按照地理分布或其他劃分標準可將一個群體劃分為若干亞群,處于同一亞群內的不同個體親緣關系較高,而亞群與亞群之間的個體則親緣關系稍遠。群體結構分析有助于理解進化過程,并且可以通過基因型和表型的關聯研究確定個體所屬的亞群。
群體進化研究的“三駕馬車”中通過PCA和進化樹分析可以直觀了解群體中個體間的分類關系。但如果我們要探究:
目標群體劃分為幾個亞群更合理?
直接按照地域劃分的群體分組是否合理?
群體間是否存在基因交流?
以及每個個體混血程度是多少?
這些疑問PCA圖和進化樹樹形圖都無法告訴我們答案,這時候就該高大上的Structure分析的遺傳結構圖出場啦!

圖1 遺傳結構分析結果圖
雖然圖形很高大上但看到花花綠綠的條形柱圖你是不是也心存疑惑,這張圖代表什么生物學意義呢?怎么解答前面的提出來疑問呢?接下來我們先幫大家一一解惑。
在進行Structure分析時我們只是獲得了樣本的基因型,并不知道這個群體實際包含幾個亞群。把群體的亞群數稱為K值,可以先預設群體亞群數等于1~n,即K=1~n,然后模擬在K=x的情況下,通過貝葉斯算法推算群體是如何分群以及每個個體的祖先來源。最后再根據每次模擬的最大似然值,找出劃分這個群體的最佳K值。
具體解讀以下面圖2為例做詳細的說明。
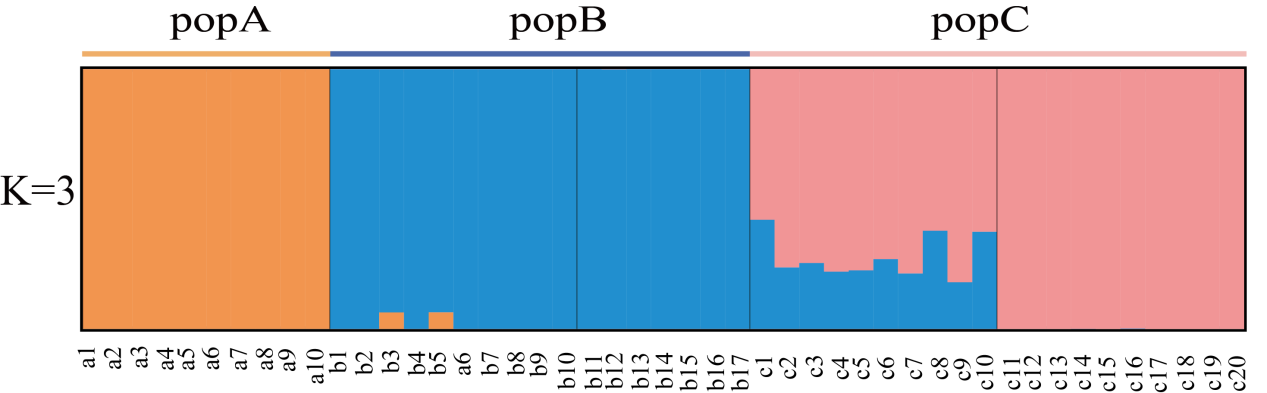
圖2 K=3時群體遺傳結構分布圖
圖2就是假設群體結構數的先驗值K=3的模擬結果。每個直方柱子代表1個樣本(這里popA包含10個樣本,樣本名a1~a10,popB包含17個樣本,樣本名b1~b17,popC包含20個樣本,樣本名c1~c20),柱子的顏色以及顏色比例代表這個樣本屬于哪個亞群以及祖先來源構成比例是多少,可以很直觀的通過顏色就可以判斷這47個個體是如何劃分為3個亞群體(橙、藍、粉)。同時也可以看出有些樣本的祖先來源單一(僅有1種顏色),有些樣本卻由2個顏色組成。例如樣本c1,它對應的直方柱子由兩種顏色構成,大概是37%的藍色和63%的粉色。說明這個個體很有可能是從兩個祖先亞群雜交而來,且兩個祖先亞群各占了37%和63%的血統。
但是每一個K值都對應著一個結果圖,哪一個K值才是最準確的呢?這里就要引出下面圖3的這張圖啦。
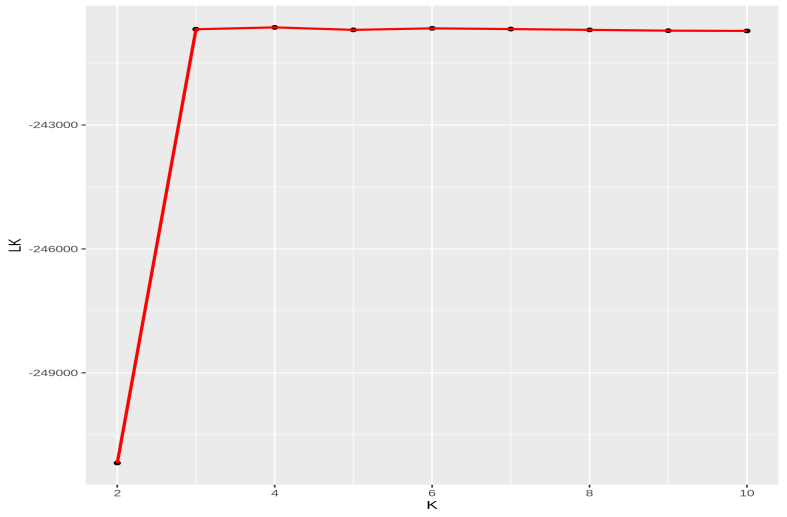
圖3 LK分布圖
每個K值基于貝葉斯模型的計算方法模擬的結果,都會產生對應的最大似然值(likelihood),LK值是取了自然對數后輸出的(ln likelihood)。ln likelihood越大,說明K值越接近于真實情況,但一般隨著K值升高,ln likelihood值也會進入平臺期。最優K值就是進入平臺期的那個拐點K值。
看到這里,大家是不是都想知道怎么才能實現群體遺傳結構分析呢?接下來我們就帶大家一起實戰遺傳結構分析。實戰示例見以下鏈接:
鏈接:https://pan.baidu.com/s/1o1gNmyrQE2AA94pa-xC-8w
提取碼:56bm
軟件Structure、FastStructure、Admixture都可以進行遺傳結構分析,Structure的運行速度較慢,Admixture使用的模型與Structure相同,但其憑借高速的運算速度逐漸成為群體遺傳結構分析的主流軟件,接下來就以軟件Admixture的使用方法為例帶大家逐步進行分析。
VCF文件格式轉換
利用軟件vcftools將群體SNP的vcf文件轉換成Plink可以分析的格式,具體轉換命令如下:
usage: vcftools --vcf test.vcf --plink --out test
##--vcf 輸入vcf文件
##--plink 將數據轉換為plink格式
##--out 輸出文件前綴
得到的結果文件test.map和test.bed。
SNP過濾
在Structure分析過程中需要滿足兩點,其一,各個亞群內部個體應該符合哈代-溫伯格平衡;其二,每個SNP位點是獨立的不連鎖的。一般基于重測序得到所有SNP作為Structure分析的輸入文件是不對的,需要利用Plink進行SNP的過濾,生成.bed文件,命令如下:
plink --noweb --file test --maf 0.05 --hwe 0.0001 --make-bed --out filter
##--noweb 不連接網絡
##--file 指定輸入文件
##--maf 過濾掉低于次等位基因頻率閾值的位點
##--hwe 過濾掉低于Hardy-Weinberg平衡檢驗p值低于閾值的位點
##--make-bed 數據轉換為二進制格式
##--out 輸出文件前綴
Admixture計算和K值的確定
K是假設的群體亞群或者祖先數,為確定最理想的K值,可以設定K=1~10,用軟件Admixture進行計算,命令如下:
for i in {1..10};do admixture --cv filter.bed $i|tee out${i};done
從結果文件中提取出Cross-validation error值即不同K值的錯誤率,一般認為CV error最小值對應的K值為最佳K值。提取CV error命令如下:
grep -h CV out|awk -F ':' '{print NR"\t"$2}'|sed '1i\\K\tCV_error' > CV_for_plot.txt
然后將結果在R中可視化,最終得到CV error的分布圖圖4。具體代碼如下:
library(ggplot2)
mydata<-read.table("CV_for_plot.txt",header=T,sep="\t")
qplot(x=K,y=CV_error,color=I('black'),data=mydata)+geom_line(color="red",lwd=1)
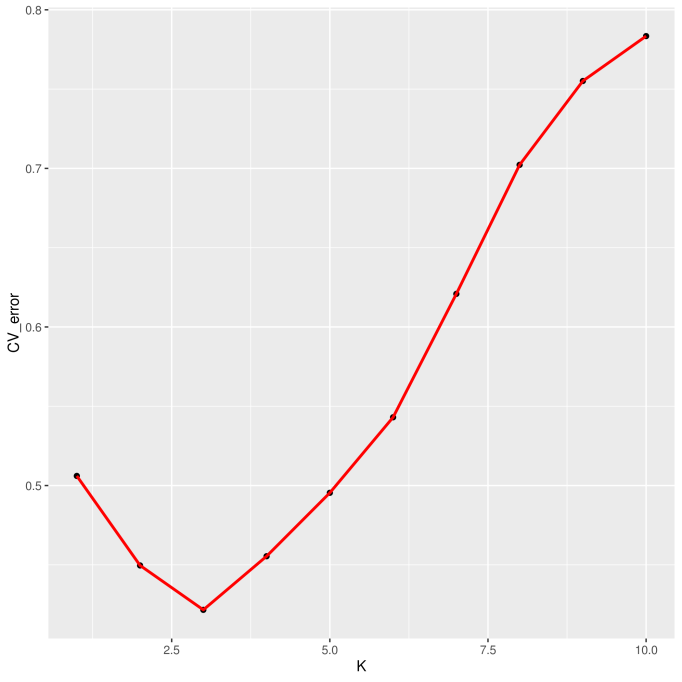
圖4 CV error的分布圖
根據結果顯示K=3對應的CV error最小,故為最佳K值。
遺傳結構圖形繪制
根據K=3時軟件Admixture計算的結果文件filter.3.Q利用R軟件繪制遺傳結構分布圖。
具體畫圖代碼如下:
mydata<-read.table("filter.3.Q")
barplot(t(as.matrix(mydata)),col=c("#E69F00","#56B4E9","#CC79A7"),border=NA)
然后就得到我們的目標結果啦!
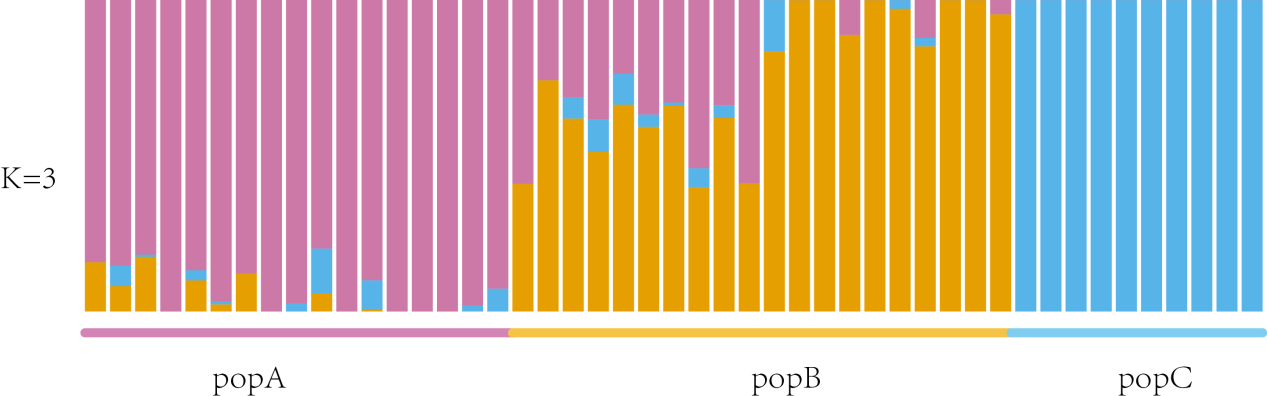
圖5 K=3時遺傳結構分布圖
以上就是基于群體SNP進行遺傳結構分析和圖形繪制的方法,是不是沒有想象的復雜呢,趕快用自己的數據試試吧!沒有服務器資源的老師也可以用Structure軟件的windows的Java圖形界面版本試試,該軟件包含三個主要參數length of burn-in period、Number of MCMC Reps after burn-in、admixture model,前兩個參數一般設置為10000,第三個參數是該軟件涉及的兩種模型 no admixture model 和admixture model,前者假設亞群間不存在雜交,后者假設亞群間存在雜交,一般情況下選擇admixture model更合理哦~
不管基于哪種軟件如果大家實戰分析的過程中有任何疑問,歡迎和我們一起探討,可在文末留言或者郵件交流([email protected])。
下面是動植物產品線的產品類型,如果對其他產品比較感興趣的,也可以郵箱反饋我們,后續我們一一為大家安排。