
2018-10-31
進化樹,也稱系統發生樹(英文:phylogenetic tree或evolutionary tree),主要是通過多序列比對結果以樹形圖形式的一個呈現,表明被認為具有共同祖先的各物種相互間演化關系的樹。
繪制進化樹,可以基于核酸序列,可以基于蛋白序列,上升到組學水平,最常見的就是基于基因組信息。即,通過基因組序列比對,獲得豐富的SNP信息......
通過NCBI搜索“population evolution”,會發現進行群體進化研究時,不僅會用到核基因組序列,還會用到細胞器基因組(如線粒體)進行分析,而且文章數量都不少。
既然核基因組和細胞器基因組都可以用于群體進化分析,那么分析結果有沒有差別,究竟哪個分析更好呢?


首先,我們來看第一個問題:
基于細胞器基因組和基于核基因組的群體進化分析有沒有差別?
要回答這個問題,還要從進化樹的繪制說起。進化樹主要通過序列比對,根據樣品間數據的差異程度判斷其遺傳距離遠近。而線粒體/葉綠體是真核細胞的一種細胞器,有其自己的基因組,能自主進行復制、轉錄和翻譯過程;線粒體/葉綠體基因組不發生重組,具有母系遺傳的特點。
換句話說,就是細胞器基因組相對獨立于核基因組,有自己的遺傳機制。那么對同樣的物種,做出不一樣的樹形結構就好理解了。用于構樹的數據(一個是源于線粒體基因組,一個是源于核基因組)所反映的遺傳關系不同,導致了樹形結構出現不一致的情況。
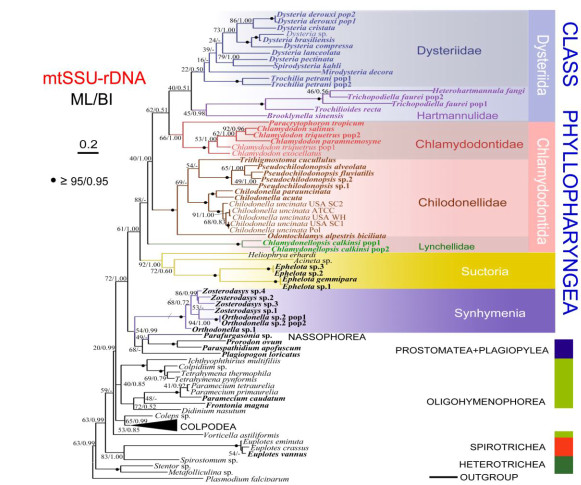
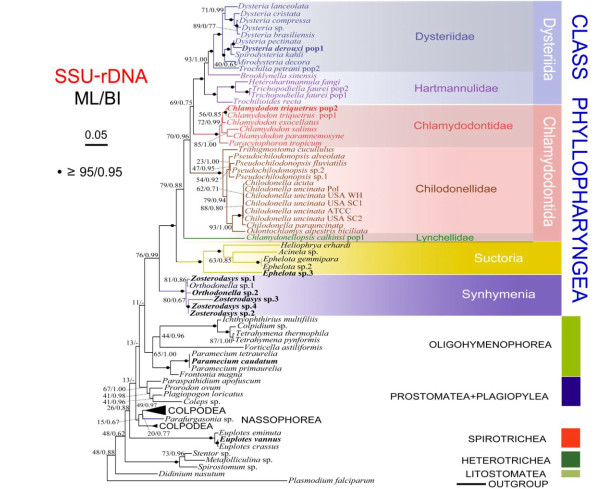
以線粒體基因組和核基因組為例:wang等(2017)利用線粒體和核基因組信息進行纖毛蟲屬纖毛門(Ciliophora)的聚類分析,可以發現物種的聚類相差不大,但樹形分枝有所不同。
(Wang et al., 2017)
我們再看第二個問題:
究竟哪個分析更靠譜呢?
Katz等(2011)研究指出,線粒體基因組序列變異率大于核基因組序列變異率,所以對于種內種間的分化檢測力較強;而核基因相比較而言比較保守,不容易區分。
Allio等(2017)研究發現,無脊椎動物中,線粒體基因組DNA的突變率是核基因組DNA突變率的2-6倍,而在脊椎動物中,線粒體基因組DNA的突變率高達20倍。相比較而言,無脊椎動物中線粒體基因組用于進化樹的分析可信度不如脊椎動物。
其實物種不同,線粒體DNA的突變率也不相同。Allio等(2017)在研究中提到:線蟲、金小峰、海洋水蚤等線粒體的突變率較高;果蠅、海膽、蛺蝶的線粒體突變率與核基因組突變率相差不大;, 珊瑚蟲等后生動物的線粒體突變率低于核基因組突變率。毫無疑問,突變率高的基因組所帶來的樣品聚類信息更為準確。
在真核生物中,哺乳動物的線粒體DNA的突變率比核DNA的突變率高,而在植物中,線粒體DNA(葉綠體DNA)的突變率比核DNA的突變率低。所以,我們常常可以看到在動物群體進化的研究中會用到線粒體/葉綠體基因組信息,而在植物的群體進化研究中常用到核基因組信息。
以水稻為例:
王榮升等(2011)用葉綠體基因組信息對水稻群體進化的研究,秈、粳稻的分群并不是很理想。
(王榮升等,2011)
而huang等(2012)用核基因組測序數據對水稻進行群體進化研究,不同亞種間能夠很好的分群。
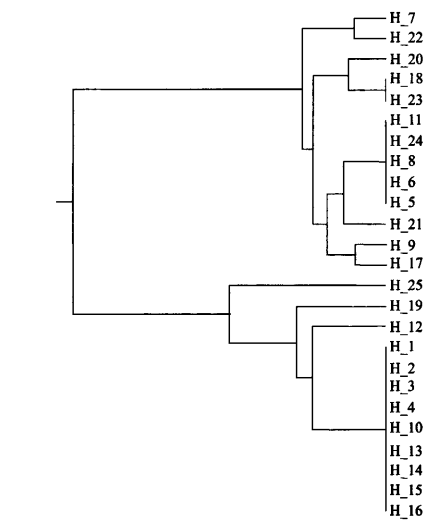
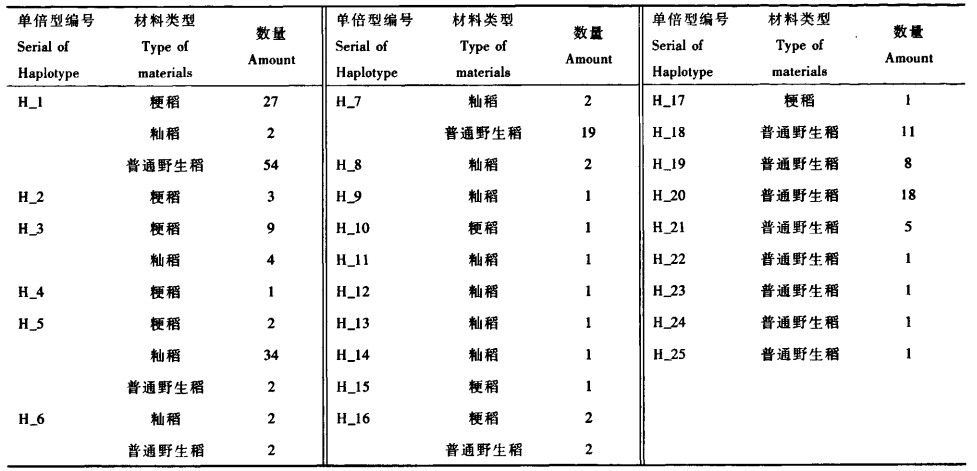
(huang et al., 2012)
總結一下,基因組突變率的差異導致了最終樹形結構的不同,而突變率高的基因組信息能夠帶來更準確的進化樹分析。所以在群體進化研究中,如果研究種內材料間差異,盡量選擇突變率大的基因組信息加以分析;如果研究種間差異,基因組的選擇將結果影響不大。
參考文獻
王榮升, 魏鑫, 曹立榮,等. 基于葉綠體基因多樣性的中國水稻起源進化研究[J]. 植物遺傳資源學報, 2011(5):686-693.
Allio R, Donega S, Galtier N, et al. Large Variation in the Ratio of Mitochondrial to Nuclear Mutation Rate across Animals: Implications for Genetic Diversity and the Use of Mitochondrial DNA as a Molecular Marker[J]. Molecular Biology and Evolution, 2017, 34(11).
Gao, F., Warren, A., Zhang, Q., Gong, J., Miao, M., Sun, P., Xu, D., Huang, J., Yi, Z., Song, W., 2016b. The all-data-based evolutionary hypothesis of ciliated protists with a revised classification of the phylum Ciliophora (Eukaryota, Alveolata). Sci. Rep. 6, 24874.
Huang X, Kurata N, Wei X, et al. A map of rice genome variation reveals the origin of cultivated rice[J]. Nature, 2012, 490(7421):497-501.
Wang P, Wang Y, Wang C, et al. Further consideration on the phylogeny of the Ciliophora: Analyses using both mitochondrial and nuclear data with focus on the extremely confused class Phyllopharyngea.[J]. Molecular Phylogenetics & Evolution, 2017, 112.
Katz, L.A., DeBerardinis, J., Hall, M.S., Kovner, A.M., Dunthorn, M., Muse, S.V., 2011. Heterogeneous rates of molecular evolution among cryptic species of the ciliate morphospecies Chilodonella uncinata. J. Mol. Evol. 73, 266-272.