
2018-09-25
根據客戶提供差異基因(轉錄本),差異蛋白,差異代謝物可進行如下關聯分析:
方案一 轉錄組與蛋白質組關聯分析
方案二 轉錄組與代謝組關聯分析
方案三 蛋白質組與代謝組關聯分析
方案四 轉錄組、蛋白質組與代謝組三者關聯網絡分析
方案五 代謝組-微生物多樣性聯合分析
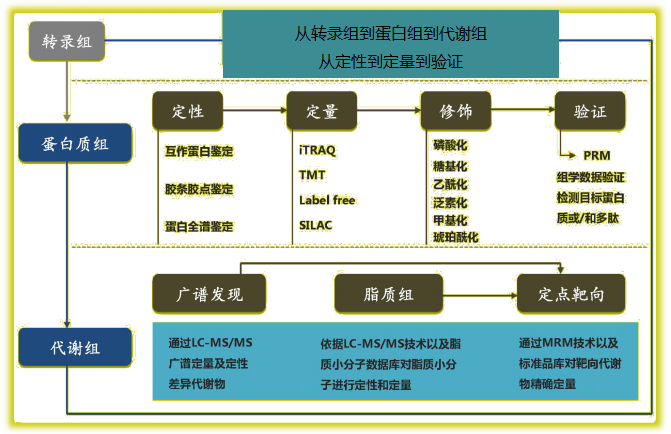
方案一 轉錄組與蛋白質組聯合分析示例
1.1 mRNA與蛋白表達相關性分析
將所有的差異表達蛋白的 mRNA 表達量計算出來,比較兩者之間的相關性。我們將基因和蛋白能夠完全對應,表達趨勢完全一致的列出:
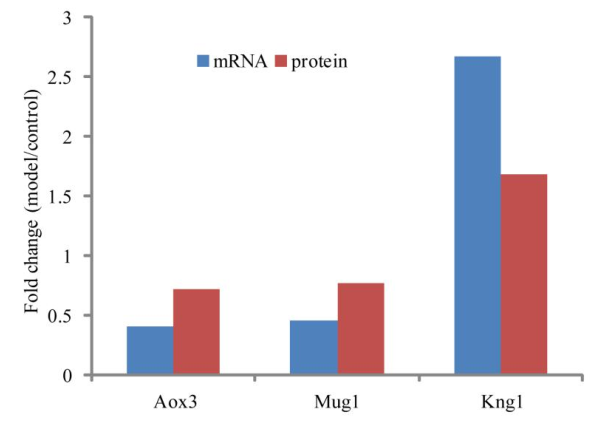
Fig.1 差異蛋白與差異基因表達相關性分析
1.2 差異基因與差異蛋白 pathway 分析
將差異基因與差異蛋白同時使用 GenMAPP v2.1 向 KEGG pathway 數據庫映射, 獲得他們的共同的 pathway 信息。
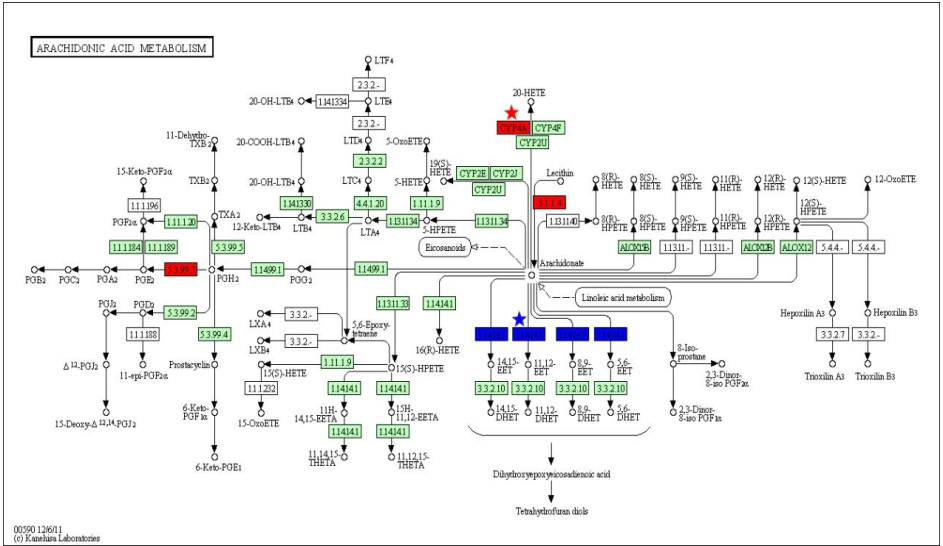
Fig.2 差異基因與差異蛋白 pathway 圖,帶☆的是蛋白,紅色的是上調,藍色的是下調
1.3 差異基因與差異蛋白互作網絡構建
整合構建 mRNA 與 protein 互作網絡如下:
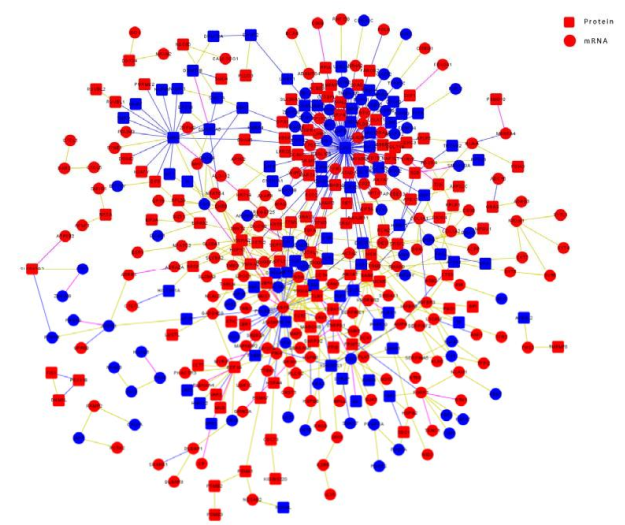
Fig.3 差異基因與差異蛋白整合調控網絡構建,圓點是基因,矩形點是蛋白
方案二 轉錄組與代謝組聯合分析示例
2.1 mRNA 與代謝物表達相關性分析
將所有的差異表達代謝物的酶基因對應 mRNA 表達量計算出來,比較兩者之間的相關性。我們將基因和代謝物能夠完全對應,表達趨勢完全一致的列出:
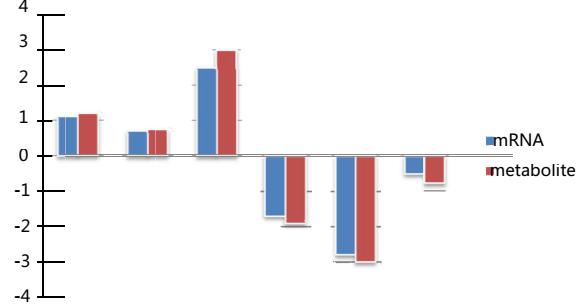
Fig.4 差異代謝物與差異基因表達相關性分析
2.2 差異基因與差異代謝物 pathway 分析
將差異基因與差異代謝物同時使用 GenMAPP v2.1 向 KEGG pathway 數據庫映射,獲得他們的共同的 pathway信息。
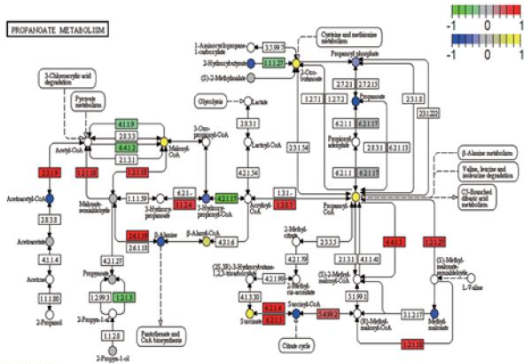
Fig.5 差異基因與差異代謝物 pathway 圖,圓點是代謝物,長方形是酶基因
2.3 差異基因與差異代謝物互作網絡構建整合構建 mRNA 與代謝物互作網絡如下:
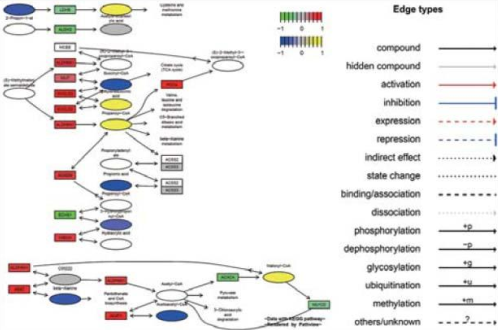
Fig.6 差異基因與差異代謝物整合調控網絡構建,長方形是基因,橢圓是代謝物
方案三 蛋白組與代謝組聯合分析示例
參考轉錄組與代謝組關聯分析
方案四 轉錄組、蛋白組與代謝組聯合分析示例
綜合上述得到的基因、蛋白與代謝之間的調控關系,構建三者的調控網絡。
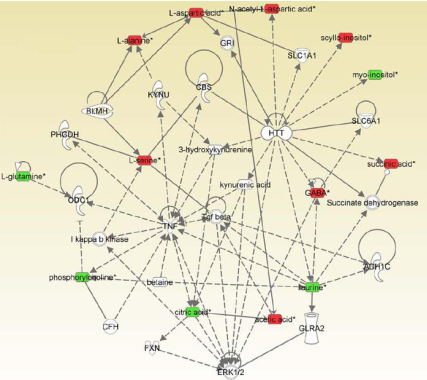
Fig7. IPA 軟件構建的基因、蛋白、代謝物互作網絡
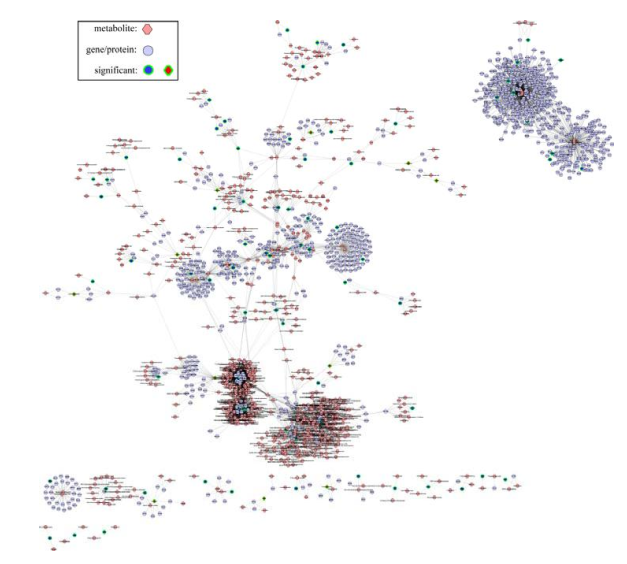
Fig8. metscape 軟件構建的基因、蛋白、代謝物互作網絡
方案五 代謝組-微生物多樣性聯合分析示例
1. Mantel test
用于檢驗來源于相同樣本集的兩個距離矩陣間的相關性強弱。
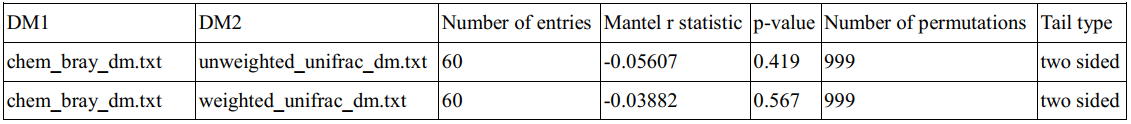
第一和第二列為用于相關性檢驗的兩套數據所對應的距離矩陣;
第三列為樣本量;
第四列為Mantel檢驗得到的相關系數r,r的絕對值越接近1,表明兩套數據間的相關性越高; 第五列為P值,P值越接近于零,表明相關性檢驗的統計顯著性越高,結果越可靠;
第六列為置換檢驗的次數;第七列表明檢驗類型為單側或雙側檢驗。
2. Procrustes分析
可以通過計算各樣本在兩套數據上的間距平方和(M2),來量化兩套數據的一致性高低。還可以對樣本進行置換檢驗,從而評估兩套數據之間相似度的統計顯著性。
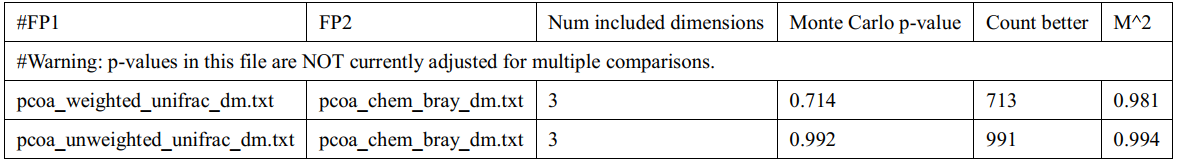
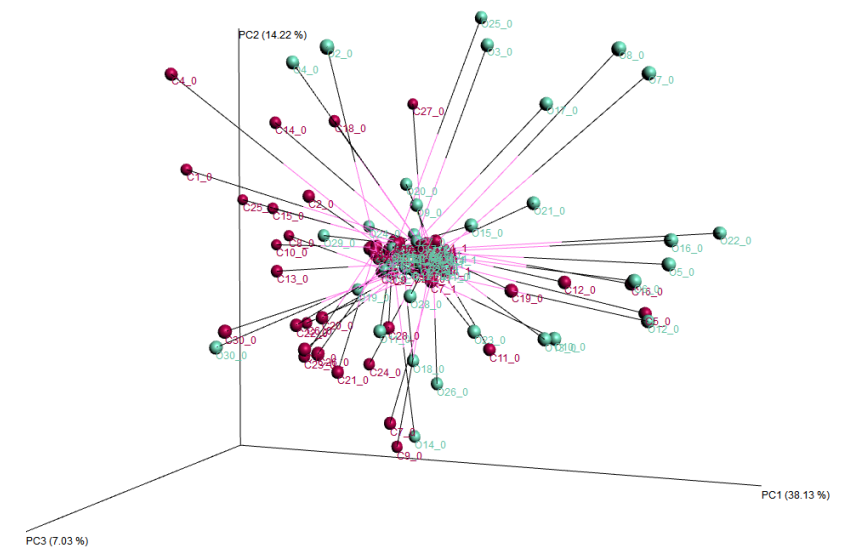
Fig9.Procrustes 分析結果的三維排序圖
3. CIA 協慣量分析
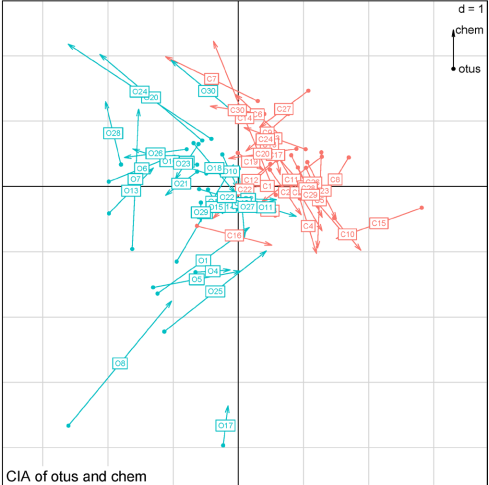
Fig10.CIA 分析結果的二維排序圖
參考文獻
[1] Sunia A. Trauger,Ewa Kalisak ,J,Gary Siuzdak et. al.Correlating theTranscriptome, Proteome, and Metabolome in the Environmental Adaptation of aHyperthermophile. Journal of Proteome Research 2008, 7, 1027–1035 1027.
[2] Cho K et al. Integrated transcriptomics, proteomics, and metabolomicsanalyses to survey ozone responses in the leaves of rice seedling. J ProteomeRes. 2008Jul;7(7):2980-98.
[3] Coen M et al. Integrated application of transcriptomics and metabonomics yields new insight into the toxicity due to paracetamol in the mouse. J Pharm Biomed Anal. 2004 Apr 1;35(1):93-105.
[4] Espinoza C et al. Interaction with diurnal and circadian regulation resultsin dynamic metabolic and transcriptional changes during cold acclimation in Arabidopsis. PLoS One. 2010 Nov 23;5(11):e14101.
[5] Bono H et al. Comprehensive analysis of the mouse metabolome based on the transcriptome. Genome Res. 2003 Jun;13(6B):1345-9.
[6] Kanani H et al. Individual vs. combinatorial effect of elevated CO2conditions and salinity stress on Arabidopsis thaliana liquid cultures:comparing the early
[7] Mantel, N., The Detection of Disease Clustering and a Generalized Regression Approach. Cancer Research, 1967. 27(2 Part 1): p. 209-220.
[8]Gower, J.C., Generalized procrustes analysis. Psychometrika, 1975. 40(1): p. 33-51.
[9]Dray, S., D. Chessel, and J. Thioulouse, Co-inertia analysis and the linking of ecological data tables.Ecology, 2003. 84(11): p. 3078-3089.