2017-10-25
絞股藍是多年生匍匐草本植物,在亞洲廣泛種植,有很多重要的藥用屬性。絞股藍皂苷,是絞股藍中的主要有效成分,已被充分研究。考慮到絞股藍重要的藥用功能,做絞股藍的轉錄組信息研究是很有必要的,有利于開展以后的轉錄調控研究。廣西醫科大學吳耀生教授團隊和上海派森諾合作,通過絞股藍測序,總共獲得了50,654,708個unigenes。通過計算unigene的表達量,并將數據和五個常用數據庫進行比對,獲得unigenes不同方面的注釋信息。發現DESeq分析結果中FPS,SS和SE酶表現出不同的表達水平。相對于根和莖,葉片中FPS,SS和SE的表達量是最高的。本研究通過對自然環境生長的絞股藍進行轉錄學分析,發現unigene的表達,酶的表達(FPS,SS和SE)和絞股藍皂苷的分布是一致的。
絞股藍的主要有效組成是絞股藍皂苷,它有多種多樣的生物活性,這可以解釋絞股藍為什么會在天然藥學中有廣泛應用。例如,絞股藍皂苷可以通過增加胰島素降低血糖。還有其他功能應用像抗癌,抗炎癥,抗焦慮,降血脂,保護肝細胞,神經保護和免疫保護等等。三帖合成途徑可以分解為三個部分:1、IPP(異戊烯基焦磷酸)或者DMAPP(二甲基烯丙基焦磷酸酯)合成;2、鯊烯合成和環化;3、功能化反應獲得鯊烯復合物(圖1)。和人參皂苷一樣,絞股藍皂苷在藥學和健康學中有很多重要應用。但是絞股藍皂苷和人參皂苷相比,有更高的異質性,其中有超過169種絞股藍皂苷。絞股藍比人參中的三萜化合物種類高出5倍。這可能和三帖合成途徑中的基因與酶的差異表達相關。
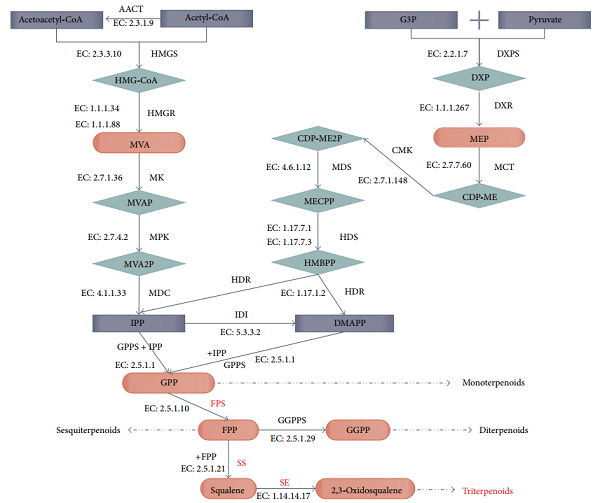
圖1 三帖合成途徑
高通量測序獲得352,999,296條原始reads,過濾掉接頭和低質量序列,剩余249,488,643條clean reads。組裝之后得到1,119,964個contigs,總長度達249,488,543bp。將contigs拼接成轉錄本,得到159,858,904個轉錄本序列。和NR數據庫比對后,獲得50,654,708個unigene,平均長度755bp。
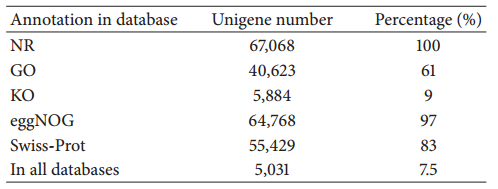
用NR,GO,KEGG,eggNOG和Swiss-Port數據庫注釋unigenes。整體看來,eggNOG中鑒定率最高,達到96.57%,KEGG中鑒定率最低,只有8.77%。
表1 unigenes 注釋結果
統計unigene的RPKM,做RPKM的密度分布圖。發現中等范圍表達量的unigenes的AUC(曲線下面積)占絕大部分,低表達或者高表達的unigenes的AUC是少部分的(圖2)。通過密度分布圖發現,纖維根中的unigenes要比莖和葉片中的unigenes表達量高。盡管莖和葉片有相似的unigene表達量,莖中的表達量稍微高于葉片中的表達量。另外分析編碼FPS,SS,SE和β-AS(β-香樹精合成酶)的unigenes的表達,發現這些unigenes在葉片中高表達,在纖維根中低表達,在莖中要比纖維根中表達量高。編碼β-AS的unigene在葉片中的表達量是纖維根中的將近125倍。通過DESeq軟件做兩兩比較分析測序數據,通過韋恩圖展現出差異表達的unigene的分布情況(圖3)。
表2 樣品中unigenes的RPKM值
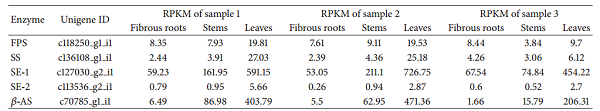
表3unigenes兩兩比較上下調基因統計結果
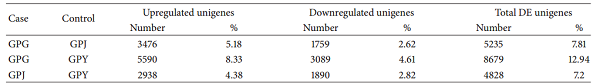
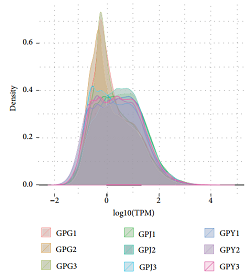
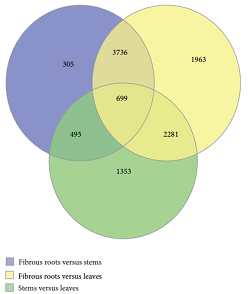
圖2 unigenes的RPKM密度分布圖 圖3差異unigenes的韋恩圖
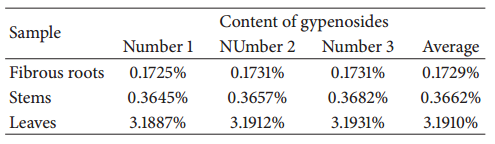
分光光度法測定絞股藍的皂苷含量分布。所有的樣品中葉片的皂苷含量最高(3.189%),在莖(0.365%)或者纖維根(0.172%)中皂苷含量比較低。
表4 絞股藍中皂苷含量分布
基于KEGG通路分析,將涉及三鐵合成通路有關的酶分為三類:1、IPP或者DMAPP合成相關的酶;2、鯊烯合成和環化相關的酶;3、鯊烯功能化反應中的酶。文中主要關注的是FPS,SS和SE三種酶,它們在三帖合成和環化中起重要作用。首先FPS比較分析結果中,只有纖維根和葉片中的表達比較有差異。再次,SS比較分析結果中,兩組分析結果中有差異,表明葉片中的表達要高于纖維根和莖。最后,在SE比較分析結果中,葉片中的表達高于莖和纖維根,而 且纖維根中的表達高于莖(圖4)。
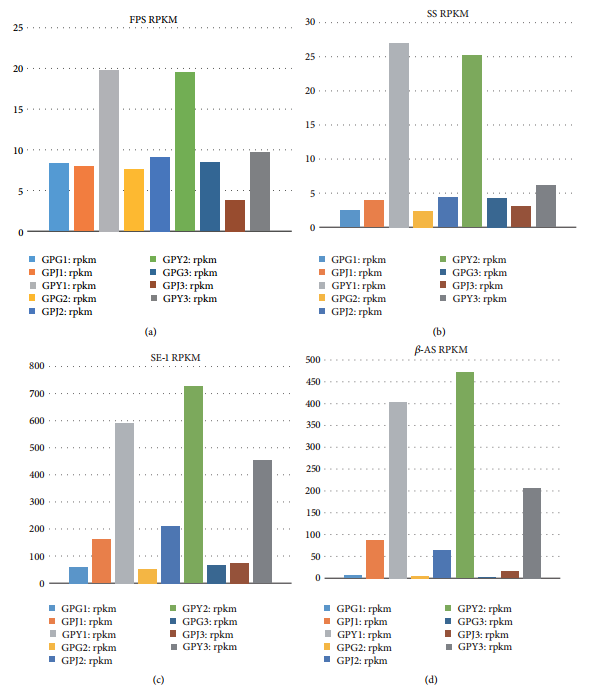
圖4 FPS,SS,SE中基因的RPKM
在unigene功能注釋,RPKM表達量統計和絞股藍含量測定之后,分析unigene表達,酶(unigene的產出物)的表達和絞股藍皂苷的含量分布之間的關聯,發現unigene和酶的表達與皂苷含量分布呈正相關。也就是說樣本(纖維根,莖,葉片)中unigene和酶(FPS,SS和SE)的表達取決于皂苷含量的分布。這一結論,有利于絞股藍中其他次級代謝產物的深入研究。
文獻來源
Chen Q, Ma C, Qian J, et al. Transcriptome Sequencing of Gynostemma pentaphyllum to Identify Genes and Enzymes Involved in Triterpenoid Biosynthesis[J]. International journal of genomics, 2016.


