資源分享 | 蛋白質(zhì)研究常用數(shù)據(jù)庫(kù)��!
2024-08-09
當(dāng)我們投身于蛋白質(zhì)的研究時(shí),探索的焦點(diǎn)通常匯聚于蛋白質(zhì)表達(dá)的動(dòng)態(tài)變化��、蛋白質(zhì)發(fā)生翻譯后修飾的精妙調(diào)控��、蛋白質(zhì)間錯(cuò)綜復(fù)雜的相互作用、蛋白質(zhì)所參與的生命過(guò)程和功能�、三維結(jié)構(gòu)的奧秘以及作為藥物靶點(diǎn)的潛力��。每一個(gè)研究方向都蘊(yùn)含著豐富的知識(shí)并彼此交織,共同編織出蛋白質(zhì)世界的復(fù)雜圖景��。為了應(yīng)對(duì)這一領(lǐng)域的復(fù)雜性���,我們精心整理了一系列常用的蛋白質(zhì)研究數(shù)據(jù)庫(kù)�����,旨在為科研工作者提供高效便捷的信息資源,節(jié)省寶貴的時(shí)間與精力。

一���、蛋白綜合數(shù)據(jù)庫(kù)
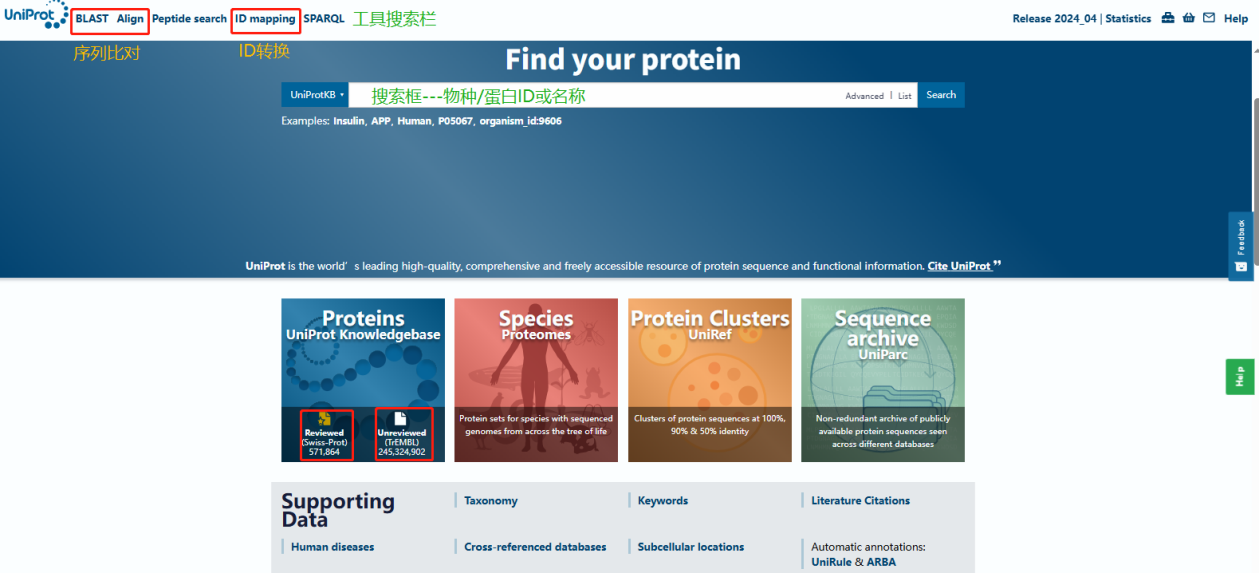
UniProt(Universal Protein Resource,https://www.uniprot.org/)是一個(gè)免費(fèi)開(kāi)放的綜合性蛋白質(zhì)數(shù)據(jù)庫(kù)。該數(shù)據(jù)庫(kù)蛋白信息來(lái)源于EMBL�、GenBank�、DDBJ等公共數(shù)據(jù)庫(kù)(非冗余蛋白質(zhì)序列數(shù)據(jù)庫(kù))��,目前使用頻率非常高����,包含信息非常全面����。該數(shù)據(jù)庫(kù)主要提供了蛋白質(zhì)序列和豐富的功能注釋信息,用途多樣���,是查詢蛋白功能和蛋白質(zhì)組學(xué)研究中搜庫(kù)匹配的首選數(shù)據(jù)庫(kù)。應(yīng)用工具搜索不僅可以進(jìn)行序列-物種/序列-序列比對(duì),還可以對(duì)不同數(shù)據(jù)來(lái)源的ID進(jìn)行轉(zhuǎn)換��。
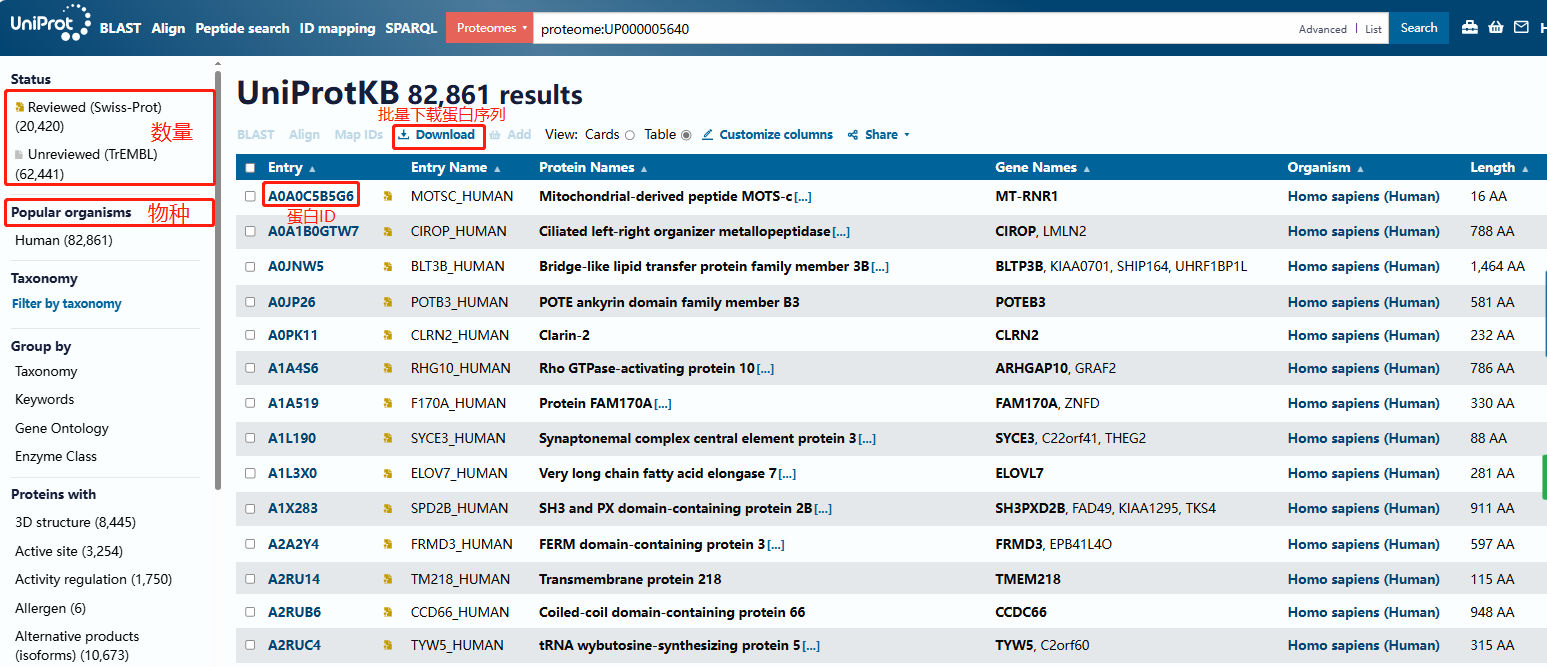
常用的UniProtKB由兩個(gè)子庫(kù)構(gòu)成:Swiss-Prot和TrEMBL。其中Swiss-Prot通常來(lái)源于已發(fā)表的文獻(xiàn)��,是經(jīng)過(guò)人工驗(yàn)證和注釋的高質(zhì)量和可靠的非冗余蛋白質(zhì)注釋數(shù)據(jù)�����,人工注釋這些數(shù)據(jù)效率較低�����?����;诨蚪M序列由機(jī)器自動(dòng)翻譯和預(yù)測(cè)的蛋白質(zhì)序列數(shù)據(jù)庫(kù)TrEMBL建立彌補(bǔ)了人工注釋的不足����,并提供了大量新蛋白質(zhì)信息�,但其注釋程度不如Swiss-Prot高。
當(dāng)我們做蛋白質(zhì)組學(xué)涉及以上兩個(gè)子庫(kù)的選擇問(wèn)題時(shí),如果對(duì)鑒定的準(zhǔn)確度要求更高���,可以選擇下載Swiss-Prot數(shù)據(jù)庫(kù)進(jìn)行搜庫(kù),常規(guī)物種做蛋白質(zhì)組學(xué)通常選擇Swiss-Prot�����。如果為了鑒定蛋白更加全面����,通常建議選擇UniprotKB的總蛋白序列信息進(jìn)行搜庫(kù)。
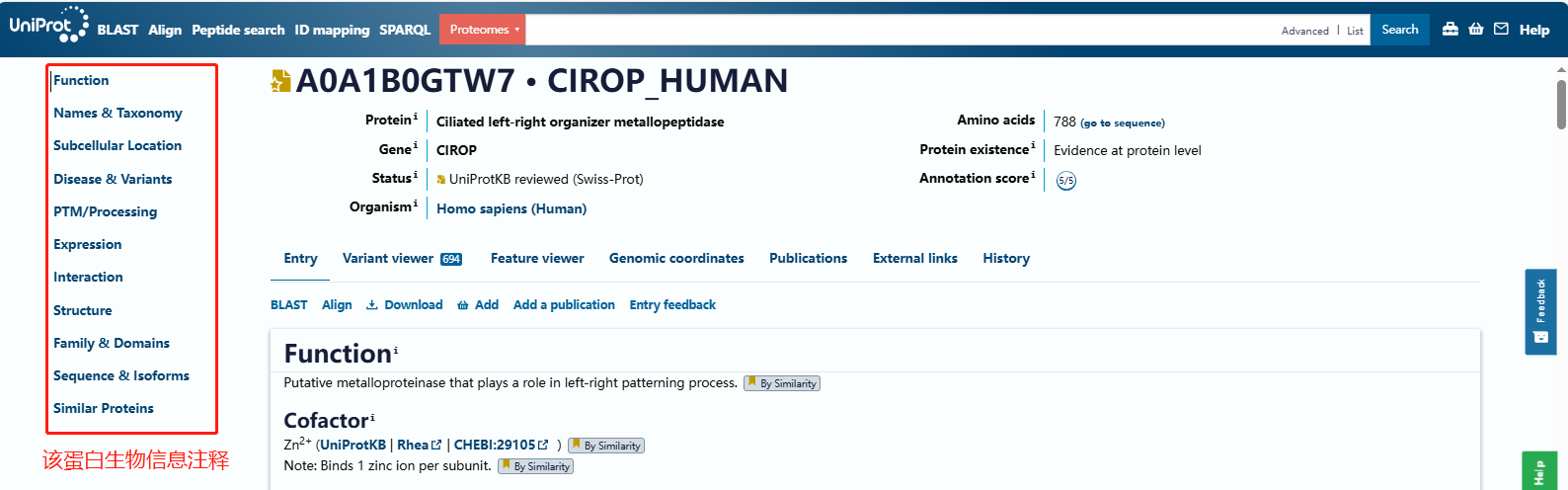
此外Uniprot數(shù)據(jù)庫(kù)包含豐富的功能模塊,主要包含:蛋白序列、結(jié)構(gòu)域�����、亞細(xì)胞定位���、翻譯后修飾�、表達(dá)情況、蛋白互作等,可以直接輸入蛋白質(zhì)ID或者名稱進(jìn)行查詢?cè)摰鞍讌⑴c的生物學(xué)過(guò)程����。
NCBI(National Center for Biotechnology Information�,美國(guó)國(guó)家生物技術(shù)信息中心���,https://www.ncbi.nlm.nih.gov/)數(shù)據(jù)庫(kù)包含了大量關(guān)于基因�、蛋白質(zhì)���、核酸序列��、疾病��、藥物、解剖學(xué)�����、文獻(xiàn)等多個(gè)方面的信息�,收錄全世界所有實(shí)驗(yàn)室檢測(cè)信息,是一個(gè)綜合性數(shù)據(jù)庫(kù)�����,NCBI的數(shù)據(jù)庫(kù)內(nèi)容更為廣泛和綜合����,可以提供36種不同的數(shù)據(jù)檢索及分析工具。
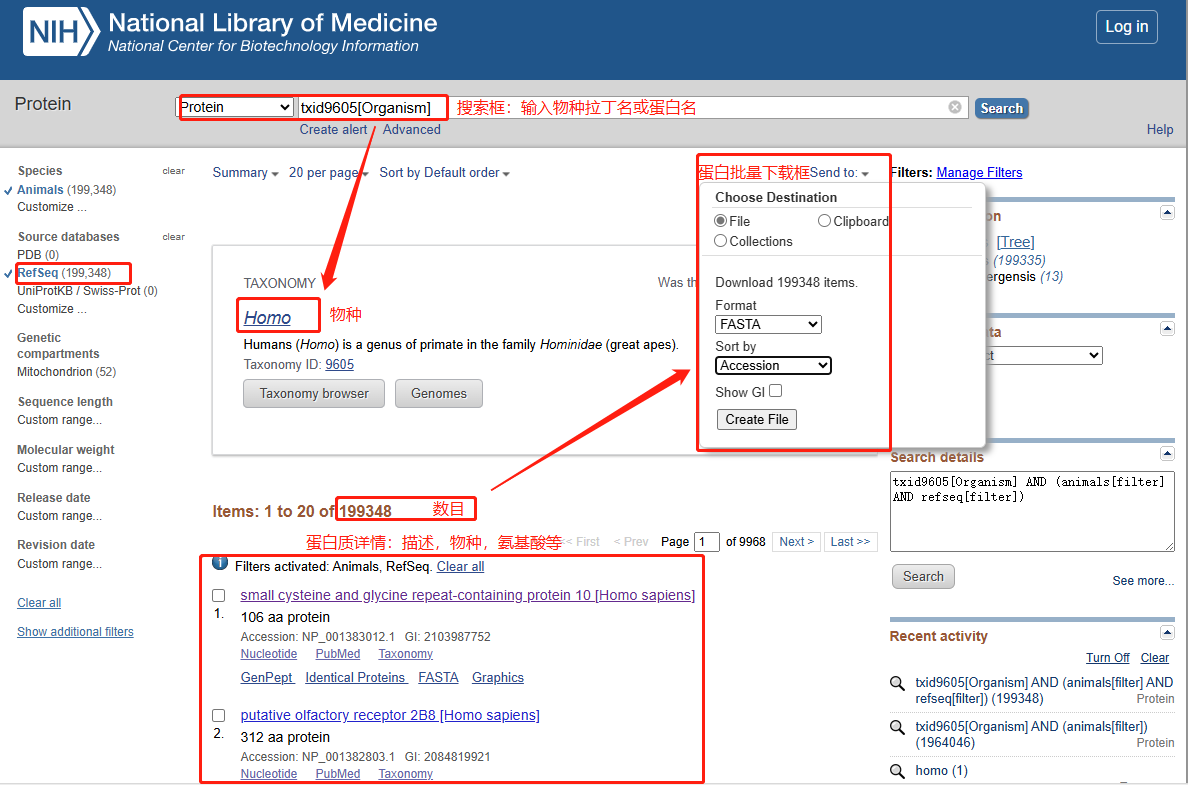
NCBI也可以作為蛋白質(zhì)組學(xué)的物種背景數(shù)據(jù)庫(kù)���,搜索物種信息即可得到RefSeq蛋白信息,但是會(huì)有很多冗余的蛋白信息�����。因此同一個(gè)物種����,NCBI的蛋白比UniProt多�,假陽(yáng)性也會(huì)隨之升高���。
關(guān)于蛋白質(zhì)組學(xué)搜庫(kù)數(shù)據(jù)庫(kù)的選擇(NCBI vs Uniprot)�����,通常建議優(yōu)先使用Uniprot數(shù)據(jù)庫(kù),若該物種Uniprot數(shù)據(jù)庫(kù)蛋白較少�����,可使用NCBI數(shù)據(jù)庫(kù)進(jìn)行搜庫(kù)�����。若特定物種在Uniprot和NCBI中均沒(méi)有蛋白數(shù)據(jù)�����,優(yōu)先考慮基因組或轉(zhuǎn)錄組測(cè)序序列翻譯成的蛋白質(zhì)數(shù)據(jù)庫(kù),也可以使用上一級(jí)或者近緣物種的蛋白質(zhì)數(shù)據(jù)庫(kù)作為備選數(shù)據(jù)庫(kù)�。
二�、蛋白翻譯后修飾數(shù)據(jù)庫(kù)
蛋白質(zhì)翻譯后修飾(Post-translational modification�����,PTM)是指在翻譯后的蛋白質(zhì)氨基酸殘基上通過(guò)添加或移除特定的基團(tuán)進(jìn)行化學(xué)修飾��,從而調(diào)節(jié)蛋白質(zhì)的活性、定位��、以及蛋白與其他生物大分子間相互作用���。
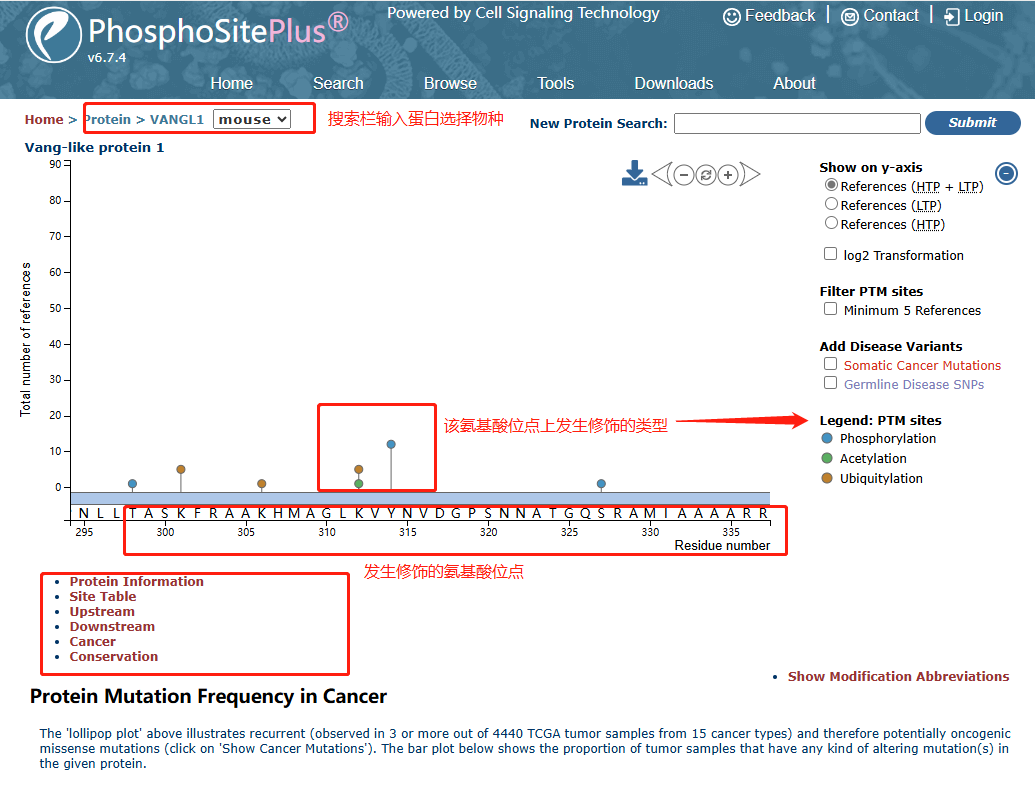
PhosphoSitePlus數(shù)據(jù)庫(kù)(https://www.phosphosite.org/)是一個(gè)由CST和NIH聯(lián)合開(kāi)發(fā)免費(fèi)的翻譯后修飾預(yù)測(cè)數(shù)據(jù)庫(kù)���,整合了大量來(lái)自高通量測(cè)序預(yù)測(cè)和科學(xué)研究實(shí)驗(yàn)驗(yàn)證的結(jié)果�,為蛋白質(zhì)翻譯后修飾的研究提供了全面的信息和工具����。該數(shù)據(jù)庫(kù)主要包括磷酸化、甲基化�����、乙?����;?�、泛素化等,共收錄了59499個(gè)蛋白的600798個(gè)翻譯后修飾位點(diǎn)。通過(guò)查詢蛋白質(zhì)可以獲得蛋白質(zhì)基本信息(結(jié)構(gòu)域���、亞細(xì)胞定位)以及蛋白質(zhì)發(fā)生修飾的類型、修飾位點(diǎn)、抗體����、修飾相關(guān)疾病����,以及激酶底物序列�。
圖2.1PhosphoSitePlus數(shù)據(jù)庫(kù)使用
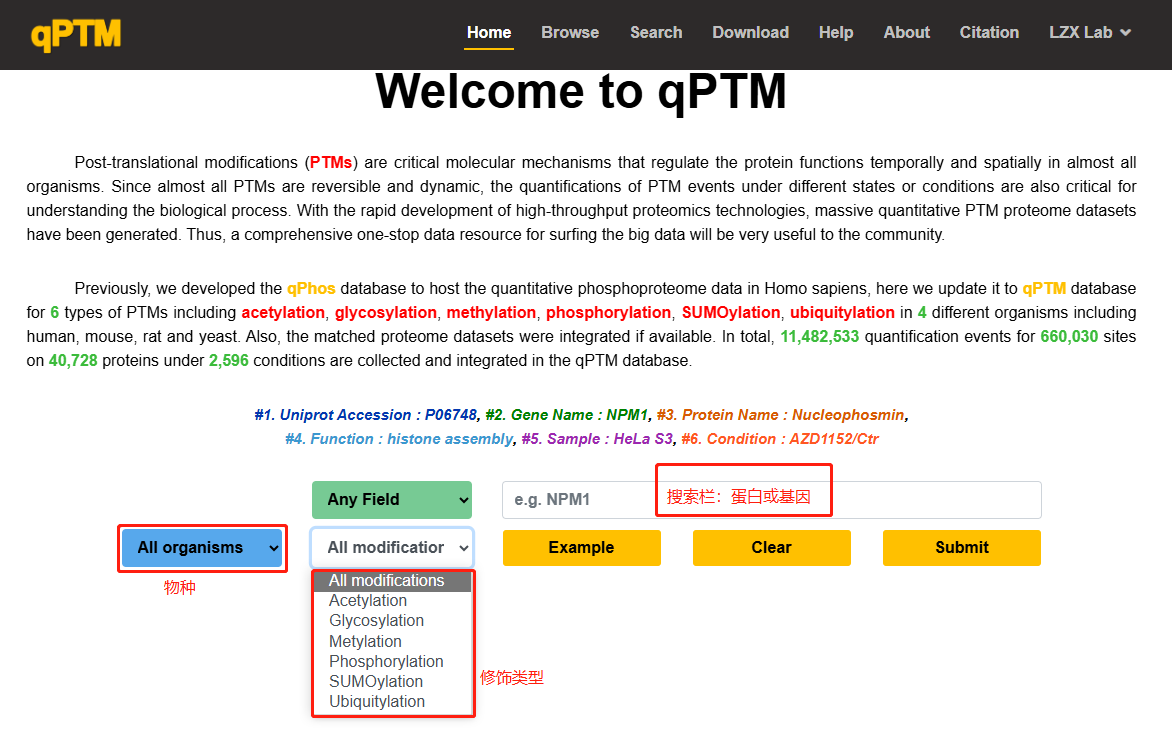
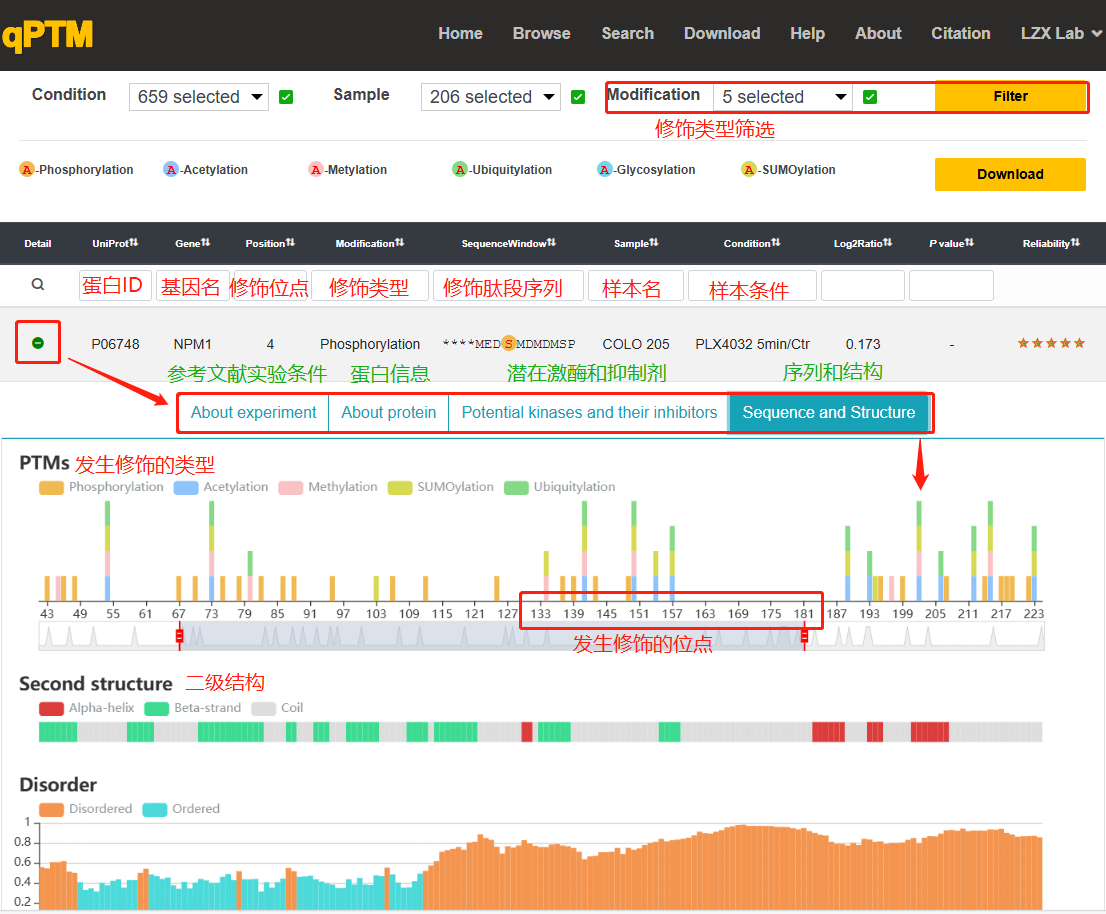
qPTM(quantification of Post-Translational Modifications,http://qptm.omicsbio.info)是中山大學(xué)腫瘤防治中心劉澤先教授團(tuán)隊(duì)收集并整合PTMs文獻(xiàn)的數(shù)據(jù)庫(kù),涉及從600多個(gè)已發(fā)表研究中收集的四種不同生物體(人��、大鼠���、小鼠�、酵母)中40728個(gè)蛋白質(zhì)在2596種條件下的660 030個(gè)非冗余PTM位點(diǎn),修飾類型包括6種(磷酸化、乙酰化�、糖基化��、甲基化、SUMO化以及泛素化修飾)。通過(guò)搜索特定物種的蛋白��,即可獲得前人研究的修飾發(fā)生的位點(diǎn)以及實(shí)驗(yàn)條件和參考文獻(xiàn)��。
圖2.2 qPTM數(shù)據(jù)庫(kù)使用
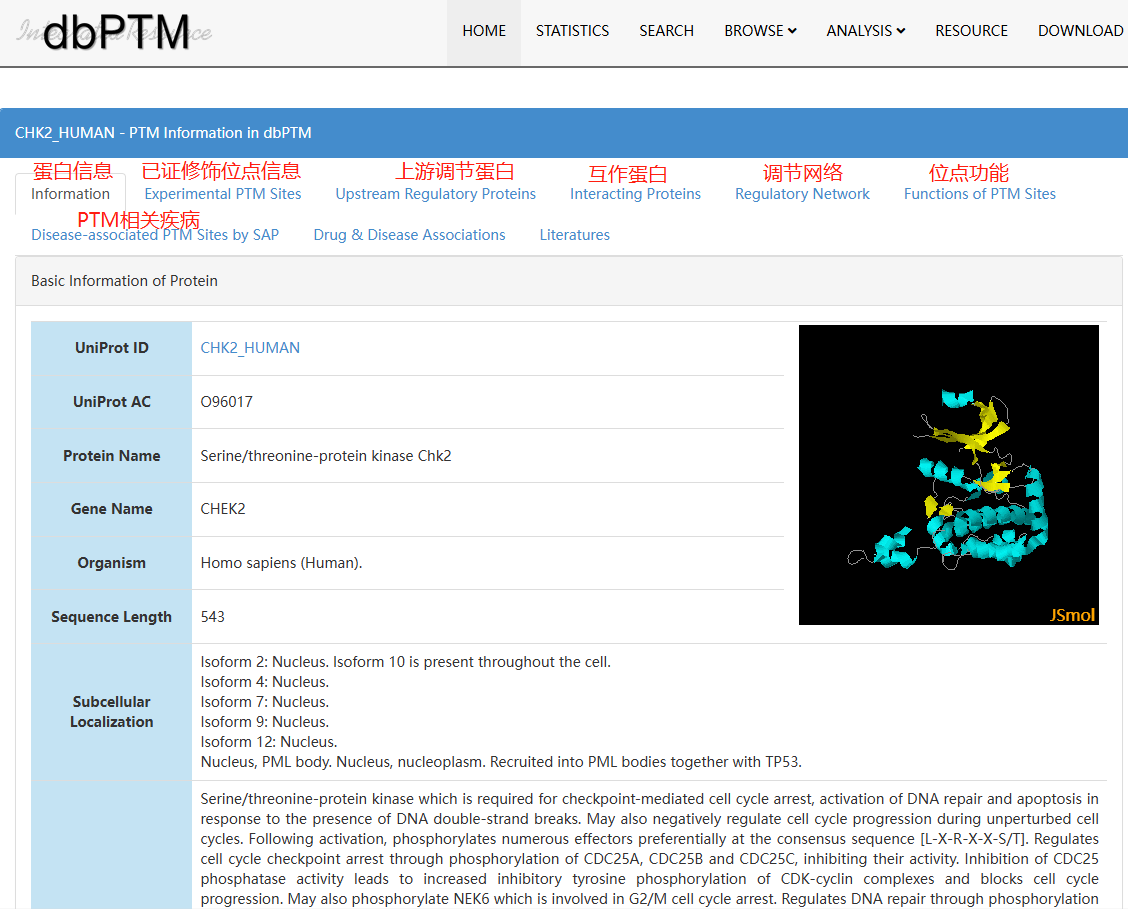
dbPTM(https://awi.cuhk.edu.cn/dbPTM/index.php)是蛋白質(zhì)翻譯后修飾 (PTM) 的綜合資源,整合來(lái)自40+數(shù)據(jù)庫(kù)����、70+種修飾類型����、已經(jīng)被實(shí)驗(yàn)/文獻(xiàn)證實(shí)的PTM位點(diǎn)和預(yù)測(cè)位點(diǎn)共2235664個(gè),其中重點(diǎn)修飾類型包括磷酸化、糖基化和硫修飾。通過(guò)搜索蛋白可獲得蛋白二級(jí)結(jié)構(gòu)、修飾位點(diǎn)信息、上游調(diào)節(jié)蛋白、位點(diǎn)功能以及疾病相關(guān)信息�����。
圖2.3dbPTM數(shù)據(jù)庫(kù)使用
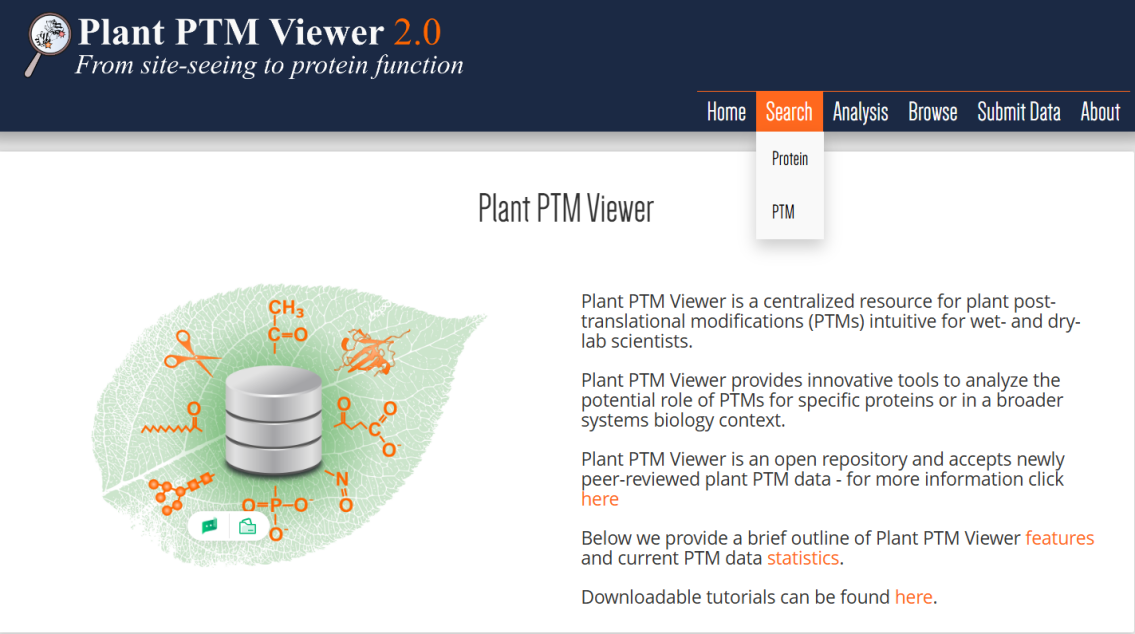
Plant PTM Viewer(http://www.psb.ugent.be/PlantPTMViewer)是植物蛋白翻譯后修飾數(shù)據(jù)庫(kù)����,包含8種不同植物(擬南芥、水稻、大豆�、小立碗蘚���、番茄�、玉米���、小麥�����、萊茵衣藻)大約128920個(gè)蛋白334255個(gè)PTM位點(diǎn)的33種蛋白質(zhì)修飾�����。通過(guò)該網(wǎng)站我們可以檢索目的蛋白在植物中的修飾情況,此外還可以搜索同源序列中的保守翻譯后修飾位點(diǎn)。
圖2.4Plant PTM Viewer數(shù)據(jù)庫(kù)使用
三���、蛋白質(zhì)互作數(shù)據(jù)庫(kù)
一般情況下蛋白質(zhì)很難單獨(dú)發(fā)揮作用�,都是由多個(gè)蛋白質(zhì)分子的相互協(xié)調(diào)共同實(shí)現(xiàn)復(fù)雜的細(xì)胞功能。對(duì)于已知蛋白與哪些未知蛋白具有結(jié)合作用���,我們可通過(guò)Co-IP結(jié)合質(zhì)譜鑒定技術(shù)(運(yùn)用蛋白質(zhì)相互作用數(shù)據(jù)庫(kù))對(duì)研究有更深入的了解。
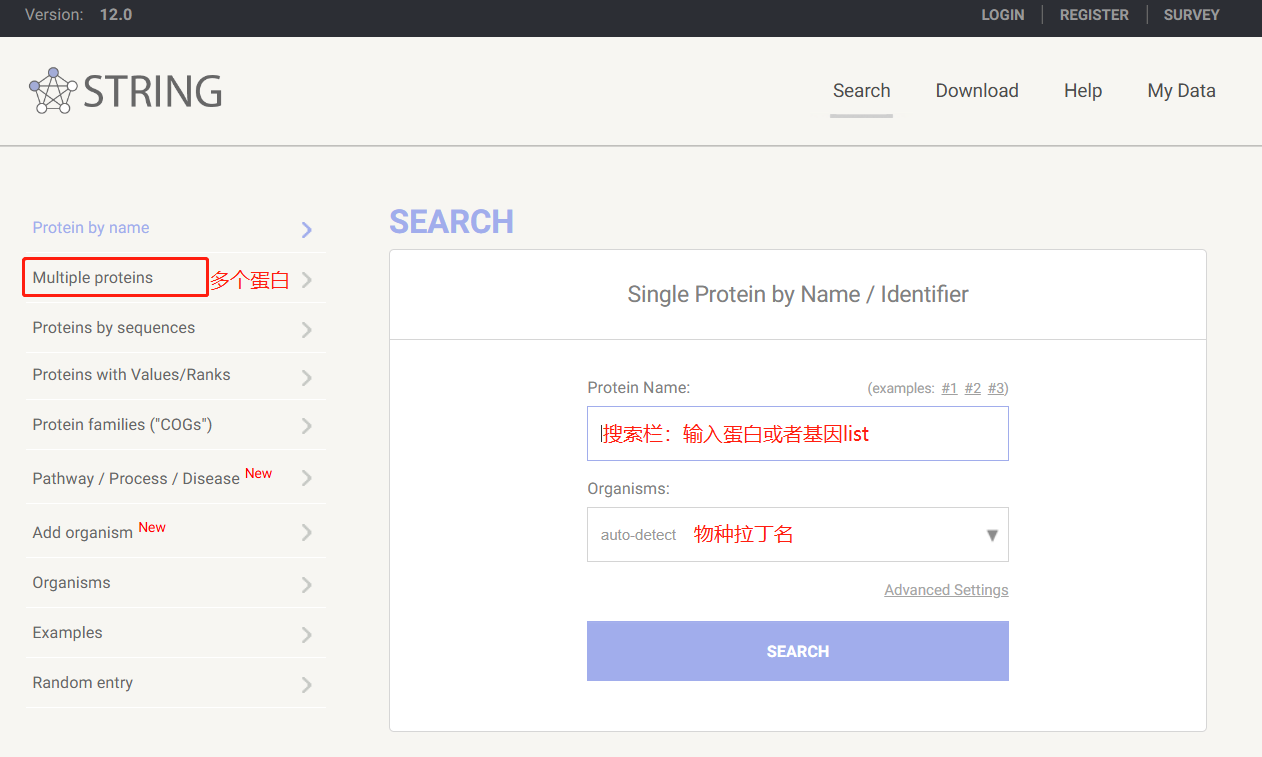
STRING(Search Tool for Retrieval of Interacting Genes/Proteins���,https://www.string-db.org/)數(shù)據(jù)庫(kù)整合了多個(gè)數(shù)據(jù)源的PPI信息,包括實(shí)驗(yàn)數(shù)據(jù)����、文獻(xiàn)挖掘和計(jì)算預(yù)測(cè)�����。它提供了廣泛的物種覆蓋和功能注釋,可用于PPI網(wǎng)絡(luò)構(gòu)建和功能分析�,涵蓋了5090種生物的兩千四百多萬(wàn)種蛋白質(zhì)���,是目前蛋白質(zhì)互作數(shù)據(jù)庫(kù)中覆蓋物種和互作信息尤其全面的一個(gè)數(shù)據(jù)庫(kù)�。主要可以進(jìn)行對(duì)已知蛋白與之互作的未知蛋白進(jìn)行分析����,結(jié)果互作網(wǎng)絡(luò)圖可根據(jù)Score值評(píng)估互作,Score分越高��,互作可能性越大�。
圖3.1STRING數(shù)據(jù)庫(kù)使用
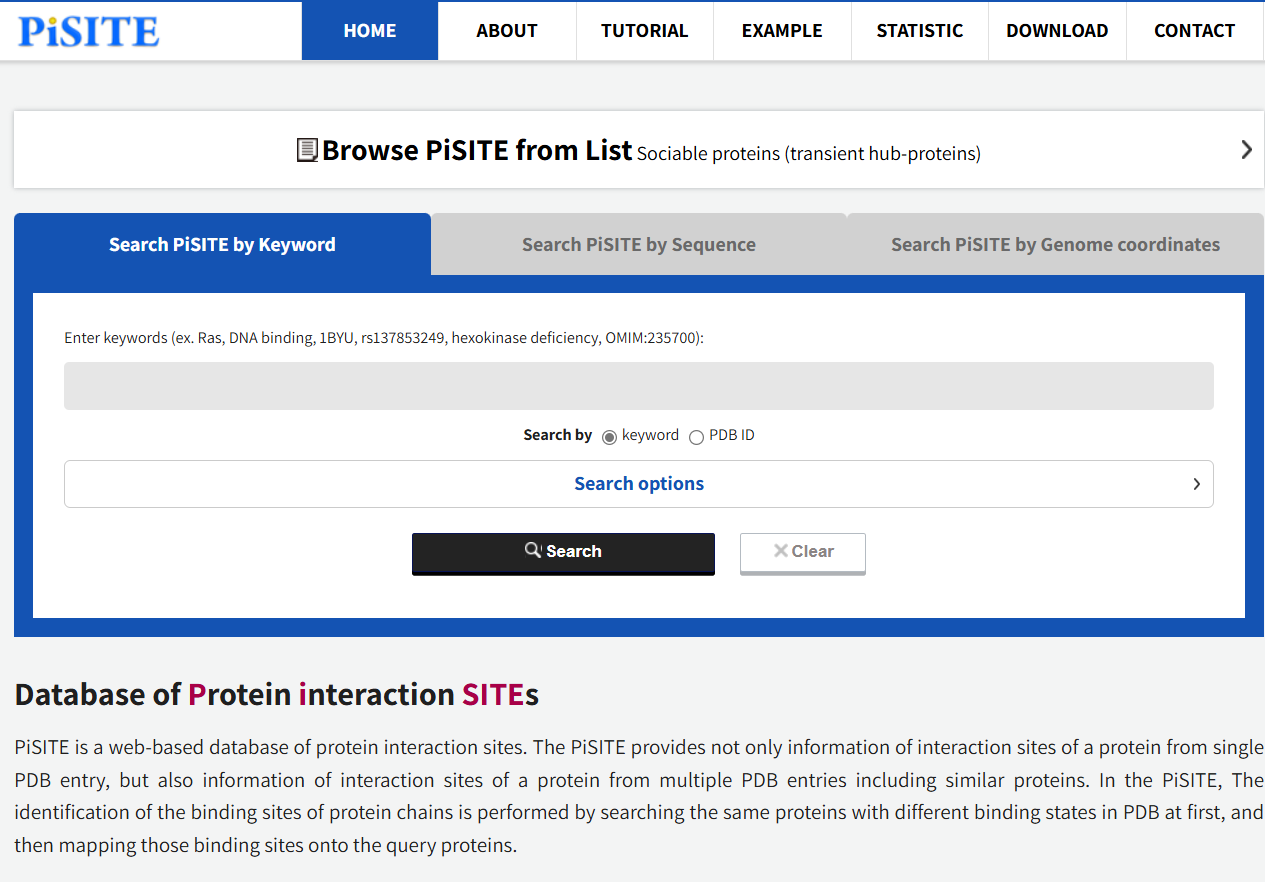
PiSite(Database of Protein Interaction Sites,http://pisite.hgc.jp)通過(guò)基于大量實(shí)驗(yàn)和計(jì)算方法中獲取的蛋白質(zhì)結(jié)構(gòu)數(shù)據(jù)��,分析不同的PDB條目來(lái)識(shí)別蛋白質(zhì)鏈上的結(jié)合位點(diǎn)��。它整合各種蛋白質(zhì)相互作用位點(diǎn)的信息���,包括氨基酸殘基相互作用���、結(jié)合能力以及結(jié)構(gòu)特征等,來(lái)構(gòu)建一個(gè)全面的數(shù)據(jù)庫(kù)��。
圖3.2PiSite數(shù)據(jù)庫(kù)使用
四�����、蛋白藥物靶點(diǎn)數(shù)據(jù)庫(kù)
IUOHAR-DB(https://www.guidetopharmacology.org/)是G蛋白偶聯(lián)受體�����、離子通道數(shù)據(jù)庫(kù)�,提供這些蛋白的基因�、功能、結(jié)構(gòu)��、配體����、表達(dá)圖譜、信號(hào)轉(zhuǎn)導(dǎo)機(jī)制�����、多樣性等數(shù)據(jù)����。可以用于藥物靶點(diǎn)查找���,可以按照免疫過(guò)程信號(hào)通路查詢或者在不同細(xì)胞特異表達(dá)查詢或者根據(jù)蛋白激酶、離子通道分類進(jìn)行查詢�。
圖4.1IUOHAR-DB數(shù)據(jù)庫(kù)使用
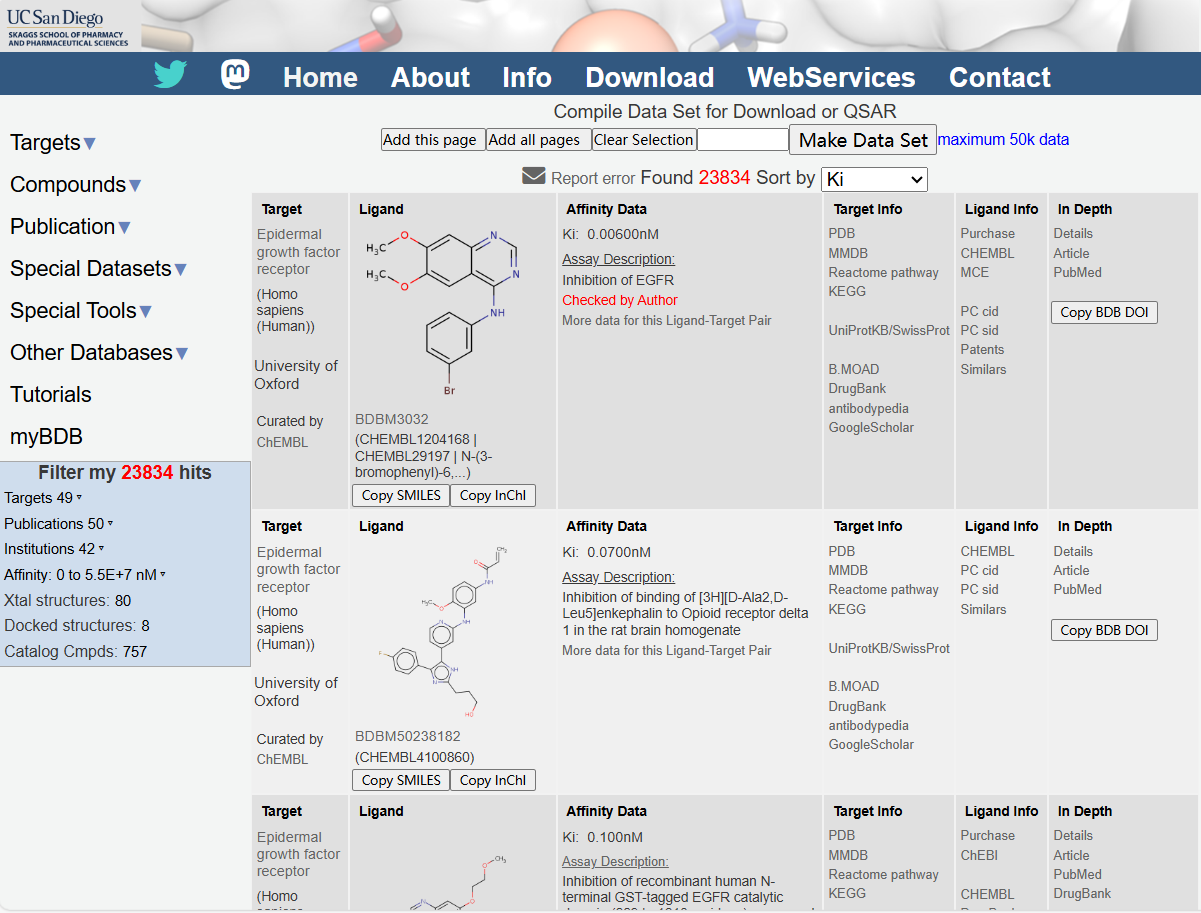
Binding-DB(Bindind Database)��,加州大學(xué)圣地亞哥分校 Michael K.Gilson實(shí)驗(yàn)室發(fā)布的一個(gè)可公開(kāi)訪問(wèn)的主要收集藥物靶點(diǎn)蛋白質(zhì)和類藥小分子之間相互作用親和力的數(shù)據(jù)庫(kù)。BindingDB的數(shù)據(jù)來(lái)自相關(guān)文獻(xiàn)報(bào)道數(shù)據(jù)����、專利信息、PubChem BioAssays 數(shù)據(jù)和 ChEMBL 記錄數(shù)據(jù)。BindingDB 收錄了110萬(wàn)個(gè)化合物與8800個(gè)靶點(diǎn)之間的250萬(wàn)個(gè)相互作用數(shù)據(jù)。
圖4.2Binding-DB數(shù)據(jù)庫(kù)使用
五���、蛋白結(jié)構(gòu)域數(shù)據(jù)庫(kù)
每一種蛋白質(zhì)都有其獨(dú)特的功能和結(jié)構(gòu),這構(gòu)成了生物多樣性的一部分。為了揭示這種多樣性需要對(duì)成千上萬(wàn)的蛋白質(zhì)進(jìn)行分類和功能預(yù)測(cè)��。
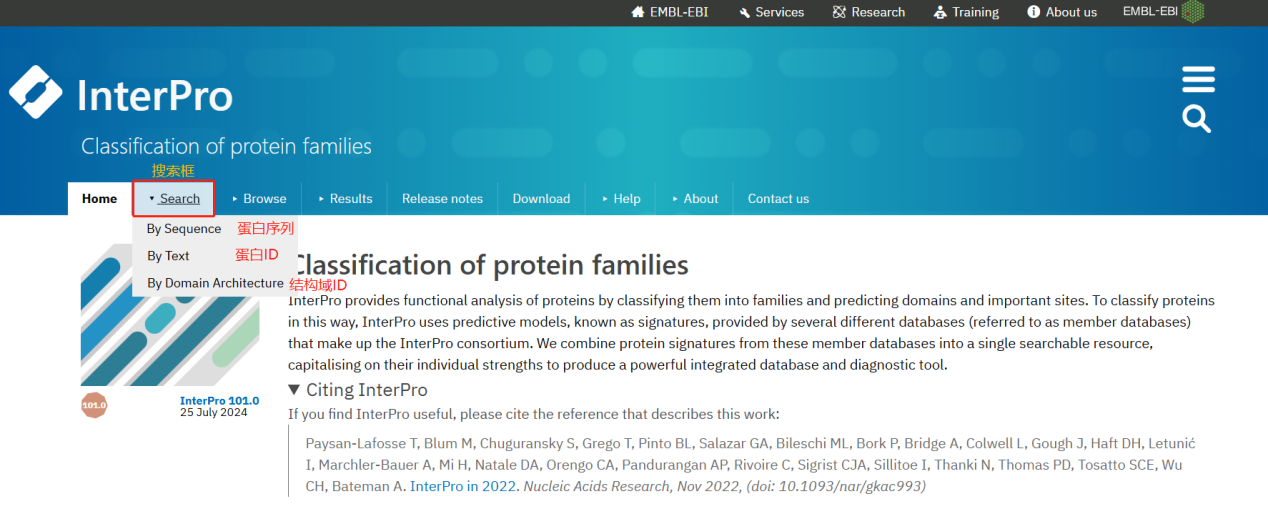
InterPro(https://www.ebi.ac.uk/interpro/)將13個(gè)蛋白質(zhì)特征數(shù)據(jù)庫(kù)合并為一個(gè)集中資源����,包括Coils、Gene3D、Pfam��、PRINTS�����、ProSitePatterns�����、ProSiteProfiles、SMART、SUPERFAMILY����、TIGRFAM�、ProDom���、PIR等數(shù)據(jù)庫(kù)�����?�?梢灾苯铀阉鞯鞍仔蛄谢蛘呓Y(jié)構(gòu)域ID獲得結(jié)構(gòu)域結(jié)果。
圖5.1InterPro數(shù)據(jù)庫(kù)使用
六、蛋白三維結(jié)構(gòu)數(shù)據(jù)庫(kù)
蛋白質(zhì)的生物活性不僅決定于蛋白質(zhì)分子的一級(jí)結(jié)構(gòu)����,而且與其特定的空間結(jié)構(gòu)密切相關(guān)。異常的蛋白質(zhì)空間結(jié)構(gòu)很可能導(dǎo)致其生物活性的降低��、喪失���。在功能和結(jié)構(gòu)細(xì)節(jié)上闡明關(guān)于蛋白質(zhì)折疊的過(guò)程將對(duì)相關(guān)疾病的預(yù)防和治療有重要意義�����。
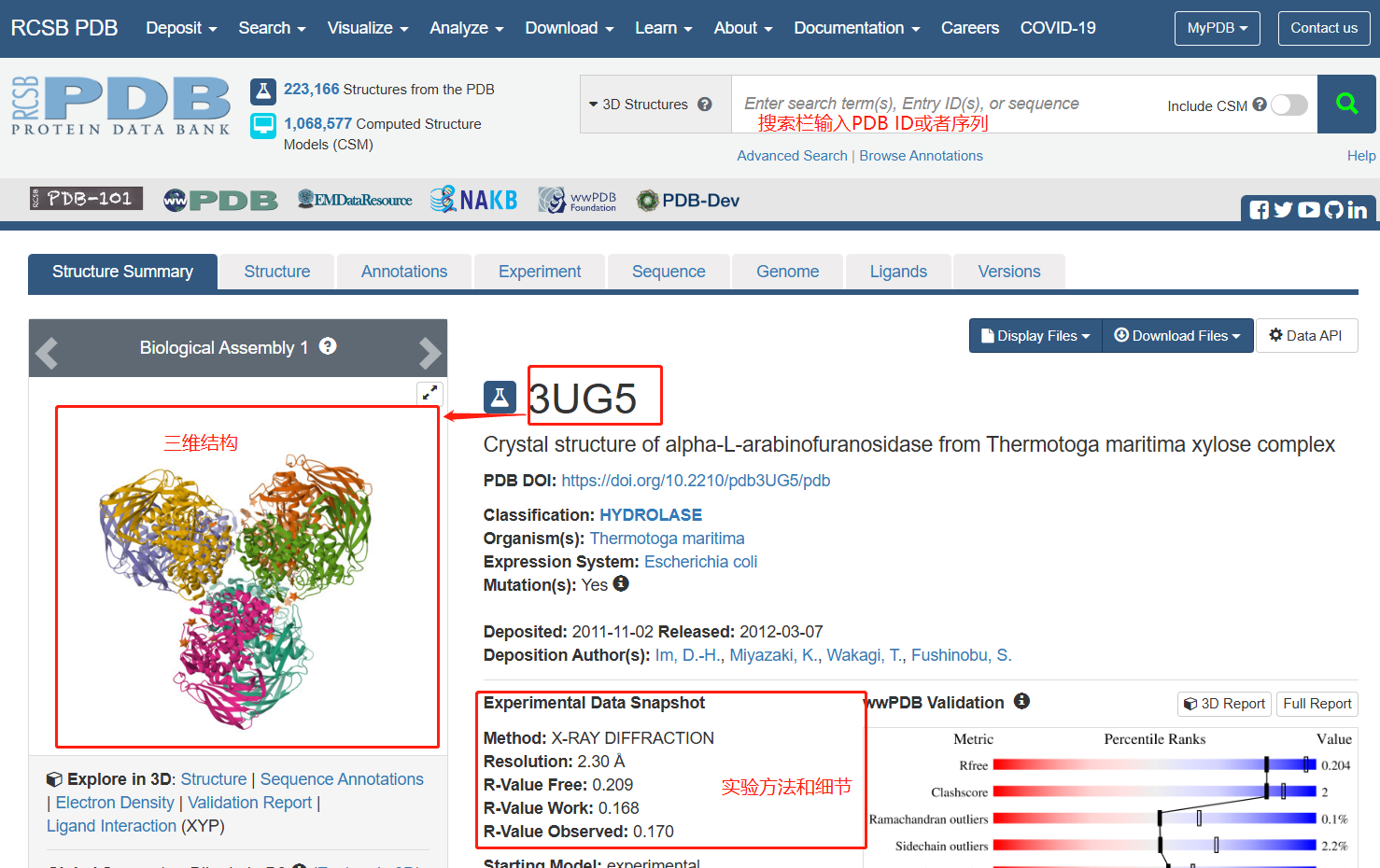
PDB(Protein Data Bank���,http://www.rcsb.org/)�����,是美國(guó)Brookhaven國(guó)家實(shí)驗(yàn)室于1971年創(chuàng)建的�,通過(guò)X射線單晶衍射�、核磁共振、電子衍射等實(shí)驗(yàn)手段確定的蛋白質(zhì)���、多糖、核酸、病毒等生物大分子的三維結(jié)構(gòu)數(shù)據(jù)庫(kù)����,通過(guò)搜索蛋白質(zhì)可以獲得蛋白質(zhì)結(jié)構(gòu)的三維可視化(如果有配體相互作用)和結(jié)構(gòu)質(zhì)量指標(biāo)��。
圖6.1PDB數(shù)據(jù)庫(kù)使用
AlphaFold(https://deepmind.google/technologies/alphafold/)是由谷歌DeepMind開(kāi)發(fā)的一款蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)程序,它采用 AI 和深度學(xué)習(xí)技術(shù)僅根據(jù)其基因序列就能預(yù)測(cè)蛋白質(zhì)的3D結(jié)構(gòu),僅需數(shù)日內(nèi)可識(shí)別蛋白質(zhì)的形狀����,從而找到藥物靶點(diǎn)��。
圖6.2AlphaFold預(yù)測(cè)蛋白
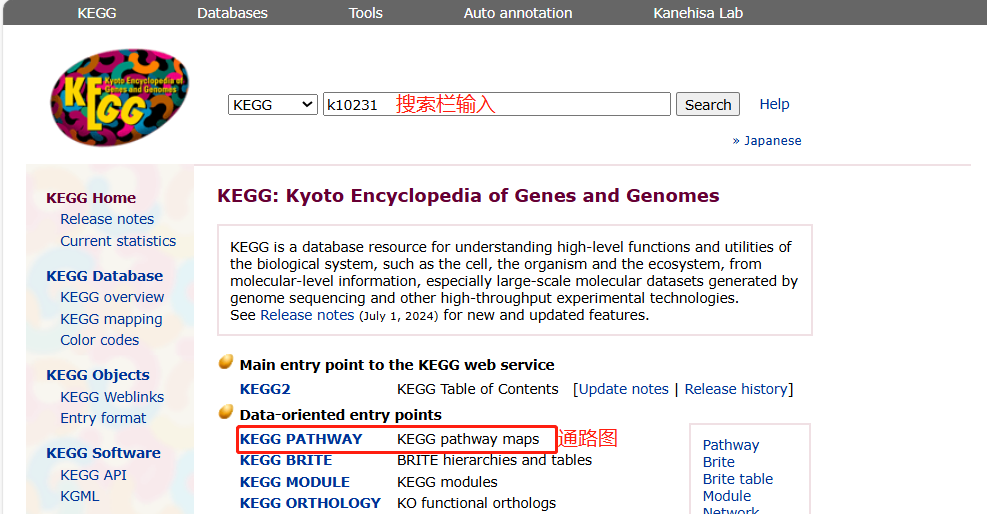
KEGG(https://www.genome.jp/kegg/)是一個(gè)整合了基因組、化學(xué)和系統(tǒng)功能信息的綜合性數(shù)據(jù)庫(kù)��,連接已知分子間相互作用的信息網(wǎng)絡(luò)���,如代謝通路�、復(fù)合物���、生化反應(yīng)����。KEGG途徑主要包括:代謝、遺傳信息處理����、環(huán)境信息處理�����、細(xì)胞過(guò)程、人類疾病����、藥物開(kāi)發(fā)等����。KEGG包含多個(gè)子數(shù)據(jù)庫(kù)����,有代謝通路、基因信息���、化合物、酶���、藥物等等,均包含大量有用的信息�,經(jīng)常使用的是Pathway查詢與分析 �。
圖7.1KEGG數(shù)據(jù)庫(kù)使用
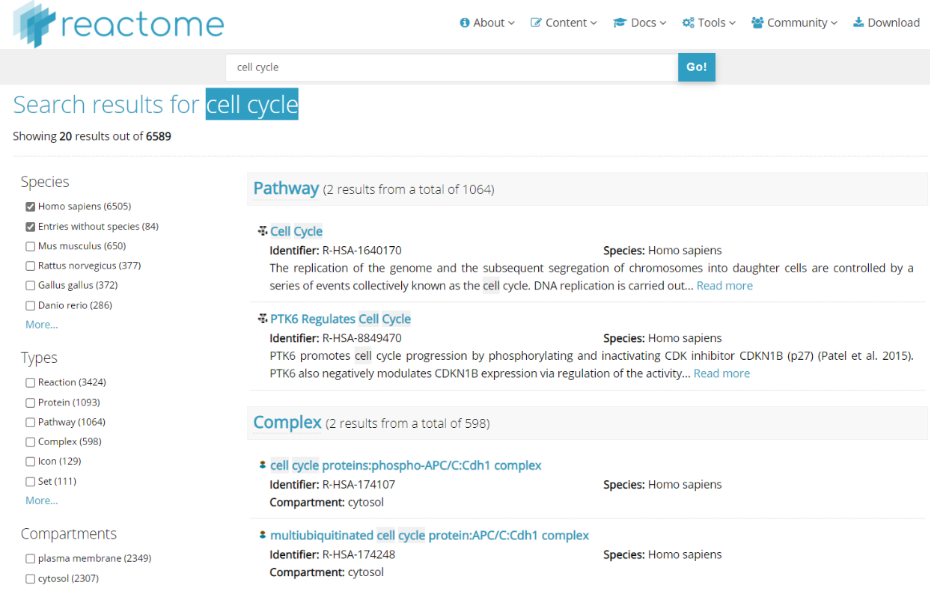
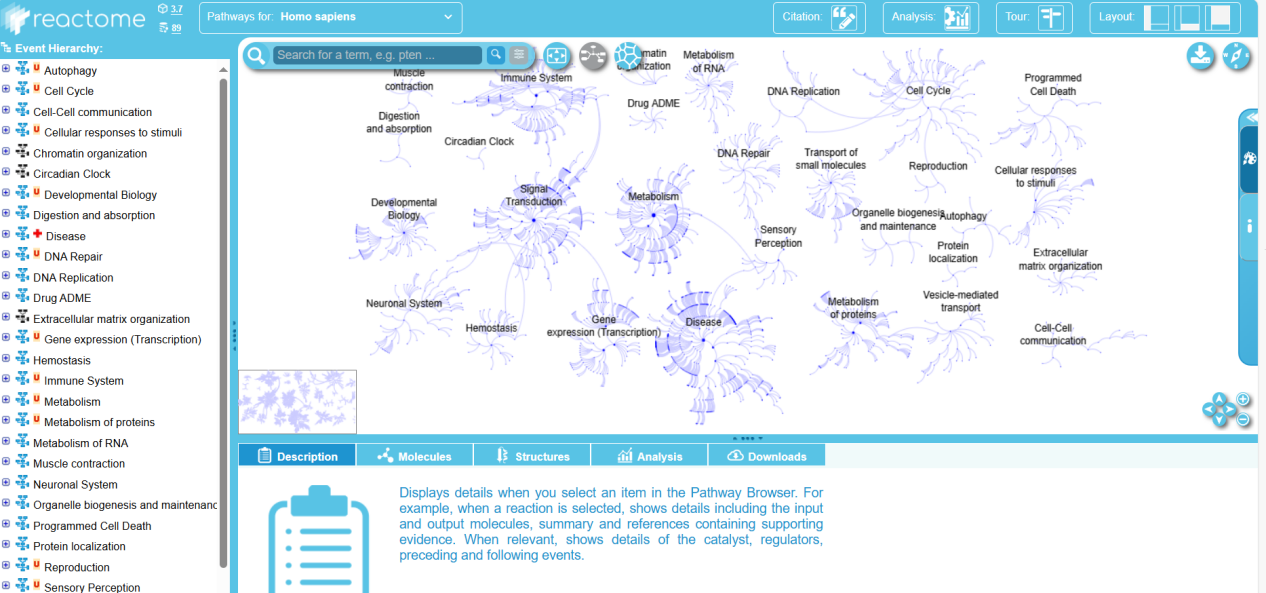
Reactome(https://reactome.org/)是一個(gè)免費(fèi)�����、開(kāi)源、數(shù)據(jù)經(jīng)過(guò)手動(dòng)篩選和同行評(píng)審的生物分子通路知識(shí)數(shù)據(jù)庫(kù)。目前該庫(kù)覆蓋了19個(gè)物種的通路研究����,包括經(jīng)典的代謝通路��、信號(hào)轉(zhuǎn)導(dǎo)、基因轉(zhuǎn)錄調(diào)控、細(xì)胞凋亡與疾病。輸入蛋白或者基因搜索即可得到相關(guān)通路信息�,并可以對(duì)感興趣的通路進(jìn)行富集分析�����。
圖7.2Reactome數(shù)據(jù)庫(kù)使用
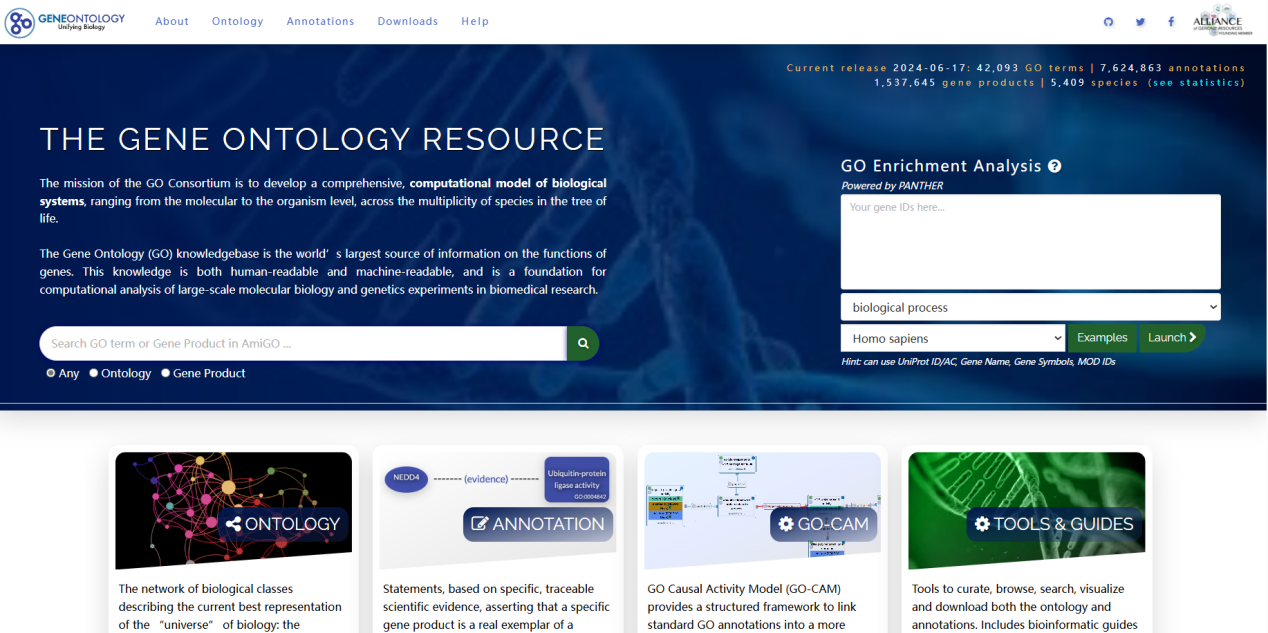
GO(Gene Ontology,https://www.geneontology.org/)數(shù)據(jù)庫(kù)為統(tǒng)一基因功能描述而建立�����,Gene Ontology把描述基因和基因產(chǎn)物功能的術(shù)語(yǔ)歸納到三個(gè)不同維度的本體中(BP-生物過(guò)程�����、CC-細(xì)胞組分����、MF-分子功能)。搜索感興趣的蛋白或者基因ID獲取相關(guān)信息�����。
八����、Human Protein Atlas
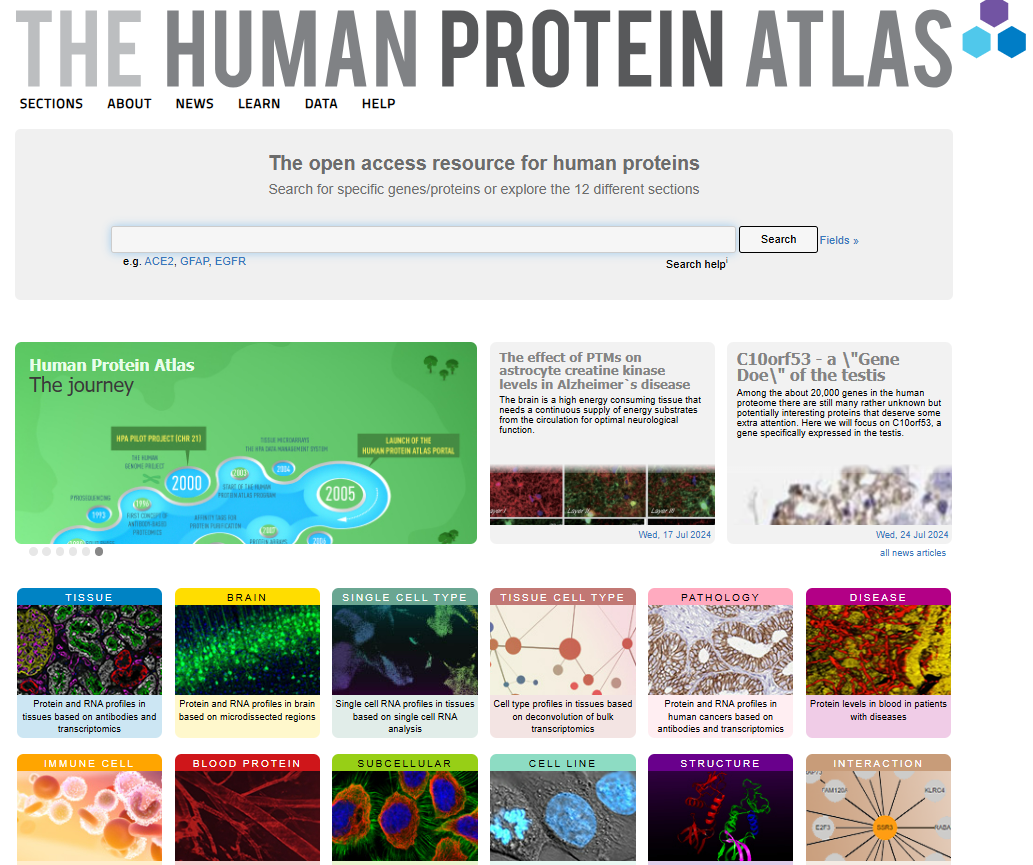
Human Protein Atlas-人類蛋白質(zhì)圖譜(HPA����,https://www.proteinatlas.org)瑞典 Knut & Alice Wallenberg基金會(huì)創(chuàng)建,利用各種技術(shù),包括基于抗體的成像技術(shù)�、基于質(zhì)譜的蛋白質(zhì)組學(xué)����、轉(zhuǎn)錄組和系統(tǒng)生物學(xué)等���,繪制細(xì)胞���、組織和器官中的人類蛋白質(zhì)圖譜����。人類蛋白質(zhì)圖譜由十二個(gè)獨(dú)立的部分組成,包括組織圖譜、腦圖譜、單細(xì)胞圖譜��、組織細(xì)胞類圖譜�、病理圖譜、疾病血液圖譜、免疫細(xì)胞圖譜����、血液蛋白圖譜��、亞細(xì)胞圖譜、細(xì)胞系圖譜、結(jié)構(gòu)圖譜�、相互作用圖譜�。通過(guò)搜索蛋白獲得蛋白質(zhì)在多種人類正常組織�����、腫瘤組織�、細(xì)胞系和血液細(xì)胞內(nèi)的分布和表達(dá)情況�。
九、蛋白組學(xué)數(shù)據(jù)存儲(chǔ)網(wǎng)站
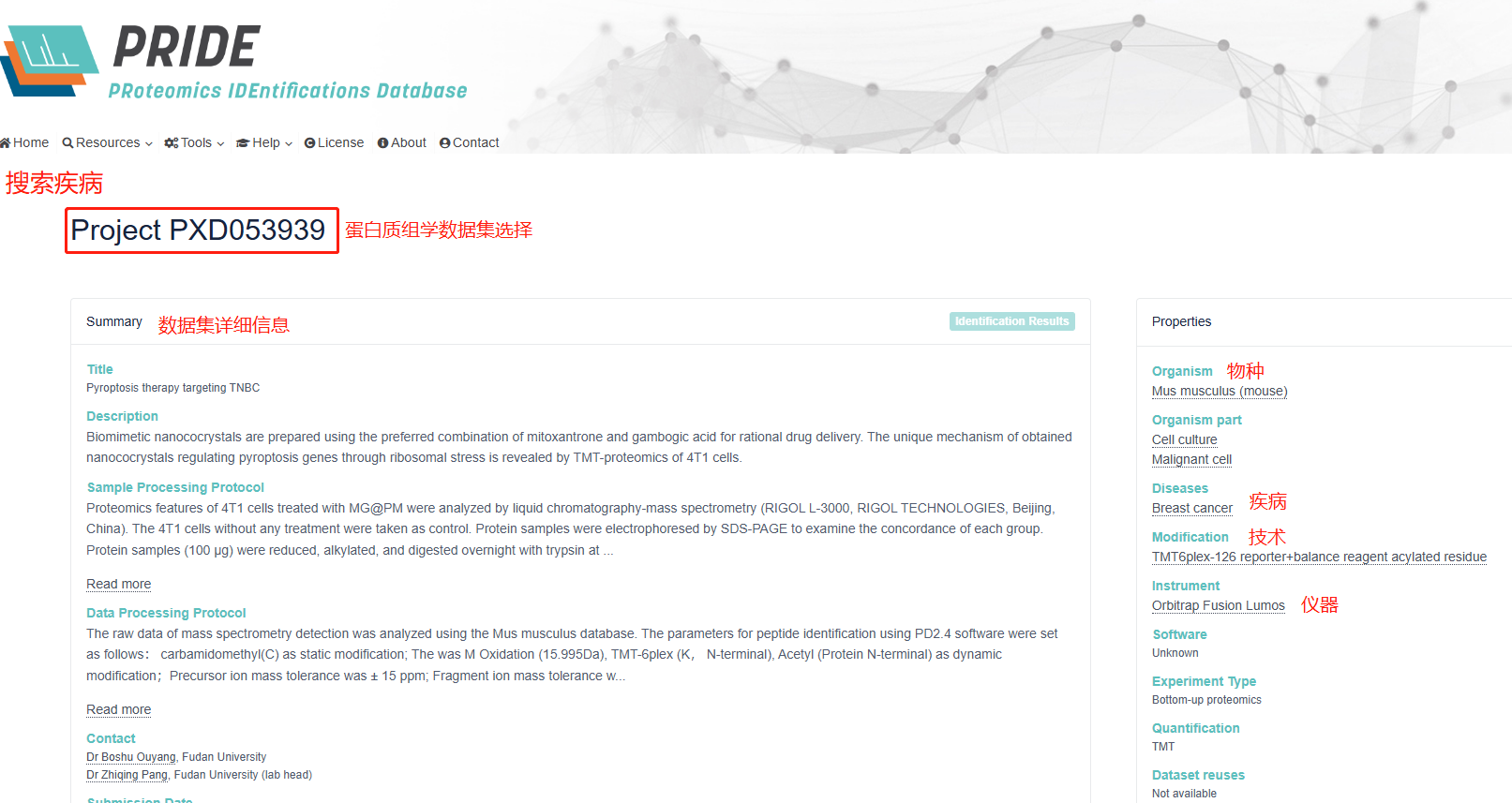
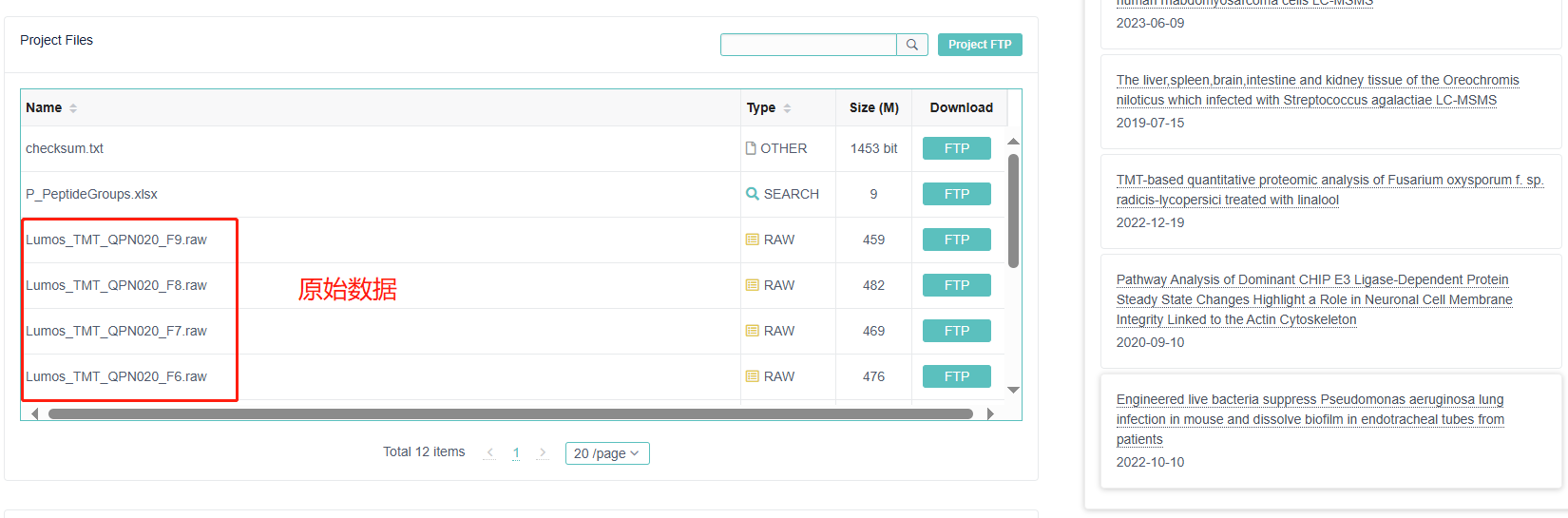
ProteomeXchange(https://www.ebi.ac.uk/pride/)是專門用來(lái)儲(chǔ)存蛋白檢測(cè)數(shù)據(jù)的網(wǎng)站。例如通過(guò)檢索特定癌癥,可以在 ProteomeXchange 得到某類癌癥有關(guān)的蛋白質(zhì)組學(xué)數(shù)據(jù)集���,并可以詳細(xì)看到每一個(gè)數(shù)據(jù)集的基本信息�,包括研究的物種、疾病��、使用的蛋白質(zhì)組學(xué)方法以及儀器等信息�,并提供該數(shù)據(jù)集的原始文件以供研究者下載。
圖9.1ProteomeXchange網(wǎng)站時(shí)使用
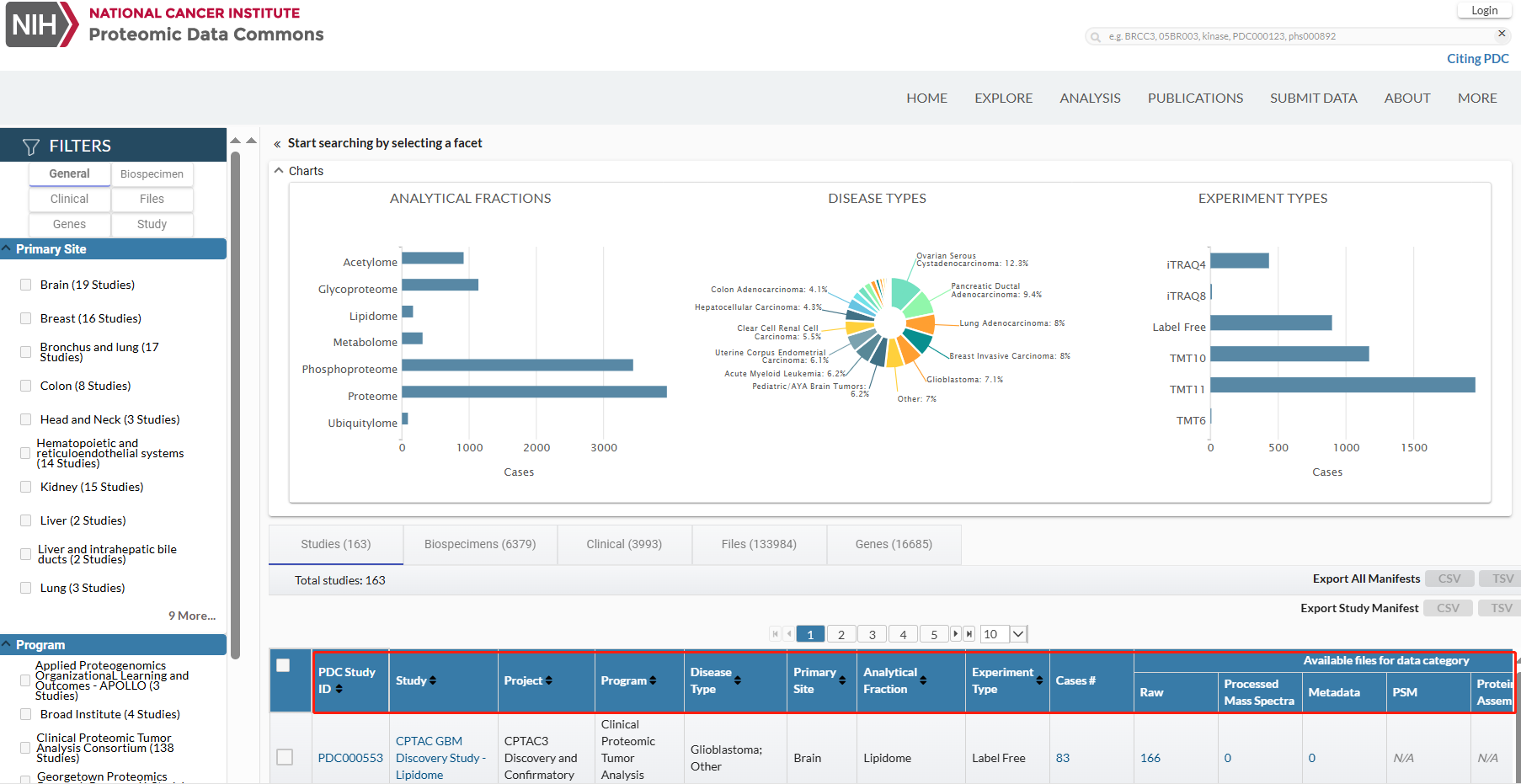
CPTAC(Clinical Proteomic Tumor Analysis Consortium����,https://pdc.cancer.gov/pdc/browse)是由美國(guó)國(guó)家癌癥研究所(NCI)資助建立的一個(gè)綜合性數(shù)據(jù)庫(kù)�,CPTAC數(shù)據(jù)庫(kù)主要提供了臨床隊(duì)列不同癌癥類型的蛋白質(zhì)組學(xué)數(shù)據(jù)�����,此外還包含基因組測(cè)序��、miRNA測(cè)序和DNA甲基化數(shù)據(jù),希望通過(guò)應(yīng)用大規(guī)模蛋白質(zhì)組學(xué)和基因組分析(proteogenomics)來(lái)加速對(duì)癌癥分子基礎(chǔ)的理解�。在首頁(yè)搜索欄輸入蛋白�����、基因或者疾病可以獲得數(shù)據(jù)集。